瘤胃微生物是反刍动物消化系统不可分割的一部分,它们不仅满足反刍动物的大部分营养需求,而且负责高达90%的代谢需求[1-2]。因此,深入了解瘤胃微生态以及复杂的微生物群落变化对提高反刍动物生产效益十分重要。随着组学技术的发展,瘤胃微生物的功能潜力及其与动物性能的关系也得到了研究[3-8]。瘤胃中不同的微生物群落成员能够进行类似的代谢途径,并在不同的代谢途径中相互替代,这是瘤胃微生物可塑性的一个关键特征;然而,这也是在解开单一成员的不同功能特性方面的一个重大挑战,因为存在大量的混杂效应。这就需要新的生物技术和计算方法来捕捉和整合不同的相互关系、动态和路径。宏转录组学可提供微生物的组成和信息表达[9]。研究表明,宏转录组学是揭示奶牛瘤胃微生物功能与饲料效率之间关系的更好方法,可以通过准确阐明注释的基因哪些被转录以及在何种程度上转录,从而能够证明细菌环境中的潜在功能,发现更多的纤维降解微生物种类和家族基因[10]。
饲料短缺导致低收入和中等收入国家的畜牧生产出现巨大的差距,虽然我国秸秆资源丰富,但是与之不相符的是秸秆资源利用率偏低。研究表明,合适的稻草添加量、饲料配比以及稻草的加工工艺都可以有效增加草食动物的经济效益[11-14]。Xie等[15]研究发现,低品质粗饲料干预对瘤胃微生物结构有显著影响,在用高品质粗饲料饲喂反刍动物的同时使用低品质粗饲料进行反复干预可以提高宿主的纤维溶解消化率。He等[16]通过宏转录组学技术发现湖羊瘤胃微生物编码了一系列能够降解纤维素的新型酶。而利用宏转录组学研究低品质粗饲料对瘤胃微生物生态系统影响的甚少。若能了解低品质高纤维粗饲料在瘤胃中的降解特性,并揭示其调控瘤胃微生物生态系统的分子机理,对饲料资源的利用以及促进畜牧业的发展有着重要的作用。本试验基于宏转录组学技术研究稻草替代部分比例全株玉米青贮对奶牛瘤胃微生物组成、差异表达基因、信号通路和碳水化合物活性酶(CAZy)的影响,可帮助人们更好地理解不同饲料来源的瘤胃微生物生态变化和调节机制以及与宿主之间的内在联系,丰富关于纤维消化与反刍动物瘤胃微生物群落之间的关系信息,挖掘多种木质纤维素降解酶,从而提高瘤胃微生物群落作为一种独特资源的利用率,并为实际生产中稻草资源的利用提供理论支撑。
1 材料与方法 1.1 试验动物与设计饲养试验在宁夏农垦贺兰山奶业茂盛牧场进行。选取8头健康、体重接近的泌乳后期中国荷斯坦奶牛,随机分为2组,每组4个重复,每个重复1头牛。CS组奶牛饲粮中粗饲料由苜蓿、苜蓿青贮与全株玉米青贮组成;RS组奶牛饲粮是由稻草替代CS组饲粮的粗饲料中1/3的全株玉米青贮,其他成分不变。试验期共21 d,其中预试期14 d,正试期7 d。
1.2 试验饲粮与饲养管理试验饲粮参照NRC(2001)营养标准配制,为全混合日粮(TMR)形式,精粗比为30 ∶ 70(干物质基础),对照组和试验组分开围栏饲养,自由饮水,每天饲喂2次。试验饲粮组成及营养水平见表 1。
|
|
表 1 试验饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of experimental diets (DM basis) |
奶牛瘤胃液的采集方法:使用牛鼻夹固定牛头,将不锈钢弹簧蛇管(内部乳胶管)从口腔送入瘤胃内,牛咀嚼过程中瘤胃液顺导管自然流出,对流出的瘤胃液进行收集即可。试验第19天开始采集瘤胃液,每天采集4次,分别为饲喂前和饲喂后2、6和12 h,连续采样3 d。将瘤胃液进行混合分装至5 mL冻存管中,快速转移至液氮中冷冻保存,随后立即转移至-80 ℃超低温冰箱中保存,以备后续检测。
1.4 测序及生物信息学分析提取样品总RNA、构建文库及测序由北京诺禾致源科技股份有限公司完成,测序平台为Illumina NovaSeq 6000测序平台。测序得到的原始数据经过质控到有效数据,然后进行物种注释分析、差异表达基因分析、功能数据库注释分析。
1.5 数据处理与分析数据均先采用Excel 2019进行初步整理,然后采用SAS 9.4进行统计分析,采用t检验进行差异显著性分析,多重比较采用Duncan氏法进行,以P < 0.05作为差异显著性的判断标准。
以|log2(差异倍数)|>0且P < 0.05为标准,使用DEGSeq 1.12和Deseq 1.10.1软件对CS组和RS组进行差异表达基因筛选和分析,采用GOSeq 1.10,topGO 2.10软件对差异表达基因集进行GO功能富集分析,利用KOBAS v2.0.12软件筛选KEGG数据库中差异表达基因富集到的信号通路,使用CAZy数据库的对应工具HMMSCAN将基因集与CAZy数据库进行比对。以P < 0.05作为差异显著性的阈值。
2 结果与分析 2.1 宏转录组文本概述经过对原始reads过滤之后,本试验共获得8个生物学独立的宏转录组测序文本(表 2),共获得377 690 128条有效reads。数据整体测序错误率在0.02%~0.03%,Q20>96%,Q30>91%,说明建库和测序质量好,满足后续分析要求。
|
|
表 2 测序结果汇总 Table 2 Summary of sequencing output |
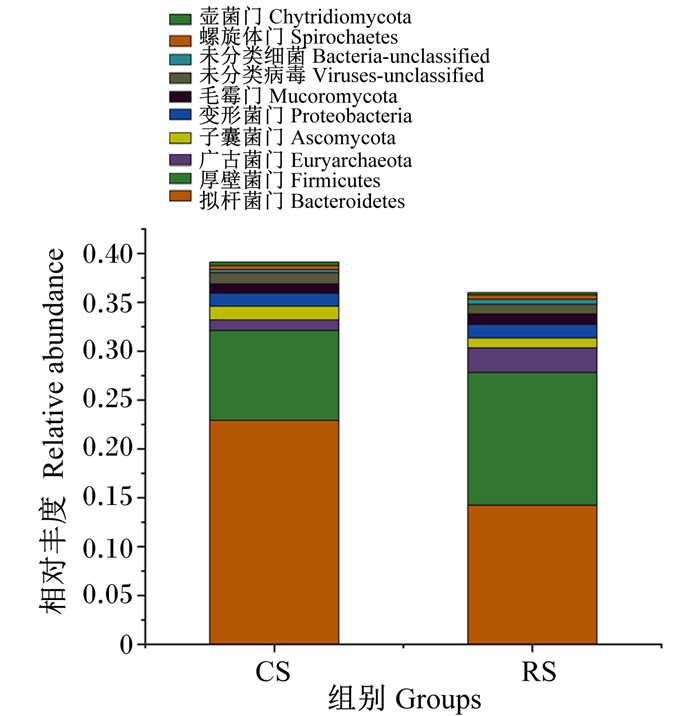
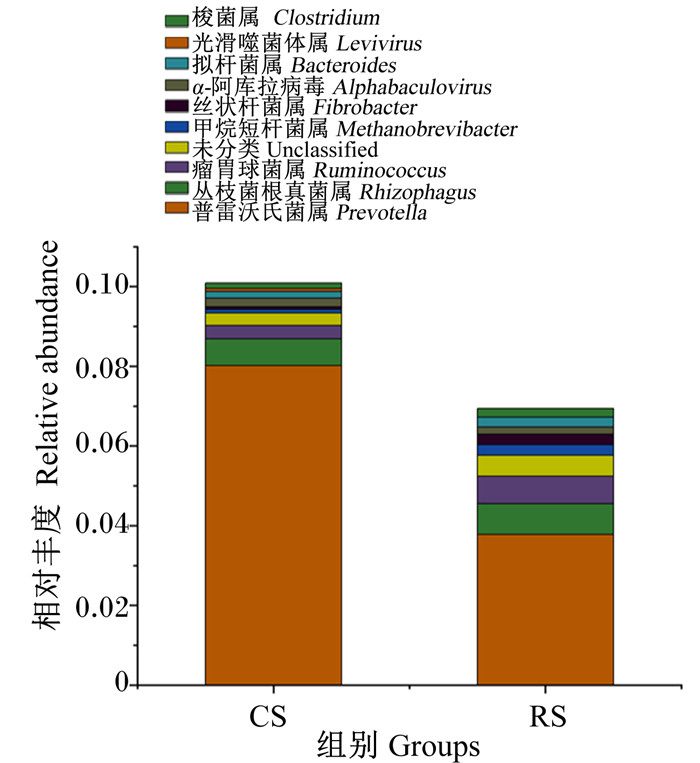
由图 1和图 2可知,在门水平上,CS组和RS组瘤胃微生物组成基本相似,CS组和RS组的优势菌门均为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)。在属水平上,饲粮类型对奶牛瘤胃微生物组成有较大影响,CS组和RS组的优势菌属均为普雷沃氏菌属(Prevotella)、丛枝菌根真菌属(Rhizophagus)和瘤胃球菌属(Ruminococcus)。由表 3可知,CS组广古菌门(Euryarchaeota)、瘤胃球菌属、甲烷短杆菌属(Methanobrevibacter)、丝状杆菌属(Fibrobacter)的相对丰度显著低于RS组(P < 0.05)。
|
图 1 门水平上瘤胃微生物组成 Fig. 1 Rumen microbial composition at phylum level |
|
图 2 属水平上瘤胃微生物组成 Fig. 2 Rumen microbial composition at genus level |
|
|
表 3 稻草替代部分全株玉米青贮对奶牛瘤胃菌群相对丰度的影响 Table 3 Effects of partial replacement of whole corn silage by rice straw on relative abundance of rumen microflora in dairy cows |
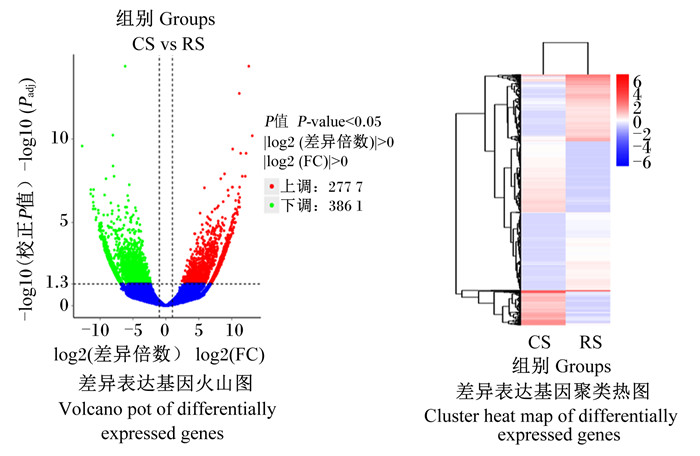
对CS组和RS组的转录数据进行比对分析,得到差异表达基因6 638个,其中上调表达的有2 777个,下调表达的有3 861个(图 3),前20个差异表达基因如表 4所示。对差异表达基因进行聚类分析,绘制聚类热图(图 3),从图中可以看出组内基因的表达量基本趋于一致,说明生物学重复性较好且这些基因可能具有共同的代谢途径及信号通路。
|
图 3 差异表达基因分析 Fig. 3 Analysis of differentially expressed genes |
|
|
表 4 前20个差异表达基因 Table 4 Top 20 differentially expressed genes |
通过GO数据库对差异表达基因进行功能富集分类。由表 5可知,差异表达基因主要富集于生物合成过程(biosynthetic process)、有机物质的生物合成过程(organic substance biosynthetic process)、细胞生物合成过程(cellular biosynthetic process)和细胞代谢过程(cellular metabolic process)等。
|
|
表 5 部分差异表达基因的GO注释 Table 5 GO functional classification of partial differentially expressed genes |
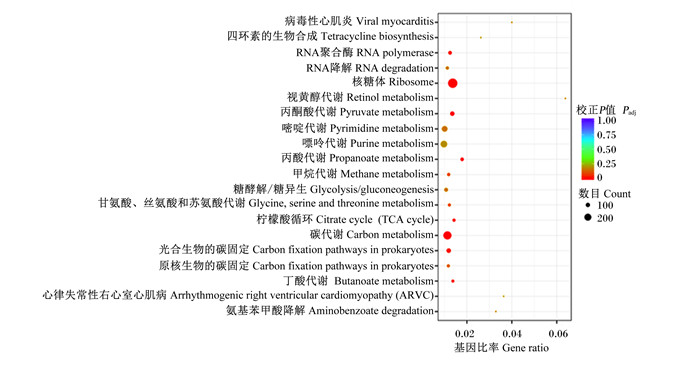
对差异表达基因进行KEEG代谢通路进行分析,结果显示,共富集到236条信号通路,选取了富集最显著的前20条信号通路,以散点图形式展示(图 4)。显著富集于前10位的代谢通路如表 6所示,涉及到1 149个差异表达基因。其中,KEEG通路富集差异表达基因较多的通路有丙酸代谢(propanoate metabolism)、碳代谢(carbon metabolism)、丙酮酸代谢(pyruvate metabolism)、柠檬酸循环(citrate cycle)、丁酸代谢(butanoate metabolism)、甲烷代谢(methane metabolism)。
|
图 4 差异表达基因KEEG通路富集分析散点图(前20个) Fig. 4 KEEG pathways enrichment analysis scatter diagram of differentially expressed genes (top 20) |
|
|
表 6 差异表达基因显著富集的KEEG通路列表 Table 6 List of KEEG pathway for differentially expressed genes |
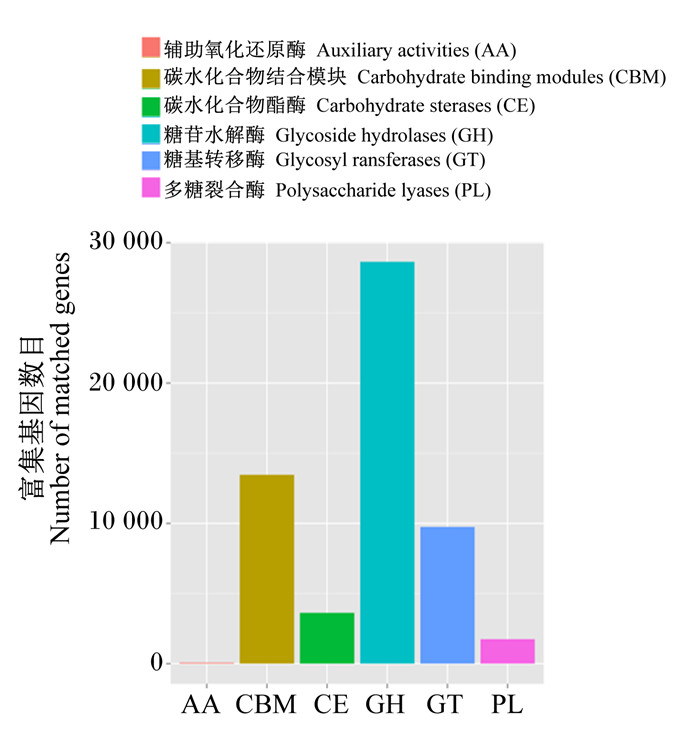
由图 5可以直观看出被富集到的基因数量,其中糖苷水解酶(glycoside hydrolases, GH)是丰度最高的类别,差异表达基因有28 631个,其次是碳水化合物结合模块(carbohydrate-binding modules, CBM)、糖基转移酶(glycosyl ransferases, GT)、碳水化合物酯酶(carbohydrate sterases, CE)、多糖裂合酶(polysaccharide lyases, PL)、辅助氧化还原酶(auxiliary activities, AA),差异表达基因分别有13 457、9 743、3 613、1 738和66个。此外,富集到纤维素、半纤维素和寡糖降解酶的差异表达基因的数量如表 7所示。由表 7可知,CS组纤维素降解酶GH48和半纤维素降解酶GH8、GH11、GH16富集的差异表达基因的丰度显著低于RS组(P < 0.05),寡糖降解酶GH43、GH3、GH2、GH31富集的差异表达基因的丰度均高于RS组(P>0.05)。
|
图 5 碳水化合物活性酶基因注释结果 Fig. 5 Gene annotation result of CAZy |
|
|
表 7 奶牛瘤胃微生物差异糖苷水解酶功能基因丰度 Table 7 Abundance of functional genes for differential GH in rumen microorganisms of dairy cows |
反刍动物的饲料利用效率主要取决于瘤胃微生物群将潜在可消化饲料转化为可代谢营养物质的能力[18]。研究表明,厚壁菌门、拟杆菌门、变形菌门是反刍动物瘤胃微生物群中的优势菌门[19-20]。厚壁菌门和拟杆菌门在DNA和蛋白质水平上都是丰度最高的成员,包括纤维素降解菌,如产琥珀酸丝状杆菌、黄色瘤胃球菌、白色瘤胃球菌,以及几种高效的半纤维素降解菌,如普雷沃氏菌、丁酸弧菌和假丁酸弧菌[21-25]。大量研究发现,这些细菌都属于核心菌群,几乎在所有反刍动物中存在。在本试验中,CS组和RS组的瘤胃微生物中优势菌门均为厚壁菌门、拟杆菌门和变形菌门,与以上研究结果一致。在本研究中,RS组奶牛瘤胃内具有降解纤维能力的瘤胃球菌及丝状杆菌等菌属的相对丰度均增加,表明在饲喂不同品质粗饲料奶牛瘤胃内,具有纤维降解能力的细菌和真菌菌群的相对丰度存在差异,且在饲喂稻草的奶牛瘤胃内,具有纤维降解能力的细菌菌属的相对丰度较高。研究表明,高纤维低品质饲粮促进瘤胃中乙酸的生成,从而产生更多的氢,促进了甲烷的生成,而利用玉米青贮等高品质饲粮则会降低甲烷排放[26-27]。在本试验中,甲烷短杆菌属在RS组中的相对丰度显著高于CS组,表明甲烷排放与反刍动物饲粮质量之间存在相关性,饲喂高纤维低质量饲粮之后产生的甲烷更多。
3.2 稻草替代部分全株玉米青贮对奶牛瘤胃微生物差异表达基因和功能代谢的影响通过GO功能注释分析发现,差异表达基因主要富集于生物合成过程、有机物质的生物合成过程、细胞生物合成过程和细胞代谢过程等GO功能。其中参与细胞代谢和细胞合成的差异表达基因较多,如topB、parC、parE等基因。topB与单链DNA紧密结合,研究表明,topB需要抑制R环的形成或积累,并且它们对于防止过度的负超螺旋及其对细胞生长和存活的有害影响至关重要[28]。parC和parE负责编码拓扑异构酶Ⅳ,拓扑异构酶Ⅳ的生理学作用是通过其解环链体和解DNA结节,从而协助子染色体的分离。但parC和parE在体内具有哪些功能,以及缺乏这些重要酶的细胞如何存活尚不清楚。孙建政[29]研究发现,在苜蓿青贮中添加松针精油抑制了DNA拓扑异构酶Ⅰ和拓扑异构酶Ⅱ的活性,从而抑制了蛋白质的表达及DNA的合成。石超峰等[30]研究发现,α-松油醇可以抑制大肠杆菌的DNA复制,可能是因为受到DNA拓扑异构酶活性的影响。Dai等[31]研究发现,低品质粗饲料可以通过降低能量供应、降低核糖体活性和增强蛋白质降解能力阻碍乳腺细胞的生长发育。在本试验中,parC、parE基因显著富集,并且在KEGG代谢通路分析中,差异表达基因显著富集到核糖体,而核糖体是蛋白质合成的主要场所。这表明不同品质粗饲料可能影响瘤胃中微生物DNA的合成和蛋白质的表达,从而影响细胞合成和细胞代谢,但具体的作用机制还需要进一步研究。通过KEGG代谢通路分析,差异表达基因主要富集于柠檬酸代谢、丙酮酸代谢、甲烷代谢和挥发性脂肪酸代谢。其中差异表达基因中IDH1基因参与柠檬酸循环途径。异柠檬酸脱氢酶1(IDH1)通过持续产生α-酮戊二酸促进瘤胃上皮细胞中的柠檬酸循环通量,α-酮戊二酸在进入线粒体后被代谢[32]。ATP由酮体和柠檬酸循环产生,为瘤胃上皮细胞的增殖和分化等生理活动提供能量。IDH1异构体催化异柠檬酸转化为α-酮戊二酸,其可被谷氨酸脱氢酶胺化形成谷氨酸[33-34]。谷氨酸是体内氨的主要转运载体,将氨转运至肝脏进行解毒[35-36]。瘤胃上皮增加对氨的吸收,以便与谷氨酸和丙酮酸结合形成无毒的谷氨酰胺,然后转移到肝脏,从而进入尿素循环。同时,瘤胃上皮对甘氨酸、缬氨酸、丝氨酸和蛋氨酸的吸收在瘤胃上皮黏膜侧的总氨基酸浓度较低时较高[37]。IDH1基因影响柠檬酸循环,柠檬酸循环可以响应机体的能量状态,积极地参与机体的代谢协调,这可能是影响KEGG代谢通路中的柠檬酸代谢、丙酮酸代谢与氨基酸代谢途径的主要原因。研究发现nrdD基因主要参与厌氧代谢,在大肠杆菌中,有氧和厌氧生长过程中2种独立的酶催化核糖核苷酸的还原,好氧酶由nrdAB基因编码,厌氧酶由nrdDG基因编码[38-39]。厌氧真菌和厌氧细菌广泛存在于反刍动物瘤胃中,参与饲料中纤维物质的消化过程。在本试验中,CS组nrdD基因表达下调,表明厌氧代谢在RS组中发挥了很大作用,可能是增加了厌氧微生物通过高活性纤维降解酶来分解纤维较高的稻草获得碳水化合物的过程。随后大部分碳水化合物通过多种下游途径发酵,导致有机酸、短链脂肪酸以及各种代谢物和气体(如CO2和H2)的产生[40-42]。其中一些排泄的代谢物,如乳酸、琥珀酸、H2和CO2,进一步转化为丙酸、丁酸、乙酸和甲烷等[43-44]。丙酸、丁酸和乙酸等挥发性脂肪酸是厌氧消化过程的中间产物,而挥发性脂肪酸代谢中的许多产物又是能量产生的底物,包括还原型黄素腺嘌呤二核苷酸(FADH2)、还原型烟酰胺腺嘌呤二核苷酸(NADH)和乙酰辅酶A,它们是柠檬酸循环的辅酶或底物。研究发现,柠檬酸和α-酮戊二酸可以通过厌氧真菌发酵产生,而柠檬酸和α-酮戊二酸都是柠檬酸循环中的关键物质。Cheng等[45]研究发现,厌氧真菌和相关产甲烷菌共培养产生柠檬酸,且厌氧真菌代谢被相关产甲烷菌所改变。因此,在本试验中,稻草替代部分全株玉米青贮后奶牛瘤胃厌氧代谢可能和甲烷短杆菌共同作用影响各种代谢途径。
3.3 稻草替代部分全株玉米青贮对奶牛瘤胃微生物CAZy的影响瘤胃微生物主要是利用其CAZy来降解各种饲料,而主要负责水解碳水化合物糖苷键的GH家族是植物多糖中最丰富和最多样的酶家族。在本试验中,编码半纤维素降解酶的主要基因丰度高于编码纤维素降解酶,这可能是由于半纤维素侧链的多样性需要多种水解酶来降解。其他CAZy家族,包括CBM、GT和PL,都参与了纤维素、木聚糖和果胶的水解[46]。在目前的研究中,CBM没有表现出自身的酶活性,但有助于CAZy与多糖结合,从而增强其活性[47]。研究表明,普雷沃氏菌与参与低聚糖和半纤维素降解的GH家族密切相关[48]。瘤胃球菌和纤维杆菌与大多数参与纤维素降解的GH家族相关,如GH5、GH9、GH45和GH48[49]。在本试验中,主要的纤维降解瘤胃微生物,如拟杆菌、瘤胃球菌、丝状杆菌等,对GH家族均有贡献。Deusch等[50]研究表明,在饲喂以玉米青贮为基础的饲粮时,大多数表达的GH属于GH57和GH2。Zhang等[51]研究发现,高精料饲粮显著减少与半纤维素酶GH10、GH11、GH54和纤维素酶GH1、GH44、GH45相关的CAZy的丰度,并增加了1种寡糖降解酶GH32的丰度。本试验结果与以上研究结果一致。在CS组中,参与纤维素GH48和半纤维素GH8、GH11、GH16的GH家族差异表达基因丰度显著低于RS组,参与寡糖降解的GH家族基因丰度均高于RS组。上述结果表明,不同饲粮组成对瘤胃微生物CAZy功能基因组成有明显影响,稻草替代部分全株玉米青贮提高了与纤维素和半纤维素降解相关的GH家族基因的丰度,而降低了与寡糖降解相关的GH家族基因的丰度。
4 结论在本试验条件下,稻草替代部分全株玉米青贮改变了奶牛瘤胃微生物群落组成,具有降解纤维能力的瘤胃球菌属、丝状杆菌属以及与甲烷排放有关的甲烷短杆菌属的相对丰度均得到提高;差异表达基因主要富集在柠檬酸代谢、丙酮酸代谢、甲烷代谢和挥发性脂肪酸代谢等途径;在CAZy变化上,稻草替代部分全株玉米青贮提高了奶牛瘤胃微生物中参与纤维素和半纤维素降解的GH家族基因的丰度,降低了参与寡糖降解的GH家族基因的丰度。
| [1] |
MIZRAHI I, JAMI E. Review: the compositional variation of the rumen microbiome and its effect on host performance and methane emission[J]. Animal, 2018, 12(Suppl.2): s220-s232. |
| [2] |
LIN L M, XIE F, SUN D M, et al. Ruminal microbiome-host crosstalk stimulates the development of the ruminal epithelium in a lamb model[J]. Microbiome, 2019, 7(1): 83. DOI:10.1186/s40168-019-0701-y |
| [3] |
ROEHE R, DEWHURST R J, DUTHIE C A, et al. Bovine host genetic variation influences rumen microbial methane production with best selection criterion for low methane emitting and efficiently feed converting hosts based on metagenomic gene abundance[J]. PLoS Genetics, 2016, 12(2): e1005846. DOI:10.1371/journal.pgen.1005846 |
| [4] |
SHI W B, MOON C D, LEAHY S C, et al. Methane yield phenotypes linked to differential gene expression in the sheep rumen microbiome[J]. Genome Research, 2014, 24(9): 1517-1525. DOI:10.1101/gr.168245.113 |
| [5] |
CARBERRY C A, WATERS S M, KENNY D A, et al. Rumen methanogenic genotypes differ in abundance according to host residual feed intake phenotype and diet type[J]. Applied and Environmental Microbiology, 2014, 80(2): 586-594. DOI:10.1128/AEM.03131-13 |
| [6] |
JEWELL K A, MCCORMICK C A, ODT C L, et al. Ruminal bacterial community composition in dairy cows is dynamic over the course of two lactations and correlates with feed efficiency[J]. Applied and Environmental Microbiology, 2015, 81(14): 4697-4710. DOI:10.1128/AEM.00720-15 |
| [7] |
HERNANDEZ-SANABRIA E, GUAN L L, GOONEWARDENE L A, et al. Correlation of particular bacterial PCR-denaturing gradient gel electrophoresis patterns with bovine ruminal fermentation parameters and feed efficiency traits[J]. Applied and Environmental Microbiology, 2010, 76(19): 6338-6350. DOI:10.1128/AEM.01052-10 |
| [8] |
CARBERRY C A, KENNY D A, HAN S, et al. Effect of phenotypic residual feed intake and dietary forage content on the rumen microbial community of beef cattle[J]. Applied and Environmental Microbiology, 2012, 78(14): 4949-4958. DOI:10.1128/AEM.07759-11 |
| [9] |
MARCHESI J R, RAVEL J. The vocabulary of microbiome research: a proposal[J]. Microbiome, 2015, 3(1): 31. DOI:10.1186/s40168-015-0094-5 |
| [10] |
XUE M Y, XIE Y Y, ZHONG Y F, et al. Integrated meta-omics reveals new ruminal microbial features associated with feed efficiency in dairy cattle[J]. Microbiome, 2022, 10(1): 32. DOI:10.1186/s40168-022-01228-9 |
| [11] |
王庆, 张巧娥, 吴少飞, 等. 宁夏不同地区稻草营养价值的评定[J]. 黑龙江畜牧兽医, 2022(1): 96-102. WANG Q, ZHANG Q E, WU S F, et al. Evaluation of nutritional value of straw in different regions of Ningxia[J]. Heilongjiang Animal Science and Veterinary Medicine, 2022(1): 96-102 (in Chinese). |
| [12] |
张建童, 计接权, 熊小文, 等. 稻草养羊研究进展[J]. 粮油与饲料科技, 2020(3): 35-36. ZHANG J T, JI J Q, XIONG X W, et al. Research progress of sheep farming with rice straw[J]. Grain Oil and Feed Technology, 2020(3): 35-36 (in Chinese). DOI:10.3969/j.issn.1008-6137.2020.03.011 |
| [13] |
KIM J G, HAM J S, LI Y W, et al. Development of a new lactic acid bacterial inoculant for fresh rice straw silage[J]. Asian-Australasian Journal of Animal Sciences, 2017, 30(7): 950-956. DOI:10.5713/ajas.17.0287 |
| [14] |
MARBUN T D, LEE K, SONG J, et al. Effect of lactic acid bacteria on the nutritive value and in vitro ruminal digestibility of maize and rice straw silage[J]. Applied Sciences, 2020, 10(21): 7801. DOI:10.3390/app10217801 |
| [15] |
XIE X, YANG C L, GUAN L L, et al. Persistence of cellulolytic bacteria Fibrobacter and Treponema after short-term corn stover-based dietary intervention reveals the potential to improve rumen fibrolytic function[J]. Frontiers in Microbiology, 2018, 9: 1363. DOI:10.3389/fmicb.2018.01363 |
| [16] |
HE B, JIN S W, CAO J W, et al. Metatranscriptomics of the Hu sheep rumen microbiome reveals novel cellulases[J]. Biotechnology for Biofuels, 2019, 12(1): 153. DOI:10.1186/s13068-019-1498-4 |
| [17] |
冯仰廉, 周建民, 张晓明, 等. 我国奶牛饲料产奶净能值测算方法的研究[J]. 中国畜牧杂志, 1987(1): 8-11. FENG Y L, ZHOU J M, ZHANG X M, et al. Study on the calculation method of feed milk net energy of dairy cows in China[J]. Chinese Journal of Animal Science, 1987(1): 8-11 (in Chinese). |
| [18] |
LØVENDAHL P, DIFFORD G F, LI B, et al. Review: selecting for improved feed efficiency and reduced methane emissions in dairy cattle[J]. Animal, 2018, 12(Suppl.2): s336-s349. |
| [19] |
BI Y L, ZENG S Q, ZHANG R, et al. Effects of dietary energy levels on rumen bacterial community composition in Holstein heifers under the same forage to concentrate ratio condition[J]. BMC Microbiology, 2018, 18(1): 69. DOI:10.1186/s12866-018-1213-9 |
| [20] |
HENDERSON G, COX F, GANESH S, et al. Erratum: rumen microbial community composition varies with diet and host, but a core microbiome is found across a wide geographical range[J]. Scientific Reports, 2016, 6: 19175. DOI:10.1038/srep19175 |
| [21] |
MORAÏS S, MIZRAHI I. Islands in the stream: from individual to communal fiber degradation in the rumen ecosystem[J]. FEMS Microbiology Reviews, 2019, 43(4): 362-379. DOI:10.1093/femsre/fuz007 |
| [22] |
JAMI E, MIZRAHI I. Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [23] |
SNELLING T J, WALLACE R J. The rumen microbial metaproteome as revealed by SDS-PAGE[J]. BMC Microbio, 2017, 17: 9. DOI:10.1186/s12866-016-0917-y |
| [24] |
HART E H, CREEVEY C J, HITCH T, et al. Meta-proteomics of rumen microbiota indicates niche compartmentalisation and functional dominance in a limited number of metabolic pathways between abundant bacteria[J]. Scientific Reports, 2018, 8(1): 10504. DOI:10.1038/s41598-018-28827-7 |
| [25] |
BOURNE D G, WEBSTER N S. Coral reef bacterial communities[M]//ROSENBERG E, DELONG E F, LORY S, et al. The prokaryotes. Berlin: Springer, 2013: 163-187.
|
| [26] |
ZHANG R Y, LIU J H, JIANG L S, et al. Effect of high-concentrate diets on microbial composition, function, and the VFAs formation process in the rumen of dairy cows[J]. Animal Feed Science and Technology, 2020, 269: 114619. DOI:10.1016/j.anifeedsci.2020.114619 |
| [27] |
WANG M, WANG R, XIE T Y, et al. Shifts in rumen fermentation and microbiota are associated with dissolved ruminal hydrogen concentrations in lactating dairy cows fed different types of carbohydrates[J]. The Journal of Nutrition, 2016, 146(9): 1714-1721. DOI:10.3945/jn.116.232462 |
| [28] |
BROCHU J, VLACHOS-BRETON, SUTHERLAND S, et al. Topoisomerases Ⅰ and Ⅲ inhibit R-loop formation to prevent unregulated replication in the chromosomal Ter region of Escherichia coli[J]. PLoS Genetics, 2018, 14(9): e1007668. DOI:10.1371/journal.pgen.1007668 |
| [29] |
孙建政. 松针精油对苜蓿青贮品质及微生物的影响研究[D]. 硕士学位论文. 石河子: 石河子大学, 2021. SUN J Z. Study on the effect of pine needle essential oil on the quality and microorganisms of alfalfa silage[D]. Master's Thesis. Shihezi: Shihezi University, 2021. (in Chinese) |
| [30] |
石超峰, 殷中琼, 魏琴, 等. α-松油醇对大肠杆菌的抑菌作用及其机理研究[J]. 畜牧兽医学报, 2013, 44(5): 796-801. SHI C F, YIN Z Q, WEI Q, et al. Bacteriostatic action and mechanism of α-terpineol on Escherichia coli[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(5): 796-801 (in Chinese). |
| [31] |
DAI W T, CHEN Q, WANG Q J, et al. Complementary transcriptomic and proteomic analyses reveal regulatory mechanisms of milk protein production in dairy cows consuming different forages[J]. Scientific Reports, 2017, 7(1): 44234. DOI:10.1038/srep44234 |
| [32] |
NAEEM A, DRACKLEY J K, STAMEY J, et al. Role of metabolic and cellular proliferation genes in ruminal development in response to enhanced plane of nutrition in neonatal Holstein calves[J]. Journal of Dairy Science, 2012, 95(4): 1807-1820. DOI:10.3168/jds.2011-4709 |
| [33] |
HOU Y Q, WANG L, DING B Y, et al. Dietary α-ketoglutarate supplementation ameliorates intestinal injury in lipopolysaccharide-challenged piglets[J]. Amino Acids, 2010, 39(2): 555-564. DOI:10.1007/s00726-010-0473-y |
| [34] |
LIU W J, CAPUCO A V, ROMAGNOLO D F. Expression of cytosolic NADP+-dependent isocitrate dehydrogenase in bovine mammary epithelium: modulation by regulators of differentiation and metabolic effectors[J]. Experimental Biology and Medicine, 2006, 231(5): 599-610. DOI:10.1177/153537020623100515 |
| [35] |
ABDOUN K, STUMPFF F, MARTENS H. Ammonia and urea transport across the rumen epithelium: a review[J]. Animal Health Research Reviews, 2006, 7(1/2): 43-59. |
| [36] |
NOCEK J E, HERBEIN J H, POLAN C E. Influence of ration physical form, ruminal degradable nitrogen and age on rumen epithelial propionate and acetate transport and some enzymatic activities[J]. The Journal of Nutrition, 1980, 110(12): 2355-2364. DOI:10.1093/jn/110.12.2355 |
| [37] |
RÉMOND D, BERNARD L, PONCET C. Amino acid flux in ruminal and gastric veins of sheep: effects of ruminal and omasal injections of free amino acids and carnosine[J]. Journal of Animal Science, 2000, 78(1): 158-166. DOI:10.2527/2000.781158x |
| [38] |
GARRIGA X, ELIASSON R, TORRENTS E, et al. nrdD and nrdG genes are essential for strict anaerobic growth of Escherichia coli[J]. Biochemical and Biophysical Research Communications, 1996, 229(1): 189-192. DOI:10.1006/bbrc.1996.1778 |
| [39] |
WU Y C, WU Y, ZHU T, et al. Staphylococcus epidermidis SrrAB regulates bacterial growth and biofilm formation differently under oxic and microaerobic conditions[J]. Journal of Bacteriology, 2015, 197(3): 459-476. DOI:10.1128/JB.02231-14 |
| [40] |
LAVERDE GOMEZ J A, MUKHOPADHYA I, DUNCAN S H, et al. Formate cross-feeding and cooperative metabolic interactions revealed by transcriptomics in co-cultures of acetogenic and amylolytic human colonic bacteria[J]. Environmental Microbiology, 2019, 21(1): 259-271. DOI:10.1111/1462-2920.14454 |
| [41] |
REICHARDT N, DUNCAN S H, YOUNG P, et al. Phylogenetic distribution of three pathways for propionate production within the human gut microbiota[J]. The ISME Journal, 2014, 8(6): 1323-1335. DOI:10.1038/ismej.2014.14 |
| [42] |
LOUIS P, FLINT H J. Formation of propionate and butyrate by the human colonic microbiota[J]. Environmental Microbiology, 2017, 19(1): 29-41. DOI:10.1111/1462-2920.13589 |
| [43] |
CHENG Y F, EDWARDS J E, ALLISON G G, et al. Diversity and activity of enriched ruminal cultures of anaerobic fungi and methanogens grown together on lignocellulose in consecutive batch culture[J]. Bioresource Technology, 2009, 100(20): 4821-4828. DOI:10.1016/j.biortech.2009.04.031 |
| [44] |
BAUCHOP T, MOUNTFORT D O. Cellulose fermentation by a rumen anaerobic fungus in both the absence and the presence of rumen methanogens[J]. Applied and Environmental Microbiology, 1981, 42(6): 1103-1110. DOI:10.1128/aem.42.6.1103-1110.1981 |
| [45] |
CHENG Y F, JIN W, MAO S Y, et al. Production of citrate by anaerobic fungi in the presence of co-culture methanogens as revealed by 1H NMR spectrometry[J]. Asian-Australasian Journal of Animal Sciences, 2013, 26(10): 1416-1423. |
| [46] |
KALA A, KAMRA D N, CHAUDHARY L C, et al. Metagenomics and CAZymes in rumen: a review[J]. Indian Journal of Animal Nutrition, 2019, 36(1): 1-10. DOI:10.5958/2231-6744.2019.00001.X |
| [47] |
BERNARDES A, PELLEGRINI V O A, CURTOLO F, et al. Carbohydrate binding modules enhance cellulose enzymatic hydrolysis by increasing access of cellulases to the substrate[J]. Carbohydrate Polymers, 2019, 211: 57-68. |
| [48] |
FLINT H J, SCOTT K P, LOUIS P, et al. The role of the gut microbiota in nutrition and health[J]. Nature Reviews Gastroenterology & Hepatology, 2012, 9(10): 577-589. |
| [49] |
DAI X, TIAN Y, LI J T, et al. Metatranscriptomic analyses of plant cell wall polysaccharide degradation by microorganisms in the cow rumen[J]. Applied and Environmental Microbiology, 2015, 81(4): 1375-1386. |
| [50] |
DEUSCH S, CAMARINHA-SILVA A, CONRAD J, et al. A structural and functional elucidation of the rumen microbiome influenced by various diets and microenvironments[J]. Frontiers in Microbiology, 2017, 8: 1605. |
| [51] |
ZHANG R Y, LIU J H, JIANG L S, et al. The remodeling effects of high-concentrate diets on microbial composition and function in the hindgut of dairy cows[J]. Frontiers in Nutrition, 2021, 8: 809406. |




