草食动物胃肠道中栖居着数以万计的细菌、产甲烷古菌、原虫和真菌等微生物,依靠这些共生的肠道微生物的协同发酵作用,草食动物可将顽固的植物纤维性碳水化合物组分转化为自身可利用的能量(多以挥发性脂肪酸的形式存在)及各种营养物质[1]。在不考虑某些具有特殊消化生理特性的草食动物(如部分食草鱼类等)的情况下,绝大多数的草食动物均进化出了专门的肠室以供肠道微生物栖息,并依靠这些微生物及其相应的功能来维持本身的健康和生产性能[2]。根据这些微生物栖息的肠室及发酵特性的不同可将草食动物分为3种类型:反刍动物(如牛和山/绵羊)、假反刍动物(如羊驼、骆驼和河马)及后肠发酵型草食动物(如兔、马、驴、斑马和大象)[3]。值得一提的是,与后肠发酵型草食动物不同,反刍动物和假反刍动物的前胃微生物也是宿主蛋白质和维生素的重要来源。
在这些微生物中,厌氧真菌由于具有高效且广谱的植物结构性聚合物降解酶体系,且可以通过菌丝的生长而物理性地穿透并破坏植物结构屏障,进而增加其他纤维分解性微生物与细胞内容物等可发酵底物的接触途径,因此在纤维性饲料的消化过程中起着至关重要的作用[4]。尽管厌氧真菌在草食动物肠道纤维消化和微生态中的重要性已被广泛认知和接受,但相较于对细菌和古菌等微生物在草食动物生产性能和环境足迹中发挥作用的认知程度,我们目前对厌氧真菌及其对宿主动物影响的了解仍十分有限。究其原因,是厌氧真菌复杂的生命周期和生物学特性,以及相关研究技术的限制,导致针对此类微生物的研究较少。近年来,先进的分子研究技术提高了对厌氧真菌的检测和分类能力,并为研究厌氧真菌的系统分类和功能多样性提供了基础。对目前尚未深入研究的草食动物厌氧真菌的进一步了解,可能增加我们对厌氧环境下生命活动的科学认知,也可为提高植物性饲料的转化效率和宿主动物的生产性能以及降低畜牧生产对环境造成的影响开辟新的途径。本文将从系统发育分类学发展史、相关研究方法及已培养鉴定的菌属等方面来论述目前对厌氧真菌系统分类学的理解,以期能为行业工作者的相关研究提供一定的参考。
1 厌氧真菌系统发育分类学发展史厌氧真菌及其分类体系的探索和研究已经持续了1个多世纪。厌氧真菌最早发现于20世纪初,但当时其具鞭毛游动孢子被错误地鉴定为鞭毛原虫,尽管这种具鞭毛游动孢子要比从瘤胃中分离得到的鞭毛虫小得多[5]。随后直至20世纪70年代,基于Orpin[6]多年收集的重要证据,如厌氧真菌生命周期包含不同的生命阶段[6]、几丁质是其细胞壁的主要结构性多糖[7]等,它们才被正确定性为厌氧真菌。这打破了当时人们普遍认为的真菌在本质上是高度需氧且在缺氧条件下无法代谢碳水化合物的真菌学教条。
厌氧真菌最初被归类于壶菌门(Chytridiomycetes)小壶菌目(Spizellomycetales)下[8],且该分类方法可由其18S核糖体RNA(ribosome RNA,rRNA)序列所佐证[9]。尽管如此,厌氧真菌独特的表型特征,如严格的厌氧生活方式、无线粒体、具氢化酶复合体和多鞭毛运动孢子等,在其他小壶菌目真菌中并未发现,故又创建了一个新的菌目——新毛孢菌目(Neocallimastigales),作为厌氧真菌的生物学分类群[10]。20世纪90年代后,随着分子生物学的发展,基于标准的多基因位点数据库、广泛的分类单元抽样和严格的生物信息学分析方法的真菌分子系统分类学逐渐成为真菌的标准分类方法。21世纪初,采用多基因法的“组装真菌生命之树”项目探究了真菌界高水平的系统发育进化关系,极大地促进了真菌系统分类学研究;该项目共采用6个基因来评估真菌的系统发育学分类信息,包括3个rRNA基因(18S rRNA、28S rRNA和5.8S rRNA)和3个蛋白质编码基因[翻译延伸因子1-α(EF1-α)、RNA聚合酶Ⅱ最大亚基(RPB1)、RNA聚合酶Ⅱ次大亚基(RPB2)][11]。基于该项目的研究成果,以及瘤胃厌氧真菌独特的形态特征,导致厌氧真菌从壶菌门中分离了出来,形成了一个新的真菌门——新毛孢菌门(Neocallimastigomycota)[12]。随后,一项类似的基于RPB1和3个rRNA基因的研究结果[13],以及另一项基于99个基因组和109个表达序列标签数据集的真菌系统发育学研究[14],均证实了新毛孢菌门具有单系起源,是壶菌门的姊妹菌群。最近,Tedersoo等[15]基于分子系统发育学特性、分化时间和单系标准在较高等级上对真菌进行了分类学研究,结果表明,新毛孢菌门与壶菌门和单眼菌门(Monoblepharomycota)为姊妹菌群,作者还引入了更高的真菌分类水平——壶菌亚界(Chytridiomyceta),来安置上述3个菌门。另外,有研究显示新毛孢菌门是分化较早的一类真菌,大约在6 600万年前就已分化产生,这与预估的草食哺乳动物的进化时间相符,表明厌氧真菌可能相伴进化产生于草食动物胃肠道环境中[16]。
在纲、目、科水平上,各项研究的结果比较一致,均认为新毛孢菌门由新毛孢菌纲(Neocallimastigomycetes)、新毛孢菌目和新毛孢菌科(Neocallimastigaceae)构成。更低水平的厌氧真菌分类信息将在后文进行详细地阐述。
2 厌氧真菌较低水平的分类学多样性与较高水平的系统发育分类有所不同,厌氧真菌即新毛孢菌门内部的多样性最初主要是采用体外纯化培养法,基于菌种的形态特征和代谢特性而进行区分的。区分属水平的形态特征主要包括菌丝体和假根的类型、孢子囊形状及发育程序、游动孢子的鞭毛数量等。然而,由于厌氧真菌具有形态变异广泛、孢子囊和假根结构存在同质多形现象、单中心和单鞭毛属的形态结构相似、不一定能产生孢子囊以及一些多中心菌种中缺少游动孢子发生现象等特性[17],仅依靠形态特征对厌氧真菌进行系统性地分类困难重重。因此,随着分子生物学的发展,结合形态特征和遗传特性基础的分类方法逐渐成为低水平厌氧真菌分类鉴定的黄金标准。
上文提到“组装真菌生命之树”项目利用3个rRNA基因和3个蛋白质编码基因来研究真菌高水平的系统进化发育关系,那么哪些基因适宜用作低水平厌氧真菌的标记物呢?首先我们需要明确这些可用于系统发育分类分析的标记基因的特点。常用的蛋白质编码基因如RPB1、RPB2通常是单拷贝的,且能编码极其重要的功能性蛋白质,因此这些基因的突变会导致菌体失去环境适应性乃至死亡,故这些基因通常高度保守[11]。因此,这些蛋白质编码基因通常可用于较高水平真菌的系统发育学分类,但在区分属种水平上的近源真菌时用处不大。相反地,包含3个常用的rRNA基因的rRNA操纵子在真菌基因组具有很高的拷贝数(约200个),这意味着只需要少量的组织或环境样品便可有效地进行PCR扩增;此外,这些基因位点并不编码蛋白质,因此可具有相对较高的突变率,进而具有良好的系统发育学分辨能力[12]。因此,源自rRNA操纵子的基因适用于较低水平真菌的系统发育学分类分析,而先前有关低水平厌氧真菌多样性的研究中所使用的标记基因也集中在rRNA操纵子的不同区域。
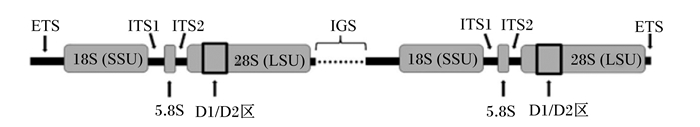
2.1 真核生物rRNA操纵子的拓扑结构真核生物的rRNA操纵子由RNA聚合酶Ⅰ作为1个转录单元而转录,通常分为小亚基rRNA基因(SSU)、大亚基rRNA基因(LSU)和内部转录间隔区(ITS)3个区域,而ITS区域则由转录后修饰过程移除的2个区域(ITS1、ITS2)和5.8S rRNA基因构成(图 1)[13]。厌氧真菌rRNA操纵子详细的拓扑结构图也可参考Edwards等[18]。
|
ETS:外部转录间隔区external transcribed spacer;SSU:小亚基rRNA基因small subunit rRNA gene;ITS:内部转录间隔区internal transcribed spacer;LSU:大亚基rRNA基因large subunit rRNA gene;IGS:基因间隔区intergenic spacer。 图 1 真核生物rRNA操纵子的拓扑结构 Fig. 1 Topological structure of rRNA operon in eukaryotes[17] |
尽管已有不少研究使用真核生物SSU(18S rRNA基因)作为厌氧真菌和原虫的分类学鉴定和定量位点[19-20],且其同源基因(16S rRNA基因)也已被作为标记基因广泛用于细菌和古菌较低水平(属/种)的分类学鉴定,但由于真核生物SSU序列相对保守,其变异程度不足以有效区分属水平厌氧真菌的系统发育关系[19]。更短的5.8S rRNA基因(约185 bp)也由于相同的原因无法作为厌氧真菌低水平的分类学标记基因。因此,迄今大多数有关厌氧真菌系统发育分类和多样性分析的研究大都使用ITS区域的部分序列作为标记基因,尤其是ITS1区域;另外,最近一些研究开始转向使用LSU的部分区域(D1/D2区)作为厌氧真菌的标记基因。后文将详细论述二者作为厌氧真菌标记基因的应用及其优劣。
2.2 ITSITS区域序列变异丰富,能够在属/种水平上区分真菌的系统发育信息,是真菌界尤其是早期分化真菌较好的系统发育学标记基因,被广泛用于培养物或各种生境中厌氧真菌的分类学鉴定和多样性研究[21-23]。在ITS区域中,ITS1和ITS2区域均曾被用作厌氧真菌的系统发育学标记基因,但ITS1区域蕴含的系统发育信息较ITS2区域更为丰富,因此使用的更为普遍[24]。在过去的10年间,基于ITS1区域的高通量测序分析,研究人员对源于不同宿主动物的厌氧真菌的多样性和群落结构进行了相当多的研究[25-26]。然而,通过扩增子测序获得的ITS1区域序列长度有限,极大地限制了基于其进行可靠的系统发育分析的能力,同时厌氧真菌ITS1区域存在着大规模的单核苷酸多态性,因此通常使用基于可操作分类单元(OTUs)的方式来进行系统发育分类学分析[26]。迄今为止,大量的ITS1区域的代表性OTUs序列被存储在公共数据库如GenBank[27]、UNITE[28]、ITSoneDB[29]和PHYMYCO-DB[30]之中。研究人员可以这些数据为参比,在高通量数据分析平台(如QIIME2、Mothur等)通过一定的算法基于序列相似度来对未知序列进行分类学注释。理想状态下,该过程需要1个以完全测序的参比基因组为基础构建的稳定的厌氧真菌系统发育体系。然而,这些数据库的质量严重依赖相关序列的数量及贡献者的科学严谨性,加之ITS1区域存在普遍的单核苷酸多态性及异源同质现象,且没有足够数量的参比基因组构建稳定的系统发育体系,因此不难解释早期参比数据集中存在相当多的序列在种属水平上被错误命名的现象[18, 21]。
为了克服早期缺乏稳定的厌氧真菌系统发育体系的缺点,科研工作者曾试图通过利用二级结构信息来辅助构建厌氧真菌系统发育体系以改善ITS1区域序列分析的可靠性[31]。随后,Koetschan等[26]提出厌氧真菌的ITS1区域具有一个共同的二级核心结构,并开发了一种自动折叠和对齐方法,进而构建一种更为稳定的ITS1区域基础的厌氧真菌系统发育体系,该体系将当时发现的所有厌氧真菌归类到8个属和12个尚未被培养的属/种水平的分类簇中。类似地,一些研究也利用二级结构信息基于ITS2区域序列来构建更准确有效的厌氧真菌系统发育树[32]。在最近的基于ITS1区域构建的厌氧真菌系统发育体系中,Paul等[24]将厌氧真菌分为34个属水平分类簇和119个种水平分类簇,其中25个属、81个种水平分类簇当时尚未得到培养。由于近几年有关厌氧真菌纯培养的研究报道不断涌现,其中一些分类簇得到了重新正式的命名[33-36]。
尽管ITS1区域迄今在厌氧真菌系统发育分类及多样性的研究中得到了十分广泛地应用,但其本身也有十分严重的缺陷。首先,如前所述,ITS1区域存在着显著的单核苷酸多态性,体现在利用单一培养株克隆得到的ITS1区域序列具有很高的变异性,甚至高达13%[23]。这种高变异性增加了对新分离株进行分类学鉴定的难度,同时很难确定一个源自环境中的未知ITS1区域序列是否代表一个新的厌氧真菌属/种。其次,ITS1区域的序列长度本身大小不一且变异较大[37],且最近的研究表明序列长度越小的ITS1区域的扩增优先级越高[38],这会影响环境样本中厌氧真菌群落结构评估的准确性。再次,扩增结果受引物的影响很大,即使是广泛使用的引物序列(如MN100F)也并非在所有的厌氧真菌中均是保守的[23]。由于这些缺陷的存在,以及基于ITS1区域构建的系统发育体系的不稳定性,科研工作者近年来开始探索使用LSU作为厌氧真菌标记基因的可能性。然而,短期内ITS1区域仍然是非常重要的厌氧真菌标记基因,尤其是旨在确定环境样品中包括新毛孢菌门在内的真菌群落结构特征的情况下。
2.3 LSULSU是rRNA操纵子中最长的基因,它编码核糖体28S rRNA亚基,在厌氧真菌中长度约为3 500 bp。它与ITS2区域相邻的上游5’区域,即D1/D2区,具有显著的变异性,能够提供丰富的系统发育信息,因此可用作真菌的标记基因[13, 39-40]。同时,其双侧翼区域序列非常保守,因此可以进行通用引物[40]或菌群特异性引物[19]的设计。此外,与ITS1区域不同,该区域序列相对比较保守,且在菌株基因组内部的变异有限,即单培养株扩增得到标记基因的一致性较高,能够构建更稳定的系统发育体系。最近的一篇研究报道显示,尽管基于LSU和ITS1区域的系统发育体系具有高度的相似性,但是LSU比ITS1区域的序列更容易对齐,且在厌氧真菌属水平上的分类学分辨率更高[41]。因此,与ITS1区域相比,LSU的D1/D2区可能是更好的厌氧真菌系统发育学标记基因。
Hausner等[42]率先开发了一种针对厌氧真菌28S rRNA基因的限制性片段长度多态性聚合酶链反应(PCR-RFLP)基础的方法,但是由于存在多个酶切位点,该1.65 kbp长的扩增子片段对于酶切定性的价值较为有限。随后,Fliegerova等[43]使用真菌通用引物对NL1/NL4扩增,成功地获得了仅覆盖厌氧真菌LSU的D1/D2区的相对较小的扩增子(约750 bp)。后来的研究证明相同的区域和方法能够区分2个密切相关的厌氧真菌物种[39],表明该区域有作为厌氧真菌标记基因片段的潜力。其后的数个研究均同时利用LSU和ITS1区域基础的系统发育关系对新发现的厌氧真菌进行了属水平分类学鉴定[22-23, 33-36, 44]。这些研究还基于LSU的D1/D2区建立了系统发育树,能够区分目前已鉴定的所有厌氧真菌属的系统发育关系。
然而,厌氧真菌LSU的D1/D2区全长约750 bp,故目前尚不能直接通过二代测序探究环境样品中厌氧真菌群落结构,仅适用于通过一代Sanger测序来对厌氧真菌进行鉴定并分析它们之间的系统发育关系。目前有一些研究探究了适用于短读长高通量测序的特异性引物[19, 45],并将之应用于某些环境样品的厌氧真菌多样性研究中[46-47]。然而,目前尚不清楚这些引物是否能够覆盖所有的厌氧真菌,同时还缺乏一个足够大的、包含目前已鉴定的所有厌氧真菌分类群及其模式菌株的LSU的D1/D2区序列的参考数据库[41]。此外,如何将LSU序列同目前尚未有代表性培养株的分类簇、以及由来源于某些环境中的ITS1区域序列分析得到的分类簇相联系,也是LSU应用于厌氧真菌系统发育学和多样性研究中所面临的重要挑战。
解决这些问题的关键是需要不断地拓展和整理厌氧真菌LSU参考数据库。另外,最新的三代高通量测序技术(如PacBio SMRT和Nanopore MinION)可产生更长的读子,理论上提供了另外一种解决这些问题的方案。应用三代高通量测序技术,可以获得覆盖整个厌氧真菌LSU的D1/D2区的读子,直接进行系统发育和多样性分析;也可进行rRNA操纵子全长序列测序,将ITS1区域序列同LSU进行整合分析,或建立整合多个标记基因的更复杂的参考数据库[48-49]。当然,要真正准确地解决上述所有问题,在忽略成本和时间的情况下,最好的办法是将整个基因组作为分子标记[18]。
综上可知,尽管LSU的D1/D2区理论上是更加先进有效的厌氧真菌系统发育标记基因,但由于技术和研究方法等的限制,目前其仅被广泛应用于培养菌株的鉴定和有限的系统发育分析研究中。因此,ITS1区域目前仍是学界最为广泛接受的厌氧真菌系统发育学标记基因,且在未来一段时间内仍将继续用于评估环境样品中厌氧真菌多样性和群落结构的主要标记基因。然而毋庸置疑的是,ITS1区域的局限性使得学界势必会持续推进开发更加准确、分辨率更高的分类方法,而目前看来将LSU的D1/D2区作为厌氧真菌的系统发育标记基因是可预见的行之有效的方向。有关ITS1区域和LSU的D1/D2区序列数据库和相关的分类文件[26, 49],包括新近发现的厌氧真菌属,读者可查阅相关网站(https://www.anaerobicfungi.org)。
3 属水平厌氧真菌分类信息上文提到,厌氧真菌直至1975年才被Orpin[6]准确地定性和描述。而在随后的近40年间,仅有6个属的厌氧真菌得以鉴定和描述,统一被归类于新毛孢菌门下[4]。随着厌氧真菌在纤维降解方面的功能和重要性被逐步认知,近5年共分离、培养和鉴定出13个特征属及其模式菌种[22-23, 33-36, 44]。截至目前,共有19个厌氧真菌属、约40个模式菌种得以鉴定和描述,其名称、形态学特征等信息被归纳于表 1中。此外,一项基于ITS1区域序列的、利用非培养学方法研究全球肠道真菌系统发育学多样性的报道显示,新毛孢菌门一共存在34个属水平分类簇和119个种水平分类簇[24],表明目前尚有相当数量的厌氧真菌未得到分离鉴定。
|
|
表 1 目前已鉴定的属水平厌氧真菌的重要形态特征 Table 1 Key morphological features of currently characterized anaerobic fungal genera |
形态特征是决定厌氧真菌分类的关键因素,区分不同厌氧真菌属的关键形态特征包括菌丝体的发育机制,假根、孢子囊和孢囊柄的形状,以及所产生游动孢子的鞭毛数量等。在已经培养鉴定的19个属中,17个属的假根呈高度分枝的丝状体,仅盲肠真菌属(Caecomyces)和肠道真菌属(Cyllamyces)的假根呈球状体。在丝状体假根的新毛孢菌中,仅厌氧真菌属(Anaeromyces)和奥宾氏真菌属(Orpinomyces)的菌丝体是多中心的,其余15个属均为单中心的;而形成2个球状体假根的盲肠真菌属和肠道真菌属则分别形成单中心和多中心的菌丝体。此外,除了新毛孢菌属(Neocallimastix)、非驯化真菌属(Feramyces)、鹿源真菌属(Ghazallomyces)、夏季牧场真菌属(Aestipascuomyces)和奥宾氏真菌属产生多鞭毛游动孢子外,其他属产生的游动孢子多以单鞭毛为主。
4 小结与展望厌氧真菌以其强大的纤维降解酶体系和侵袭性生长特性,在饲料降解和消化过程中发挥着重要作用。基于特定系统发育标记基因的分子生物学方法的出现和发展,克服了之前基于形态特征进行分类的缺点,极大地促进了厌氧真菌的系统发育分类研究进程。在潜在的系统发育标记基因中,ITS1区域目前是普遍接受的厌氧真菌系统发育学标记基因,且可能在短时间内仍将广泛用于环境样品中厌氧真菌多样性和群落结构的评估研究中。然而,其本身的局限性使得学界势必会持续推进开发更加准确、分辨率更高的分类方法,其中LSU的D1/D2区非常有潜力成为未来厌氧真菌系统发育和多样性研究的标记基因。尽管我们已经对厌氧真菌的多样性有了更深的了解,目前已经有19个菌属得以鉴定,但毫无疑问的是动物胃肠道微生态系统中仍存在较多属水平的厌氧真菌有待描述。考虑到其本身的消化代谢特性,结合最近报道的其多样性和菌群结构同宿主遗传和饲料特性之间的密切相关性,厌氧真菌在畜牧生产中具有较大的研究和应用潜力,可能是未来一段时间内草食动物肠道微生物研究的热点之一。
| [1] |
DEARING M D, KOHL K D. Beyond fermentation: other important services provided to endothermic herbivores by their gut microbiota[J]. Integrative and Comparative Biology, 2017, 57(4): 723-731. DOI:10.1093/icb/icx020 |
| [2] |
MORGAVI D P, KELLY W J, JANSSEN P H, et al. Rumen microbial (meta) genomics and its application to ruminant production[J]. Animal, 2013, 7(Suppl.1): 184-201. |
| [3] |
DEHORITY B A. Gastrointestinal tracts of herbivores, particularly the ruminant: anatomy, physiology and microbial digestion of plants[J]. Journal of Applied Animal Research, 2002, 21(2): 145-160. DOI:10.1080/09712119.2002.9706367 |
| [4] |
GRUNINGER R J, PUNIYA A K, CALLAGHAN T M, et al. Anaerobic fungi (phylum Neocallimastigomycota): advances in understanding their taxonomy, life cycle, ecology, role and biotechnological potential[J]. FEMS Microbiology Ecology, 2014, 90(1): 1-17. DOI:10.1111/1574-6941.12383 |
| [5] |
JENSEN E A, HAMMOND D M. A morphological study of trichomonads and related flagellates from the bovine digestive tract[J]. The Journal of Protozoology, 1964, 11(3): 386-394. DOI:10.1111/j.1550-7408.1964.tb01768.x |
| [6] |
ORPIN C G. Studies on the rumen flagellate Neocallimastix frontalis[J]. Journal of General Microbiology, 1975, 91(2): 249-262. DOI:10.1099/00221287-91-2-249 |
| [7] |
ORPIN C G. The rumen flagellate Piromonas communis: its life-history and invasion of plant material in the rumen[J]. Journal of General Microbiology, 1977, 99(1): 107-117. DOI:10.1099/00221287-99-1-107 |
| [8] |
BARR D J S. An outline for the reclassification of the Chytridiales, and for a new order, the Spizellomycetales[J]. Canadian Journal of Botany, 1980, 58(22): 2380-2394. DOI:10.1139/b80-276 |
| [9] |
DORE J, STAHL D A. Phylogeny of anaerobic rumen Chytridiomycetes inferred from small subunit ribosomal RNA sequence comparisons[J]. Canadian Journal of Botany, 1991, 69(9): 1964-1971. DOI:10.1139/b91-246 |
| [10] |
LI J L, HEATH I B, PACKER L. The phylogenetic relationships of the anaerobic chytridiomycetous gut fungi (Neocallimasticaceae) and the Chytridiomycota.Ⅱ.Cladistic analysis of structural data and description of Neocallimasticales ord.nov.[J]. Canadian Journal of Botany, 1993, 71(3): 393-407. DOI:10.1139/b93-044 |
| [11] |
JAMES T Y, KAUFF F, SCHOCH C L, et al. Reconstructing the early evolution of fungi using a six-gene phylogeny[J]. Nature, 2006, 443(7113): 818-822. DOI:10.1038/nature05110 |
| [12] |
HIBBETT D S, BINDER M, BISCHOFF J F, et al. A higher-level phylogenetic classification of the fungi[J]. Mycological Research, 2007, 111(Pt 5): 509-547. |
| [13] |
SCHOCH C L, SEIFERT K A, HUHNDORF S, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(16): 6241-6246. DOI:10.1073/pnas.1117018109 |
| [14] |
EBERSBERGER I, DE MATOS SIMOES R, KUPCZOK A, et al. A consistent phylogenetic backbone for the fungi[J]. Molecular Biology and Evolution, 2012, 29(5): 1319-1334. DOI:10.1093/molbev/msr285 |
| [15] |
TEDERSOO L, SÁNCHEZ-RAMÍREZ S, KÕLJALG U, et al. High-level classification of the Fungi and a tool for evolutionary ecological analyses[J]. Fungal Diversity, 2018, 90(1): 135-159. DOI:10.1007/s13225-018-0401-0 |
| [16] |
WANG Y, YOUSSEF N H, COUGER M B, et al. Molecular dating of the emergence of anaerobic rumen fungi and the impact of laterally acquired genes[J]. mSystems, 2019, 4(4): e00247-19. |
| [17] |
HESS M, PAUL S S, PUNIYA A K, et al. Anaerobic fungi: past, present, and future[J]. Frontiers in Microbiology, 2020, 11: 584893. DOI:10.3389/fmicb.2020.584893 |
| [18] |
EDWARDS J E, FORSTER R J, CALLAGHAN T M, et al. PCR and omics based techniques to study the diversity, ecology and biology of anaerobic fungi: insights, challenges and opportunities[J]. Frontiers in Microbiology, 2017, 8: 1657. DOI:10.3389/fmicb.2017.01657 |
| [19] |
DOLLHOFER V, CALLAGHAN T M, DORN-IN S, et al. Development of three specific PCR-based tools to determine quantity, cellulolytic transcriptional activity and phylogeny of anaerobic fungi[J]. Journal of Microbiological Methods, 2016, 127: 28-40. DOI:10.1016/j.mimet.2016.05.017 |
| [20] |
ZHANG Y W, LI F Y, CHEN Y H, et al. Metatranscriptomic profiling reveals the effect of breed on active rumen eukaryotic composition in beef cattle with varied feed efficiency[J]. Frontiers in Microbiology, 2020, 11: 367. DOI:10.3389/fmicb.2020.00367 |
| [21] |
KITTELMANN S, NAYLOR G E, KOOLAARD J P, et al. A proposed taxonomy of anaerobic fungi (class Neocallimastigomycetes) suitable for large-scale sequence-based community structure analysis[J]. PLoS One, 2012, 7(5): e36866. DOI:10.1371/journal.pone.0036866 |
| [22] |
DAGAR S S, KUMAR S, GRIFFITH G W, et al. A new anaerobic fungus (Oontomyces anksri gen. nov., sp. nov.) from the digestive tract of the Indian camel (Camelus dromedarius)[J]. Fungal Biology, 2015, 119(8): 731-737. DOI:10.1016/j.funbio.2015.04.005 |
| [23] |
CALLAGHAN T M, PODMIRSEG S M, HOHLWECK D, et al. Buwchfawromyces eastonii gen. nov., sp. nov.: a new anaerobic fungus (Neocallimastigomycota) isolated from buffalo faeces[J]. MycoKeys,, 2015, 9: 11-28. DOI:10.3897/mycokeys.9.9032 |
| [24] |
PAUL S S, BU D P, XU J C, et al. A phylogenetic census of global diversity of gut anaerobic fungi and a new taxonomic framework[J]. Fungal Diversity, 2018, 89(1): 253-266. DOI:10.1007/s13225-018-0396-6 |
| [25] |
LIGGENSTOFFER A S, YOUSSEF N H, COUGER M B, et al. Phylogenetic diversity and community structure of anaerobic gut fungi (phylum Neocallimastigomycota) in ruminant and non-ruminant herbivores[J]. The ISME Journal, 2010, 4(10): 1225-1235. DOI:10.1038/ismej.2010.49 |
| [26] |
KOETSCHAN C, KITTELMANN S, LU J L, et al. Internal transcribed spacer 1 secondary structure analysis reveals a common core throughout the anaerobic fungi (Neocallimastigomycota)[J]. PLoS One, 2014, 9(3): e91928. DOI:10.1371/journal.pone.0091928 |
| [27] |
BENSON D A, CAVANAUGH M, CLARK K, et al. GenBank[J]. Nucleic Acids Research, 2013, 41(Database Issue): D36-D42. |
| [28] |
KÕLJALG U, NILSSON R H, ABARENKOV K, et al. Towards a unified paradigm for sequence-based identification of fungi[J]. Molecular Ecology, 2013, 22(21): 5271-5277. DOI:10.1111/mec.12481 |
| [29] |
SANTAMARIA M, FOSSO B, CONSIGLIO A, et al. Reference databases for taxonomic assignment in metagenomics[J]. Briefings in Bioinformatics, 2012, 13(6): 682-695. DOI:10.1093/bib/bbs036 |
| [30] |
MAHÉ S, DUHAMEL M, LE CALVEZ T, et al. PHYMYCO-DB: a curated database for analyses of fungal diversity and evolution[J]. PLoS One, 2012, 7(9): e43117. DOI:10.1371/journal.pone.0043117 |
| [31] |
TUCKWELL D S, NICHOLSON M J, MCSWEENEY C S, et al. The rapid assignment of ruminal fungi to presumptive genera using ITS1 and ITS2 RNA secondary structures to produce group-specific fingerprints[J]. Microbiology, 2005, 151(Pt 5): 1557-1567. |
| [32] |
KELLER A, FÖRSTER F, MVLLER T, et al. Including RNA secondary structures improves accuracy and robustness in reconstruction of phylogenetic trees[J]. Biology Direct, 2010, 5: 4. DOI:10.1186/1745-6150-5-4 |
| [33] |
HANAFY R A, ELSHAHED M S, YOUSSEF N H. Feramyces austinii, gen. nov., sp. nov., an anaerobic gut fungus from rumen and fecal samples of wild Barbary sheep and fallow deer[J]. Mycologia, 2018, 110(3): 513-525. DOI:10.1080/00275514.2018.1466610 |
| [34] |
JOSHI A, LANJEKAR V B, DHAKEPHALKAR P K, et al. Liebetanzomycespolymorphus gen. et sp. nov., a new anaerobic fungus (Neocallimastigomycota) isolated from the rumen of a goat[J]. MycoKeys, 2018, 40: 89-110. DOI:10.3897/mycokeys.40.28337 |
| [35] |
STABEL M, HANAFY R A, SCHWEITZER T, et al. Aestipascuomyces dupliciliberans gen. nov, sp. nov., the first cultured representative of the uncultured SK4 clade from aoudad sheep and alpaca[J]. Microorganisms, 2020, 8(11): 1734. DOI:10.3390/microorganisms8111734 |
| [36] |
HANAFY R A, LANJEKAR V B, DHAKEPHALKAR P K, et al. Seven new Neocallimastigomycota genera from wild, zoo-housed, and domesticated herbivores greatly expand the taxonomic diversity of the phylum[J]. Mycologia, 2020, 112(6): 1212-1239. DOI:10.1080/00275514.2019.1696619 |
| [37] |
EDWARDS J E, KINGSTON-SMITH A H, JIMENEZ H R, et al. Dynamics of initial colonization of nonconserved perennial ryegrass by anaerobic fungi in the bovine rumen[J]. FEMS Microbiology Ecology, 2008, 66(3): 537-545. DOI:10.1111/j.1574-6941.2008.00563.x |
| [38] |
EDWARDS J E, HERMES G D A, KITTELMANN S, et al. Assessment of the accuracy of high-throughput sequencing of the ITS1 region of Neocallimastigomycota for community composition analysis[J]. Frontiers in Microbiology, 2019, 10: 2370. DOI:10.3389/fmicb.2019.02370 |
| [39] |
DAGAR S S, KUMAR S, MUDGIL P, et al. D1/D2 domain of large-subunit ribosomal DNA for differentiation of Orpinomyces spp[J]. Applied and Environmental Microbiology, 2011, 77(18): 6722-6725. DOI:10.1128/AEM.05441-11 |
| [40] |
DETHERIDGE A P, BRAND G, FYCHAN R, et al. The legacy effect of cover crops on soil fungal populations in a cereal rotation[J]. Agriculture Ecosystems & Environment, 2016, 228: 49-61. |
| [41] |
WANG X W, LIU X Z, GROENEWALD J Z. Phylogeny of anaerobic fungi (phylum Neocallimastigomycota), with contributions from yak in China[J]. Antonie Van Leeuwenhoek, 2017, 110(1): 87-103. DOI:10.1007/s10482-016-0779-1 |
| [42] |
HAUSNER G, INGLIS G D, YANKE L J, et al. Analysis of restriction fragment length polymorphisms in the ribosomal DNA of a selection of anaerobic chytrids[J]. Canadian Journal of Botany, 2000, 78(7): 917-927. DOI:10.1139/b00-067 |
| [43] |
FLIEGEROVA K, MRAZEK J, VOIGT K. Differentiation of anaerobic polycentric fungi by rDNA PCR-RFLP[J]. Folia Microbiologica, 2006, 51(4): 273-277. DOI:10.1007/BF02931811 |
| [44] |
HANAFY R A, ELSHAHED M S, LIGGENSTOFFER A S, et al. Pecoramyces ruminantium, gen. nov., sp. nov., an anaerobic gut fungus from the feces of cattle and sheep[J]. Mycologia, 2017, 109(2): 231-243. DOI:10.1080/00275514.2017.1317190 |
| [45] |
NAGLER M, PODMIRSEG S M, GRIFFITH G W, et al. The use of extracellular DNA as a proxy for specific microbial activity[J]. Applied Microbiology and Biotechnology, 2018, 102(6): 2885-2898. DOI:10.1007/s00253-018-8786-y |
| [46] |
DOLLHOFER V, CALLAGHAN T M, GRIFFITH G W, et al. Presence and transcriptional activity of anaerobic fungi in agricultural biogas plants[J]. Bioresource Technology, 2017, 235: 131-139. DOI:10.1016/j.biortech.2017.03.116 |
| [47] |
NAGLER M, KOZJEK K, ETEMADI M, et al. Simple yet effective: microbial and biotechnological benefits of rumen liquid addition to lignocellulose-degrading biogas plants[J]. Journal of Biotechnology, 2019, 300: 1-10. DOI:10.1016/j.jbiotec.2019.05.004 |
| [48] |
WURZBACHER C, LARSSON E, BENGTSSON-PALME J, et al. Introducing ribosomal tandem repeat barcoding for fungi[J]. Molecular Ecology Resources, 2019, 19(1): 118-127. DOI:10.1111/1755-0998.12944 |
| [49] |
HANAFY R A, JOHNSON B, YOUSSEF N H, et al. Assessing anaerobic gut fungal diversity in herbivores using D1/D2 large ribosomal subunit sequencing and multi-year isolation[J]. Environmental Microbiology, 2020, 22(9): 3883-3908. DOI:10.1111/1462-2920.15164 |
| [50] |
HEATH I B, BAUCHOP T, SKIPP R A. Assignment of the rumen anaerobe Neocallimastix frontalis to the Spizellomycetales (Chytridiomycetes) on the basis of its polyflagellate zoospore ultrastructure[J]. Canadian Journal of Botany, 1983, 61(1): 295-307. DOI:10.1139/b83-033 |
| [51] |
BARR D J S, KUDO H, JAKOBER K D, et al. Morphology and development of rumen fungi: Neocallimastix sp., Piromyces communis, and Orpinomyces bovis gen.nov., sp.nov.[J]. Canadian Journal of Botany, 1989, 67(9): 2815-2824. DOI:10.1139/b89-361 |
| [52] |
BRETON A, BERNALIER A, DUSSER M, et al. Anaeromyces mucronatus nov. gen., nov. sp.A new strictly anaerobic rumen fungus with polycentric thallus[J]. FEMS Microbiology Letters, 1990, 58(2): 177-182. |
| [53] |
GOLD J J, HEATH I B, BAUCHOP T. Ultrastructural description of a new chytrid genus of caecum anaerobe, Caecomyces equi gen. nov., sp. nov., assigned to the Neocallimasticaceae[J]. Bio Systems, 1988, 21(3/4): 403-415. |
| [54] |
OZKOSE E, THOMAS B J, DAVIES D R, et al. Cyllamyces aberensis gen.nov.sp.nov., a new anaerobic gut fungus with branched sporangiophores isolated from cattle[J]. Canadian Journal of Botany, 2001, 79(6): 666-673. DOI:10.1139/b01-047 |



