厌氧真菌(新毛孢菌门,Neocallimastigomycota)广泛存在于草食哺乳动物的瘤胃及后肠道中,在纤维性饲料原料的消化过程中起至关重要的作用[1]。厌氧真菌是动物摄食的植物性饲料原料首要定植微生物,其通过菌丝顶端的高活性纤维降解酶及生长过程中产生的机械穿透力来破坏纤维性饲料,进而增加其他类型的纤维降解微生物获得碳水化合物的途径和可能性。先前的研究表明,尽管厌氧真菌仅占肠道微生物数量7%~9%,但它们能从动物摄入的植物性饲料原料中释放出超过50%的可发酵糖类物质[2-3];而体外研究显示,厌氧真菌甚至能在4 d的培养期内从未经处理的植物叶片中释放出高达95%的可发酵糖类物质[4]。
在1975年Orpin[5]首次准确定性厌氧真菌后的40年间,仅有6个属得以鉴定和命名[1]。近些年,随着研究者逐渐意识到厌氧真菌在草食动物胃肠道纤维发酵中的重要作用及其在生物技术领域巨大的应用潜力,使其受到了广泛的关注和研究,近5年共有14个新的菌属得到鉴定和命名[6-7]。同时,由于基因组、转录组、蛋白质组及代谢组等组学技术的广泛应用,使得我们对厌氧真菌生物学特性的了解和认识日趋深入,取得了相当大的进展。在此基础上,本文拟从生命周期、酶学特性和代谢特性等方面对厌氧真菌的生物学特性做一个简单的综述,以期能为相关研究人员提供一定的启发和帮助。
1 厌氧真菌的生命周期目前对厌氧真菌生命周期的理解主要是基于研究瘤胃厌氧真菌而获得的。与大多数真菌不同,厌氧真菌在其生命周期中会进行独特的变态发育过程,即通过无性繁殖在动态的游动孢子和静态的菌体之间交替变换(图 1)。在瘤胃中,新摄入的植物性饲料会释放出大量的水溶性血红素和其他卟啉类物质以及某些不稳定的未知植物成分等,这些物质可诱导成熟孢子囊中孢子的增殖分化及孢囊壁的溶解破裂,从而释放出游动孢子[8]。这些具鞭毛的游动孢子在鞭毛的推动下,基于趋化反应向可溶性糖[9]和/或酚酸[10]丰富的地方运动,而此类物质通常由植物性饲料原料的气孔或破碎的组织向外释放,最终促使游动孢子接触并优先附着在这些部位上[9, 11]。研究表明尽管游动孢子可以游动存活达数小时[12],但它们往往在从孢子囊释放后1 h内便附着在植物碎片上,这对其与瘤胃细菌等其他瘤胃微生物竞争性生长至关重要[13]。
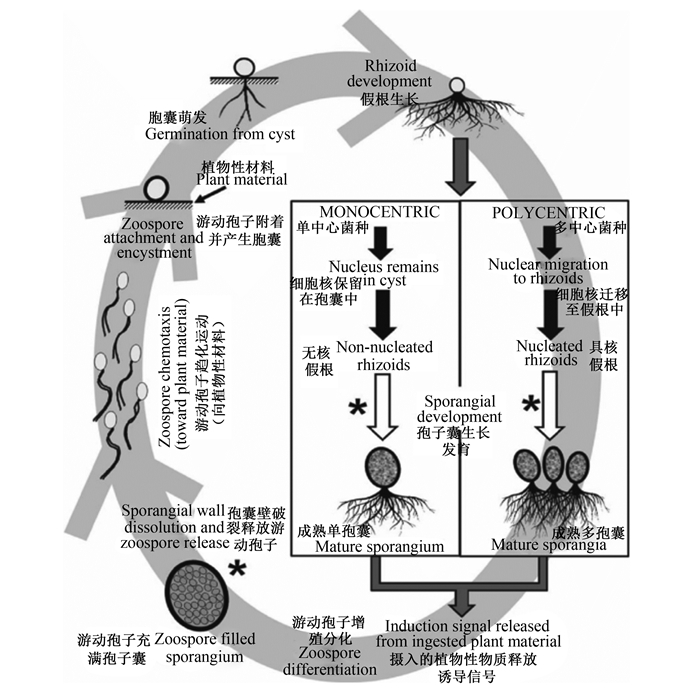
|
*代表目前已经报道的在厌氧真菌生命周期中可能会形成“抗性”结构的阶段。 * indicated the 'resistant' structures may be formed in anaerobic fungal life cycle that have been reported. 图 1 厌氧真菌生命周期概略图 Fig. 1 Summary of anaerobic fungal life cycle[1] |
在游动孢子附着于植物碎片后,其鞭毛会自动脱落并形成胞囊,随后细胞壁增厚,同时在鞭毛部位的反向一极(多数情况下即与饲料碎片接触部位)形成芽管,芽管进一步萌发生长成为新的菌体[14]。此处涉及3个概念,其一为丝/球状假根型厌氧真菌,分别代表形成纤维状分枝型假根和球茎状假根系统的厌氧真菌。其二为单/多中心厌氧真菌,它是根据菌体产生的游动孢子囊的数量来定义的,单中心厌氧真菌仅产生1个/簇孢子囊,而多中心厌氧真菌则可以产生数个/簇孢子囊。其三为内/外源性孢子囊发育类型,内源性孢子囊发育是指游动孢子中携带的遗传物质保留在胞囊中,随着菌体的生长逐渐发育成新的孢子囊;而外源性孢子囊发育是指游动孢子中携带的遗传物质先转移至营养菌体中,再于其他部位发育形成孢子囊。这3类特征也是区分属水平厌氧真菌系统发育分类的重要依据。
大多数单中心厌氧真菌均通过内源性孢子囊发育模式进行增殖,即胞囊萌发出的芽管发育成无核假根,而具核胞囊则发育成新的游动孢子囊。然而,有些单中心厌氧真菌如瘤胃壶菌属菌种(Piromyces spp.),可通过外源性孢子囊发育模式进行增殖,其假根发育方式和内源性单中心厌氧真菌相同,但在假根发育完成之后,原胞囊相对于假根的另一侧会产生1个孢囊柄,而原胞囊中的细胞核会转移至孢囊柄中发育成新的孢子囊[15]。上述2种发育类型都是严格定向的。另外还有一些单中心厌氧真菌如山羊真菌属(Capellomyces),则同时存在内/外源性2种孢子囊发育类型[7, 16-19]。与此相反,多中心厌氧真菌则进行外源性孢子囊发育,表现在原胞囊进行单侧萌发,且在萌发的过程中,包括细胞核在内的胞囊内容物会迁移到芽管中,发育成具核假根系统,并能够产生多个孢子囊;而剩余的胞囊仅保留一个空壳而不具有特殊功能。由此可见,多中心厌氧真菌的菌体发育是非定向的。具有球状假根或锚定器的厌氧真菌常表现为外源性发育类型,细胞核在营养菌体(假根/锚定器和孢囊柄)及生殖体(孢子囊)中均可发现。此类厌氧真菌可产生单中心或有限的多中心菌体,原胞囊可形成具有(如盲肠真菌属,Caecomyces)[20-21]或不具有(如肠道真菌属,Cyllamyces)[22]假根的球状锚定器。球状锚定器产生管状或分枝型的孢囊柄,孢囊柄顶端产生游动孢子囊。此类真菌的生长不像多中心菌体那样具有不定向性,但也不像单中心丝状假根型厌氧真菌那样严格定向[22]。
厌氧真菌的假根在生长的过程中可以物理性的穿透植物性饲料原料,并通过多种纤维降解酶和膨胀蛋白对植物组织进行分解消化,进而为其本身和孢子囊的生长发育提供所需的营养物质。待孢子囊发育成熟后,若存在合适的诱导物,便可由顶囊部或整体产生裂解,释放出发育分化后的游动孢子,完成1个生命周期(图 1)。然而,除了正常的生命周期循环之外,研究者普遍认为在孢子囊成熟和释放之间还存在一个特殊的生命阶段——休眠期。处于休眠期的厌氧真菌可以通过形成某种耐氧结构而对有氧环境产生一定的耐受性,从而确保游动孢子能够在有氧环境下存活,并在合适的环境条件下再次激活并发育。迄今为止已报道的休眠结构包括静息孢子囊[23]、黑化抗性孢子囊[24]和多室芽胞状结构[25]等,但目前对其形成及后期再次萌发过程知之甚少,且不同种类厌氧真菌的休眠结构可能并不相同[6]。尽管如此,休眠期及其特殊结构的存在对厌氧真菌的宿主个体间传递起着至关重要的作用,同时也有力地解释了可以从干燥的、长期暴露于氧气条件下的粪便样品中分离获得厌氧真菌的原因[19, 26]。
如前文所述,我们目前对厌氧真菌生命周期的理解多来自于瘤胃厌氧真菌的相关研究,而假反刍动物前肠道来源的厌氧真菌由于所栖息的生境与瘤胃相似,因此很可能具有类似的生命周期[6]。然而,对于(假)反刍动物后肠道以及后肠发酵型草食动物来源的厌氧真菌,由于目前尚不清楚瘤胃中存在的诱导性物质(如血红素等)能否通过前消化道到达后肠道,也不清楚后肠道中是否存在充足的趋化信号(如水溶性糖和酚酸)可供厌氧真菌利用,因此这些来源的厌氧真菌的生命周期特性仍有待进一步研究。
2 厌氧真菌的酶学特性上文提到,假根在生长发育过程中可通过其产生的多种纤维降解酶和膨胀蛋白及物理侵袭性生长能力、基于穿透性的菌丝顶端来破坏纤维性物质。这些菌丝顶端部位具有非常高的纤维降解酶浓度/活性,而厌氧真菌这种生长特性也增加了其他纤维降解微生物获得营养物质的途径和可能性[3, 27-28],因此通常被认为是植物性饲料原料降解的主要发起者。厌氧真菌降解植物纤维性物质的能力与其酶学特性密不可分,报道显示厌氧真菌具有庞大且多样的碳水化合物活性酶(carbohydrate-active enzymes,CAZymes)库,包括糖苷水解酶(glycoside hydrolases,GHs)、碳水化合物酯酶(carbohydrate esterases,CEs)、多糖裂解酶(polysaccharide lyases,PLs)、糖基转移酶(glycosyl transferases,GTs)和碳水化合物结合模块(carbohydrate-binding modules,CBMs)等[6]。此外,转录组研究结果表明,编码CAZymes的转录本的数量占总转录本数量的8%~12%[29-30]。
GHs是一类水解配糖体之间糖苷键的酶,在纤维素和半纤维素的水解中发挥重要作用,也是厌氧真菌中种类最为丰富的CAZymes。最近的基因组和转录组研究表明,约有43个GHs家族可由厌氧真菌产生,其中最为丰富的9个家族为GH3、GH5、GH6、GH10、GH11、GH43、GH45、GH48、GH114[6, 29, 31]。CEs催化糖类取代物的脱氧或脱氮酰化反应,主要起促进木质化植物细胞壁降解和消化的作用[32]。相关研究表明,厌氧真菌至少能够表达10个家族的CEs,其中普遍丰富表达4个CEs家族为CE1、CE4、CE6和CE16[29, 31],CE8和CE10在某些菌株中的表达量也很高[31]。PLs是一类非水解性裂解糖苷键的酶,它通过β消除机制裂解含糖醛酸的多糖链,同时生成不饱和的己糖醛酸残基和新的还原末端,可能对果胶具有降解特异性[29, 32]。厌氧真菌可产生至少6个家族的PLs[29, 31],其中PL1、PL3和PL4在目前已研究的厌氧真菌中普遍存在[29]。GTs可催化糖基从活化的供体分子转移到特定的受体分子而形成糖苷键,在糖类的合成中起重要作用[32]。厌氧真菌中含量最为丰富的GTs家族包括GT2、GT34、GT17、GT34、GT39和GT71[29],这些酶可能涉及细胞壁的生物合成、几丁质合成和糖基化反应等代谢过程[32]。CBMs是CAZymes内具有碳水化合物结合活性的离散折叠的连续氨基酸序列,它可以促进酶与底物的结合[32]。迄今在厌氧真菌中检测到约19个CBMs家族,其中CBM1、CBM10、CBM13和CBM18在已研究的菌株中普遍存在[29]。
厌氧真菌通常通过以下3种机制来降解纤维性物质:1)分泌多种游离CAZymes;2)分泌游离态的纤维复合酶体(cellulosomes,也称纤维小体);3)产生细胞结合态的纤维复合酶体(图 2)[30]。先前的研究表明,厌氧真菌能够分泌前文所述的多种功能相似、但机制和结构均具有显著差异的游离CAZymes,从多个方向攻击植物细胞壁聚合物,进而完成纤维素和半纤维素的糖化过程[3, 31]。除了能产生大量且功能多样的游离CAZymes,厌氧真菌非常重要的一个酶学特性是其能够产生纤维复合酶体。纤维复合酶体最初发现于厌氧细菌中,它能将多种纤维降解酶和相关的辅酶连接在一起,形成一种大分子多蛋白复合体[33-34]。纤维复合酶体一般由数种半纤维素酶或纤维素酶组成,这些酶通常均包含1个催化活性位点、1个或数个碳水化合物结合模块、1个黏连蛋白以及1个促进多酶复合体中各模块之间对接的锚定蛋白;而对于细胞结合态的纤维复合酶体,这些酶解模块多固定排布在位于细胞膜中的支架蛋白上(图 2)[33, 35]。通过这种模块化的结合,纤维复合酶体可利用各种单体酶之间的协同催化作用,显著提高其生物质降解效率,已有研究表明纤维复合酶体的降解效率约为游离酶的12倍[36]。细胞结合态的纤维复合酶体主要位于假根菌丝顶端,其强大的纤维降解能力可以增加菌丝的穿透性,促进纤维性结构的裂解和破坏过程。上述多种纤维降解机制的共存,在很大程度上可能是厌氧真菌能够高效降解未经处理的植物性生物质的关键所在,也是它们能在草食动物胃肠道等复杂的微生态环境中竞争性的获得营养物质的重要原因[6]。值得一提的是,不同属的厌氧真菌中游离CAZymes和纤维复合酶体的表达活性可能不尽相同,例如最近的转录组研究表明,丝状体假根型厌氧真菌产生的CAZymes转录本中约有30%包含非催化结构域(即可参与构成纤维复合酶体),远高于球茎状假根型厌氧真菌(约15%)[30]。
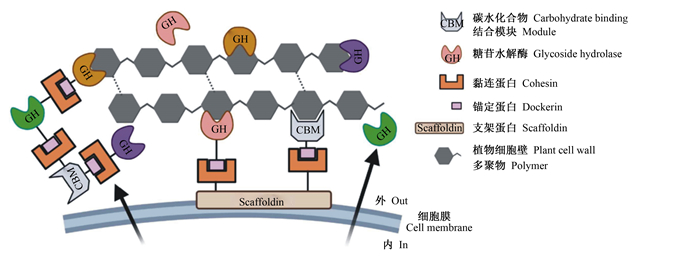
|
图 2 厌氧真菌利用多种酶学策略降解植物纤维性物质 Fig. 2 Anaerobic fungi deploy various zymologic strategies for degradation of plant biomass[6] |
尽管厌氧真菌和厌氧细菌中均存在纤维复合酶体,且一些学者认为厌氧真菌中的许多CAZymes基因是通过水平基因转移(horizontal gene transfer,HGT)从厌氧细菌中获得的[37-39],但是厌氧真菌来源的纤维复合酶体也有区别于厌氧细菌来源的纤维复合酶体的独特特性。首先,厌氧真菌中既存在固定于真菌细胞壁上的结合态纤维复合酶体,也存在分泌至胞外的游离态纤维复合酶体[27],而厌氧细菌来源的纤维复合酶体只能通过锚定域-支架蛋白模块固定于细菌细胞壁上[33]。其次,厌氧真菌来源的纤维复合酶体的组装过程是由真菌锚定蛋白介导的,其特点是可以利用不同菌体来源的单酶模块合成纤维复合酶体[40-41],而厌氧细菌来源的纤维复合酶体具有高度的物种特异性[42]。再次,厌氧真菌来源的纤维复合酶体存在一些厌氧细菌中所不具有的β葡萄糖苷酶活性(GH3、GH6和GH45),使前者可以直接将纤维素转化为可发酵的单糖,而后者如梭菌纤维复合酶体仅能产生低分子质量的寡糖[35]。值得一提的是,最近以基因组为中心的宏蛋白组和宏转录组学研究表明,在瘤胃微生态环境中,厌氧真菌和厌氧细菌产生的CAZymes之间具有互补作用,厌氧细菌产生的CAZymes主要用于分解半纤维素等较易降解的碳水化合物,而厌氧真菌来源的CAZymes(GH5、GH6和GH48)则更特异性的靶向难降解的结构性纤维素[43]。
基于上述厌氧真菌来源的CAZymes的特性和优势,加之很难构建大规模的厌氧真菌连续培养体系,近些年相关研究多聚焦于其相关生物技术开发上,例如通过基因重组体系工业化合成生产纤维复合酶体等[3, 44-45]。然而,尽管近些年取得了相当大的进步,但目前我们对厌氧真菌来源的纤维复合酶体仍然知之甚少[35]。此外,相对于厌氧细菌,目前学界对厌氧真菌及其CAZymes在促进草食动物胃肠道植物生物质降解方面的了解仍相当有限,这在一定程度上可归因于后者的基因组资源较前者要少的多,目前仅公开发表了4个属共7株厌氧真菌的基因组信息[35, 46-48]。随着学界对厌氧真菌在植物生物质降解过程中重要性的愈加了解和重视,以及更多基因组、转录组、蛋白质组、代谢组及相应的宏组学数据的生成和多组学联合分析,我们对纯培养和各种微生态系统背景下厌氧真菌及其分泌酶的理解也会日益深入。
3 厌氧真菌的代谢特性厌氧真菌在利用自身产生的各种酶将生物质降解为可利用的营养物单体后,单糖等物质还需要进一步发酵以为菌体生长发育提供必需的能量和各种营养素。酶解产生的单糖进入菌体内后,首先通过类似糖酵解途径进行糖酵解生成丙酮酸等发酵产物,随后进行与肠道厌氧细菌类似的混合酸发酵。在无菌培养条件下,所有种类的厌氧真菌混合酸发酵的终产物主要包括甲酸盐、乙醇、乙酸盐、乳酸盐和氢气,并通常产生少量的琥珀酸盐[44, 48],但这些代谢终产物的相对丰度可能因底物或菌种及其所处生长阶段的不同而有所差异。最近基于核磁共振的代谢组学研究表明,α-酮戊二酸也是厌氧真菌的主要发酵产物[49]。
与肠道厌氧细菌不同的是,厌氧真菌的混合酸发酵可同时在细胞质和特殊的细胞器——氢化酶体(hydrogenosomes)中进行。丙酮酸在2处发酵位点的化学计量比目前尚没有一致的结论,Boxma等[50]认为在纯培养条件下丙酮酸在细胞质和氢化酶体中降解的比例分别为35%和65%;而Wilken等[48]的研究结果却恰恰相反,其原因可能是二者使用的菌种[E2瘤胃壶菌(Piromyces sp. E2)vs.拉纳蒂新毛孢菌(Neocallimastix lanati)]和所使用的碳源及其浓度(0.3%果糖vs.0.5%葡萄糖)有所不同。细胞质中的丙酮酸有以下2种发酵途径:其一是在乳酸脱氢酶的作用下生成乳酸,其二是在丙酮酸甲酸裂解酶(PFL)的催化下生成甲酸盐和乙酰辅酶A(Ace-CoA),Ace-CoA随后在乙醇脱氢酶E的作用下生成乙醇[46, 50]。
氢化酶体最初发现于多种原生动物(如鞭毛原虫)中,其功能与线粒体相似,但通过底物水平的磷酸化分解丙酮酸等糖酵解产物,并产生氢气和ATP等[51-52]。然而,厌氧真菌来源的氢化酶体具有不同的代谢机制。最近基于基因组重建的研究表明,细胞质中的丙酮酸进入氢化酶体后,可通过2种途径进行代谢:其一是在PFL的催化下生成甲酸盐和Ace-CoA;其二则与原生动物中相似,是在丙酮酸铁氧化还原蛋白氧化酶(PFO)的作用下生成二氧化碳和Ace-CoA,同时耦合铁氧化还原蛋白氢化酶生成氢气;二者生成的Ace-CoA在乙酰琥珀酰辅酶A转移酶和琥珀酰辅酶A合成酶的作用下生成乙酸和ATP(图 3)[46, 48]。需要注意的是,代谢终产物浓度验证试验表明,在纯培养条件下,厌氧真菌氢化酶体中PFL通路占据优势地位,因此产生氢气的量较少,该代谢过程可能受到菌体自身的调控[48, 50]。另外,细胞质中产生的苹果酸盐也可转移至氢化酶体中,随后在苹果酸酶的作用下生成二氧化碳、还原型烟酰胺腺嘌呤二核苷酸(磷酸)[NAD(P)H]和丙酮酸,丙酮酸再通过上述途径进行代谢(图 3)。
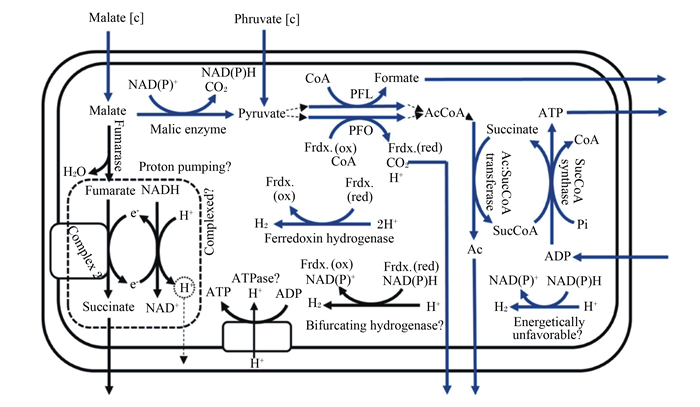
|
蓝色箭头表示已验证的核心酶解通路,黑色箭头表示根据基因组推定的酶解通路。 PFL:丙酮酸甲酸裂解酶pyruvate formate lyase;PFO:丙酮酸铁氧化还原蛋白氧化还原酶pyruvate ferredoxin oxidoreductase;Ac:乙酸acetate;SucCoA:琥珀酰辅酶A succinyl coenzyme A;CoA:辅酶A coenzyme A;AcCoA:乙酰辅酶A acetyl coenzyme A;Frdx.(ox/red):铁氧化还原蛋白(氧化态/还原态) iron redox protein (oxidized/reduced);Pi:磷酸基团phosphate group;Ac: SucCoA transferase:乙酰: 琥珀酰辅酶A转移酶acetyl: succinyl coenzyme A transferase;SucCoA synthase:琥珀酰辅酶A合成酶succinyl coenzyme A transferase;Malate:苹果酸盐;Malic enzyme:苹果酸酶;Pyruvate:丙酮酸盐;Fumarase:延胡索酸酶;Fumarase:延胡索酸;Succinate:琥珀酸盐;Formate:甲酸盐;Ferredoxin hydrogenase铁氧还蛋白氢化酶;Bifurcating hydrogenase:分支氢化酶;Complex:功能复合体;Energetically unfavorable:能量负平衡;ADP/ATP:二/三磷酸腺苷;ATPase:三磷酸腺苷酶;NAD(P)H:还原型烟酰胺腺嘌呤二核苷酸(磷酸) reduced nicotinamide adenine dinucleotide (phosphate);NAD(P)+:烟酰胺腺嘌呤二核苷酸(磷酸) Nicotinamide adenine dinucleotide (phosphate);Proton pumping:质子泵;H+:氢离子hydrogen ion;e-:电子electron;H2:氢气hydrogen;CO2:二氧化碳carbon dioxide。 The blue arrow indicated the verified core enzymatic pathways, and the black arrow indicated the speculative enzymatic pathways according genome. 图 3 厌氧真菌氢化酶体代谢示意图 Fig. 3 Schematic diagram of hydrogenosome in anaerobic fungi[48] |
综上可知,PFL通路在细胞质和氢化酶体中均为丙酮酸裂解的主要途径,因此理论上终产物中甲酸盐与乙醇和乙酸的化学计量比应为1 ∶ 1,这也得到了试验验证[48, 50]。此外,先前的报道认为,琥珀酸盐和α-酮戊二酸盐等代谢终产物均通过不完整的三羧酸循环(TCA循环)过程在细胞质中生成[44, 49]。然而,考虑到真核生物的TCA循环主要在线粒体中进行,而氢化酶体作为线粒体的变体应该具有相似的功能,且目前的研究结果并不能很好地说明氢化酶体中产生的NAD(P)H和烟酰胺腺嘌呤二核苷酸(磷酸)[NAD(P)+]之间的循环再生过程;而最近的基因组重构研究表明,氢化酶体中存在许多与NAD(P)H再循环、琥珀酸等脂肪酸合成相关酶以及未知功能蛋白[46, 48],因此不能排除这些代谢终产物(如琥珀酸盐)在氢化酶体中合成的可能性。总之,目前我们对厌氧真菌氢化酶体及代谢机制的理解还十分有限,需要进一步的多组学(如蛋白质组、代谢组等)和理化试验(如酶纯化定性等)来对其进行研究。
对于合成代谢方面,最近基于拉纳蒂新毛孢菌(N. lanati)纯培养株的研究表明,该菌株具有所有蛋白质源性氨基酸和典型脂肪酸(软/硬脂酸、豆蔻酸)的完整合成途径,除叶酸、亚铁血红素和生物素之外的大部分典型维生素和辅酶因子生物合成途径,以及完整的核酸合成途径,并通过控制添加试验得以间接证实[48]。此外,报道显示,除了初级代谢产物外,许多真菌还能产生次级代谢产物,在细胞中起到防御、信号传导和应激反应等作用[53]。次级代谢产物是一类重要的分子研究对象,目前从其他类型的真菌中分离到的次级代谢产物已有用作抗菌化合物、抗肿瘤剂和杀虫剂的先例[44]。但由于它们通常产量较低且往往是在某些特定条件下产生的,因此往往难以描述,例如上文厌氧真菌生命周期部分描述的厌氧真菌抗性结构及其形成信号机制等。据我们所知,迄今尚未有厌氧真菌次级代谢产物的相关报道,但在许多厌氧真菌基因组中均发现了聚酮合成酶(polyketide synthases)和非核糖体肽合成酶(nonribosomal peptide synthetases)等经典的次级代谢产物生物合成酶基因[35],表明厌氧真菌具有合成次级代谢产物的潜力,且有报道称厌氧真菌次级代谢产物可能是新型抗生素、治疗药剂和替代生物燃料的重要来源[54]。
此外,上述研究结果均是在纯培养条件下获得的,而在天然环境如草食动物肠道中,厌氧真菌往往会同其他类型微生物产生交互作用,导致典型代谢终产物无法积累或代谢途径发生变化。例如,Cheng等[49]研究表明,当厌氧真菌与产甲烷菌共培养时,主要发酵终产物是柠檬酸盐。因此,在研究复杂的自然微生态环境下厌氧真菌的代谢特性和功能时,需要考虑不同类型微生物间的交互作用。
4 小结与展望厌氧真菌在生长发育过程中,会在动态的游动孢子至静态的营养菌体之间周期性的转变,这种独特的变态发育特性使之被发现半个多世纪后才得以准确定性,同时也致使准确测定其在特定环境中的数量和生物量变得异常困难。目前对厌氧真菌生命周期的理解多来自于瘤胃厌氧真菌,其生命周期中可能存在的休眠阶段以及后肠发酵型厌氧真菌的生命周期特性尚有待进一步研究。厌氧真菌优异的植物纤维降解性能与其具有庞大且功能多样的碳水化合物活性酶分泌能力密不可分,而能够形成特殊的真菌纤维复合酶体是其高效降解纤维性物质的关键。厌氧真菌主要通过混合酸发酵来获得自身生长所需的能量和各种营养物质,并产生甲酸等一系列代谢终产物,终产物的相对丰度随菌种和所处生命阶段及发酵底物的不同而变化。厌氧真菌中发酵过程主要在细胞质和氢化酶体中进行,详细发酵机制尚需进一步研究和完善。总之,尽管最近的研究已大大增加了我们对厌氧真菌的生物学特性的认知,但还远远达不到全面了解的程度,而组学等新型分子生物学技术的普遍应用有望使我们对其生物学特性获得更深层次的理解。此外,近年来对厌氧真菌的相关研究多集中于生物技术开发方面,但在其寄居的复杂的草食动物肠道微生态环境中,厌氧真菌的生态作用及其对宿主的功能和健康的贡献程度,仍有待进一步研究和阐明。
| [1] |
GRUNINGER R J, PUNIYA A K, CALLAGHAN T M, et al. Anaerobic fungi (phylum Neocallimastigomycota): advances in understanding their taxonomy, life cycle, ecology, role and biotechnological potential[J]. FEMS Microbiology Ecology, 2014, 90(1): 1-17. DOI:10.1111/1574-6941.12383 |
| [2] |
THEODOROU M K, ZHU W Y, RICKERS A, et al. Biochemistry and ecology of anaerobic fungi[M]//HOWARD D H, MILLER J D. Human and animal relationships. Berlin, Heidelberg: Springer, 1996: 265-295.
|
| [3] |
SOLOMON K V, HAITJEMA C H, HENSKE J K, et al. Early-branching gut fungi possess a large, comprehensive array of biomass-degrading enzymes[J]. Science, 2016, 351(6278): 1192-1195. DOI:10.1126/science.aad1431 |
| [4] |
SIJTSMA L, TAN B. Degradation of perennial ryegrass leaf and stem cell walls by the anaerobic fungus Neocallimastix sp. strain CS3b[J]. Applied and Environmental Microbiology, 1996, 62(4): 1437-1440. DOI:10.1128/aem.62.4.1437-1440.1996 |
| [5] |
ORPIN C G. The rumen flagellate Piromonas communis: its life-history and invasion of plant material in the rumen[J]. Journal of General Microbiology, 1977, 99(1): 107-117. DOI:10.1099/00221287-99-1-107 |
| [6] |
HESS M, PAUL S S, PUNIYA A K, et al. Anaerobic fungi: past, present, and future[J]. Frontiers in Microbiology, 2020, 11: 584893. DOI:10.3389/fmicb.2020.584893 |
| [7] |
STABEL M, HANAFY R A, SCHWEITZER T, et al. Aestipascuomyces dupliciliberans gen. nov, sp. nov., the first cultured representative of the uncultured SK4 clade from aoudad sheep and alpaca[J]. Microorganisms, 2020, 8(11): 1734. DOI:10.3390/microorganisms8111734 |
| [8] |
ORPIN C G, GREENWOOD Y. The role of haems and related compounds in the nutrition and zoosporogenesis of the rumen chytridiomycete Neocallimastx frontalis H8[J]. Journal of General Microbiology, 1986, 132(8): 2179-2185. |
| [9] |
ORPIN C G, BOUNTIFF L. Zoospore chemotaxis in the rumen phycomycete Neocallimastix frontalis[J]. Journal of General Microbiology, 1978, 104(1): 113-122. DOI:10.1099/00221287-104-1-113 |
| [10] |
WUBAH D A, KIM D S. Chemoattraction of anaerobic ruminal fungi zoospores to selected phenolic acids[J]. Microbiological Research, 1996, 151(3): 257-262. DOI:10.1016/S0944-5013(96)80022-X |
| [11] |
ORPIN C G. On the induction of zoosporogenesis in the rumen phycomycetes Neocallimastix frontalis, Piromonas communis and Sphaeromonas communis[J]. Journal of General Microbiology, 1977, 101(2): 181-189. DOI:10.1099/00221287-101-2-181 |
| [12] |
LOWE S E, GRIFFITH G G, MILNE A, et al. The life cycle and growth kinetics of an anaerobic rumen fungus[J]. Journal of General Microbiology, 1987, 133(7): 1815-1827. |
| [13] |
EDWARDS J E, KINGSTON-SMITH A H, JIMENEZ H R, et al. Dynamics of initial colonization of nonconserved perennial ryegrass by anaerobic fungi in the bovine rumen[J]. FEMS Microbiology Ecology, 2008, 66(3): 537-545. DOI:10.1111/j.1574-6941.2008.00563.x |
| [14] |
ORPIN C G. Studies on the rumen flagellate Neocallimastix frontalis[J]. Journal of General Microbiology, 1975, 91(2): 249-262. DOI:10.1099/00221287-91-2-249 |
| [15] |
BARR D J S, KUDO H, JAKOBER K D, et al. Morphology and development of rumen fungi: Neocallimastix sp., Piromyces communis, and Orpinomyces bovis gen. nov., sp. nov.[J]. Canadian Journal of Botany, 1989, 67(9): 2815-2824. DOI:10.1139/b89-361 |
| [16] |
HANAFY R A, ELSHAHED M S, LIGGENSTOFFER A S, et al. Pecoramyces ruminantium, gen. nov., sp. nov., an anaerobic gut fungus from the feces of cattle and sheep[J]. Mycologia, 2017, 109(2): 231-243. DOI:10.1080/00275514.2017.1317190 |
| [17] |
JOSHI A, LANJEKAR V B, DHAKEPHALKAR P K, et al. Liebetanzomycespolymorphus gen. et sp. nov., a new anaerobic fungus (Neocallimastigomycota) isolated from the rumen of a goat[J]. MycoKeys, 2018, 40: 89-110. DOI:10.3897/mycokeys.40.28337 |
| [18] |
HANAFY R A, ELSHAHED M S, YOUSSEF N H. Feramyces austinii, gen. nov., sp. nov., an anaerobic gut fungus from rumen and fecal samples of wild Barbary sheep and fallow deer[J]. Mycologia, 2018, 110(3): 513-525. DOI:10.1080/00275514.2018.1466610 |
| [19] |
HANAFY R A, LANJEKAR V B, DHAKEPHALKAR P K, et al. Seven new Neocallimastigomycota genera from wild, zoo-housed, and domesticated herbivores greatly expand the taxonomic diversity of the phylum[J]. Mycologia, 2020, 112(6): 1212-1239. DOI:10.1080/00275514.2019.1696619 |
| [20] |
GOLD J J, HEATH I B, BAUCHOP T. Ultrastructural description of a new chytrid genus of caecum anaerobe, Caecomyces equi gen. nov., sp. nov., assigned to the Neocallimasticaceae[J]. BioSystems, 1988, 21(3/4): 403-415. |
| [21] |
CHEN Y C, TSAI S D, CHENG H L, et al. Caecomyces sympodialis sp. nov., a new rumen fungus isolated from Bos indicus[J]. Mycologia, 2007, 99(1): 125-130. DOI:10.1080/15572536.2007.11832607 |
| [22] |
OZKOSE E, THOMAS B J, DAVIES D R, et al. Cyllamyces aberensis gen. nov. sp. nov., a new anaerobic gut fungus with branched sporangiophores isolated from cattle[J]. Canadian Journal of Botany, 2001, 79(6): 666-673. DOI:10.1139/b01-047 |
| [23] |
ORPIN C G. Isolation of cellulolytic phycomycete fungi from the caecum of the horse[J]. Journal of General Microbiology, 1981, 123(2): 287-296. |
| [24] |
WUBAH D A, FULLER M S, AKIN D E. Resistant body formation in Neocallimastix sp., an anaerobic fungus from the rumen of a cow[J]. Mycologia, 1991, 83(1): 40-47. DOI:10.2307/3759831 |
| [25] |
BROOKMAN J L, MENNIM G, TRINCI A P J, et al. Identification and characterization of anaerobic gut fungi using molecular methodologies based on ribosomal ITS1 and 18S rRNA[J]. Microbiology, 2000, 146(2): 393-403. DOI:10.1099/00221287-146-2-393 |
| [26] |
GRIFFITH G W, OZKOSE E, THEODOROU M K, et al. Diversity of anaerobic fungal populations in cattle revealed by selective enrichment culture using different carbon sources[J]. Fungal Ecology, 2009, 2(2): 87-97. DOI:10.1016/j.funeco.2009.01.005 |
| [27] |
HAITJEMA C H, SOLOMON K V, HENSKE J K, et al. Anaerobic gut fungi: advances in isolation, culture, and cellulolytic enzyme discovery for biofuel production[J]. Biotechnology and Bioengineering, 2014, 111(8): 1471-1482. DOI:10.1002/bit.25264 |
| [28] |
DAGAR S S, KUMAR S, GRIFFITH G W, et al. A new anaerobic fungus (Oontomyces anksri gen. nov., sp. nov.) from the digestive tract of the Indian camel (Camelus dromedarius)[J]. Fungal Biology, 2015, 119(8): 731-737. DOI:10.1016/j.funbio.2015.04.005 |
| [29] |
GRUNINGER R J, NGUYEN T T M, REID I D, et al. Application of transcriptomics to compare the carbohydrate active enzymes that are expressed by diverse genera of anaerobic fungi to degrade plant cell wall carbohydrates[J]. Frontiers in Microbiology, 2018, 9: 1581. DOI:10.3389/fmicb.2018.01581 |
| [30] |
HENSKE J K, GILMORE S P, KNOP D, et al. Transcriptomic characterization of Caecomyces churrovis: a novel, non-rhizoid-forming lignocellulolytic anaerobic fungus[J]. Biotechnology for Biofuels, 2017, 10: 305. DOI:10.1186/s13068-017-0997-4 |
| [31] |
COUGER M B, YOUSSEF N H, STRUCHTEMEYER C G, et al. Transcriptomic analysis of lignocellulosic biomass degradation by the anaerobic fungal isolate Orpinomyces sp. strain C1A[J]. Biotechnology for Biofuels, 2015, 8: 208. DOI:10.1186/s13068-015-0390-0 |
| [32] |
LOMBARD V, GOLACONDA RAMULU H, DRULA E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2013, 42(D1): D490-D495. |
| [33] |
FONTES C M G A, GILBERT H J. Cellulosomes: highly efficient nanomachines designed to deconstruct plant cell wall complex carbohydrates[J]. Annual Review of Biochemistry, 2010, 79: 655-681. DOI:10.1146/annurev-biochem-091208-085603 |
| [34] |
LAMED R, SETTER E, BAYER E A. Characterization of a cellulose-binding, cellulase-containing complex in Clostridium thermocellum[J]. Journal of Bacteriology, 1983, 156(2): 828-836. DOI:10.1128/jb.156.2.828-836.1983 |
| [35] |
HAITJEMA C H, GILMORE S P, HENSKE J K, et al. A parts list for fungal cellulosomes revealed by comparative genomics[J]. Nature Microbiology, 2017, 2: 17087. DOI:10.1038/nmicrobiol.2017.87 |
| [36] |
KRAUSS J, ZVERLOV V V, SCHWARZ W H. In vitro reconstitution of the complete Clostridium thermocellum cellulosome and synergistic activity on crystalline cellulose[J]. Applied and Environmental Microbiology, 2012, 78(12): 4301-4307. DOI:10.1128/AEM.07959-11 |
| [37] |
MURPHY C L, YOUSSEF N H, HANAFY R A, et al. Horizontal gene transfer as an indispensable driver for evolution of Neocallimastigomycota into a distinct gut-dwelling fungal lineage[J]. Applied and Environmental Microbiology, 2019, 85(15): e00988-19. DOI:10.1128/AEM.00988-19 |
| [38] |
GARCIA-VALLVÉ S, ROMEU A, PALAU J. Horizontal gene transfer of glycosyl hydrolases of the rumen fungi[J]. Molecular Biology and Evolution, 2000, 17(3): 352-361. DOI:10.1093/oxfordjournals.molbev.a026315 |
| [39] |
WANG Y, YOUSSEF N H, COUGER M B, et al. Molecular dating of the emergence of anaerobic rumen fungi and the impact of laterally acquired genes[J]. mSystems, 2019, 4(4): e00247-19. DOI:10.1128/mSystems.00247-19 |
| [40] |
FANUTTI C, PONYI T, BLACK G W, et al. The conserved noncatalytic 40-residue sequence in cellulases and hemicellulases from anaerobic fungi functions as a protein docking domain[J]. Journal of Biological Chemistry, 1995, 270(49): 29314-29322. DOI:10.1074/jbc.270.49.29314 |
| [41] |
NAGY T, TUNNICLIFFE R B, HIGGINS L D, et al. Characterization of a double dockerin from the cellulosome of the anaerobic fungus Piromyces equi[J]. Journal of Molecular Biology, 2007, 373(3): 612-622. DOI:10.1016/j.jmb.2007.08.007 |
| [42] |
BAYER E A, BELAICH J P, SHOHAM Y, et al. The cellulosomes: multienzyme machines for degradation of plant cell wall polysaccharides[J]. Annual Review of Microbiology, 2004, 58: 521-554. DOI:10.1146/annurev.micro.57.030502.091022 |
| [43] |
HAGEN L H, BROOKE C G, SHAW C A, et al. Proteome specialization of anaerobic fungi during ruminal degradation of recalcitrant plant fiber[J]. The ISME Journal, 2021, 15(2): 421-434. DOI:10.1038/s41396-020-00769-x |
| [44] |
EDWARDS J E, FORSTER R J, CALLAGHAN T M, et al. PCR and omics based techniques to study the diversity, ecology and biology of anaerobic fungi: insights, challenges and opportunities[J]. Frontiers in Microbiology, 2017, 8: 1657. DOI:10.3389/fmicb.2017.01657 |
| [45] |
GILMORE S P, LILLINGTON S P, HAITJEMA C H, et al. Designing chimeric enzymes inspired by fungal cellulosomes[J]. Synthetic and Systems Biotechnology, 2020, 5(1): 23-32. DOI:10.1016/j.synbio.2020.01.003 |
| [46] |
YOUSSEF N H, COUGER M B, STRUCHTEMEYER C G, et al. The genome of the anaerobic fungus Orpinomyces sp. strain C1A reveals the unique evolutionary history of a remarkable plant biomass degrader[J]. Applied and Environmental Microbiology, 2013, 79(15): 4620-4634. DOI:10.1128/AEM.00821-13 |
| [47] |
LI Y F, LI Y Q, JIN W, et al. Combined genomic, transcriptomic, proteomic, and physiological characterization of the growth of Pecoramyces sp. F1 in monoculture and co-culture with a syntrophic methanogen[J]. Frontiers in Microbiology, 2019, 10: 435. DOI:10.3389/fmicb.2019.00435 |
| [48] |
WILKEN S E, MONK J M, LEGGIERI P A, et al. Experimentally validated reconstruction and analysis of a genome-scale metabolic model of an anaerobic Neocallimastigomycota fungus[J]. mSystems, 2021, 6(1): e00002-21. DOI:10.1128/mSystems.00002-21 |
| [49] |
CHENG Y F, JIN W, MAO S Y, et al. Production of citrate by anaerobic fungi in the presence of co-culture methanogens as revealed by 1H NMR spectrometry[J]. Asian-Australasian Journal of Animal Sciences, 2013, 26(10): 1416-1423. |
| [50] |
BOXMA B, VONCKEN F, JANNINK S, et al. The anaerobic chytridiomycete fungus Piromyces sp. E2 produces ethanol via pyruvate: formate lyase and an alcohol dehydrogenase E[J]. Molecular Microbiology, 2004, 51(5): 1389-1399. |
| [51] |
HACKSTEIN J H P, BAKER S E, VAN HELLEMOND J J, et al. Hydrogenosomes of anaerobic chytrids: an alternative way to adapt to anaerobic environments[M]//TACHEZY J. Hydrogenosomes and mitosomes: mitochondria of anaerobic eukaryotes. Berlin, Heidelberg: Springer, 2008: 147-162.
|
| [52] |
HACKSTEIN J H P, TJADEN J, KOOPMAN W, et al. Hydrogenosomes (and related organelles, either) are not the same[M]//MARTIN W F, MVLLER M. Origin of Mitochondria and Hydrogenosomes. Berlin, Heidelberg: Springer, 2007: 135-159.
|
| [53] |
KELLER N P, TURNER G, BENNETT J W. Fungal secondary metabolism-from biochemistry to genomics[J]. Nature Reviews Microbiology, 2005, 3(12): 937-947. DOI:10.1038/nrmicro1286 |
| [54] |
PODOLSKY I A, SEPPÄLÄ S, LANKIEWICZ T S, et al. Harnessing nature's anaerobes for biotechnology and bioprocessing[J]. Annual Review of Chemical and Biomolecular Engineering, 2019, 10: 105-128. DOI:10.1146/annurev-chembioeng-060718-030340 |




