2. 聊城大学农学院, 山东黑毛驴高效繁育与生态工程 技术研究中心, 山东毛驴产业科技协同创新中心, 聊城 252059
2. Shandong Donkey Industry Technology Collaborative Innovation Center, Shandong Engineering Technology Research Center for Efficient Breeding and Ecological Feeding of Black Donkey, College of Agronomy, Liaocheng University, Liaocheng 252059, China
在电子显微镜下观察到成年的骨骼肌纤维外围附着圆形的单核细胞,这些细胞就是骨骼肌卫星细胞(skeletal muscle satellite cells,SCs),它们与肌纤维质膜有密切联系。骨骼肌再生是由骨骼肌卫星细胞增殖分化开始的,它是一群附着在肌肉组织中的成体干细胞,对骨骼肌的生长、内环境稳定和损伤后修复非常重要。在新生儿或者幼年阶段,肌纤维的数量保持不变,但是每个肌纤维可以通过骨骼肌卫星细胞的融合而变大,在正常情况下,成年的哺乳动物骨骼肌卫星细胞处于静止状态[1],受到刺激或诱导时被激活,进行增殖、分化并与原有的骨骼肌细胞融合,形成新的肌纤维[2-3]。大量的研究表明,肌肉再生过程中骨骼肌卫星细胞的增殖和分化受多种因素的影响,包括营养素、表观遗传调控及信号通路介导的生肌调控因子的影响[4-8]。研究发现,添加多酚化合物也可以使腺苷酸活化的蛋白激酶(AMP activated protein kinase,AMPK)信号通路激活受阻,导致泛素-蛋白酶解途径受阻,下调下游叉头框转录因子(Forkhead box O,Foxo)的磷酸化水平,进而抑制成肌细胞的分化和线粒体生物合成[9]。Irazoqui等[10]通过体外成肌细胞(C2C12)试验证明,补充维生素D3能够介导丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)信号通路参与调节细胞周期进程,促进成肌细胞的增殖。研究还发现氨基酸添加水平可以通过激活信号通路进而促进骨骼肌卫星细胞的分化[11-12]。以上研究均说明了营养素参与了骨骼肌卫星细胞的生命过程。骨骼肌卫星细胞必须经过多种细胞命运的转化才能达到完全分化状态,以完成肌纤维的修复。这些中间细胞的命运均共享同一个基因组,但是每种细胞的命运需要基因组中特异基因的表达决定,而表观遗传学在不改变DNA序列的基础上,在功能水平调控基因表达,增加基因调控的复杂性,进而实现不同细胞特征和功能的可遗传变异,主要包括组蛋白修饰、长非编码RNA、DNA甲基化以及转录因子的结合等[13-14]。组蛋白在调控骨骼肌再生和修复过程中经历了各种各样的翻译后修饰,包括乙酰化、泛素化、二磷酸腺苷(adenosine diphosphate,ADP)核糖化和甲基化,组蛋白修饰和转录之间的关系揭示了表观遗传是如何调控基因表达的。
每个细胞内的DNA都是独一无二的,大多数没有表达的基因均存在于细胞质中,而多数转录的基因聚集在细胞核内,细胞间这种独特的核组织结构是建立在特异基因表达的基础上。DNA与核小体的结合成就了染色质的流动状态,核小体移位允许未标记的组蛋白掺入其中,组蛋白修饰改变了核小体的化学性质和稳定性,这种更新机制对调节细胞命运和分化的过程至关重要。本文章概述了骨骼肌再生过程中肌卫星细胞命运转变的发生机理,并结合前人的研究对组蛋白修饰调控骨骼肌卫星细胞静息、激活以及增殖分化过程的最新研究内容进行综述,为进一步研究骨骼肌卫星细胞命运调控机制、促进哺乳动物骨骼肌发育及损伤修复提供参考依据。
1 骨骼肌卫星细胞调节骨骼肌的再生骨骼肌是哺乳动物机体的重要组成部分,约占体重的40%[15]。骨骼肌卫星细胞在正常情况下处于静息状态,当受到刺激后如受伤或患病诱导骨骼肌卫星细胞激活,其开始进行增殖分化并促进新纤维的形成,排列成肌肉组织[16],骨骼肌卫星细胞这种自我更新的状态说明其具有干细胞的特性,并且参与肌肉的发育和再生[17]。
Pax基因家族(Pax3和Pax7)是成肌过程中的2个上游转录因子,可以通过调节下游一系列的肌源调控因子(MRFs),如生肌决定因子(myogenic determination gene,Myod)、生肌调节因子5(myofactor 5,Myf5)、生肌调节因子4(myofactor 4,Myf4)和肌细胞生成素(myogenin,Myog)[5, 18]的表达决定肌卫星细胞命运[19]。Pax3主要在胚胎的肌肉发育过程中发挥作用,仅在膈肌等肌肉组织细胞中高表达,在其他肌肉中表达水平较低;而Pax7则在个体出生后的肌肉发育中起作用,在成年个体肌卫星细胞中高度表达[5]。Pax3和Pax7通过表观遗传机制调节Myf5和Myod的表达,使Myf5和Myod在肌细胞中快速上调以激活骨骼肌卫星细胞[20],由此肌卫星细胞开始分化为成肌细胞,融合为肌管,成熟为肌纤维。骨骼肌卫星细胞在静息状态下Pax7和Foxo高度表达[21],同时Myf5和Myod也被转录,但是转录后可抑制其翻译[5, 22]。肌肉受到损伤后,部分肌卫星细胞开始进行有丝分裂,细胞中Myf5和Myod开始表达,而Pax7表达开始减少,并且不再表达Foxo蛋白。随着肌卫星细胞的不断分裂,Pax7和Myf5表达受到抑制,而Myog诱导肌卫星细胞退出细胞周期,开始分化[5, 23]。最后随着多核肌纤维的形成,Myod表达逐渐下降,而Myf4和Myf6在肌纤维中开始高度表达[24-26]。试验发现肌肉在受损3 h之后就可以检测到Myod和Myf5的表达,说明细胞激活后Myod和Myf5能够快速有效地促进成肌细胞的状态转换[27-28]。由此可见,MRFs在调控骨骼肌卫星细胞命运的过程中有重要的作用,但是MRFs需要与广泛表达的E蛋白以及肌细胞增强家族的转录调控因子合作,从而诱导肌肉特异性基因表达[29]。
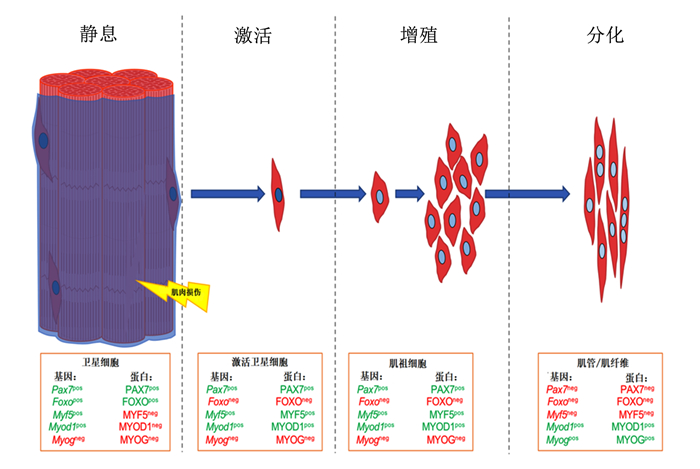
2 组蛋白修饰调控骨骼肌卫星细胞的静息和激活 2.1 骨骼肌卫星细胞的静息和激活状态静息状态是指细胞在细胞周期的G0期进入了可逆循环的骤停状态。通过分离培养未受损伤的骨骼肌卫星细胞研究染色质和表观遗传调控在维持细胞静息状态的作用,以确定细胞保留了静息状态的特征。Machado等[30]采用原位固定技术将分离前细胞锁定在静息状态,发现在肌肉再生过程中,MRFs的表达具有明显的顺序性。肌肉损伤后,肌卫星细胞被激活并退出静息状态,激活的肌卫星细胞经MRFs调节分化为成肌细胞,成肌细胞能够相互融合为成肌管或肌纤维,以恢复受损的肌肉(图 1)[31-32]。研究发现,在骨骼肌卫星细胞分离的短时间内观察到组蛋白H3上的表观遗传标记发生了广泛的变化,但是这段时间内没有发现DNA甲基化的差异[33-34]。基于这些发现,我们推测使用原位固定技术分析收集的表观遗传信息并不能完全代表细胞的静息状态,而是一种处于静息和激活之间的一种转变,即早期激活[35-36]。
|
Pax7:Pax基因家族7 paired box gene 7;Foxo:叉头框转录因子O亚家族forkhead box O;Myf5:生肌调节因子5 myofactor 5;Myod1:生肌决定因子1 myogenic determination gene 1;Myog:肌细胞生成素myogenin。 肌肉损伤后,肌肉卫星细胞被激活并退出静息状态,激活的卫星细胞迅速增殖为肌祖细胞,然后转化为分化的成肌细胞,成肌细胞能够相互融合成肌管并形成新的肌纤维,以恢复受损的肌肉。绿色:表达的基因或蛋白;红色:未表达的基因或蛋白[32]。 After a muscle injury, muscle satellite cells are activated and exit the quiescence state, the activated satellite cells transit to proliferative progenitor cells (myoblasts) which next transit into differentiated myocytes, myocytes are able to fuse to each other into myotubes, or to newly formed myofibers in order to restore the damaged muscle. Green: expressed genes or protein; red: unexpressed genes or proteins[32]. 图 1 肌肉再生过程中MRFs表达的层次性 Fig. 1 Hierarchy of MRFs expression during muscle regeneration process |
静息状态的维持需要抑制编码细胞周期蛋白和促进细胞周期退出的基因,在静息期的肌卫星细胞中,肿瘤抑制基因(P53)被证实了可以保持可逆的细胞周期停滞,而肿瘤抑制因子(ARF)可导致细胞周期停滞和衰老[35, 37]。为了维持这种平衡状态,不同途径促进了静息期骨骼肌卫星细胞的转录网络,特别是Foxo转录因子的表达,如Foxo1、Foxo3和Foxo4是维持卫星细胞静息所必需的[21]。Foxo3参与Notch通路的调控,使肌卫星细胞在自我更新过程中恢复静息状态[38],激活Notch信号通路会维持骨骼肌卫星细胞的静息和增殖状态,抑制其分化,故激活的Notch信号通路导致癌基因(MDM2)表达减少,P53表达的积累可以维持细胞周期的停滞[37, 39]。ARF则需要保持抑制状态,以防止肌肉干细胞衰老[37]。
到目前为止,骨骼肌卫星细胞维持静息状态的调节机制研究有限,体外细胞培养试验发现,多条信号通路参与了维持骨骼肌卫星细胞的静息状态[40]。细胞在静息状态时,染色质的高度聚合状态导致转录水平相对较低[41],但是许多基因仍然表达,包括DNA转录但不编码蛋白的MRFs,这说明了除了转录调控以外的其他机制也会调节细胞的命运,包括Myf5和Myod1 mRNA的核保留情况。换句话说,Myf5和Myod1的表达需要最小化,这种最小化必须通过表观遗传遗传机制调控。此外,有人推测,骨骼肌卫星细胞长时间的静息和遗传物质的保真度是由表观遗传学决定的,尽管这一假说还未有明确的证据[34, 42]。但事实上组蛋白H4第20位赖氨酸二甲基化标记(H4K20me2)是染色质高度紧密折叠和抑制Myod1表达所必需的[41, 43]。
2.2 组蛋白修饰调控骨骼肌卫星细胞的静息和激活Pax7和Foxo转录因子的表达是骨骼肌卫星细胞静息状态的关键特征,但是它们在静息状态的表达是如何调控的尚不清楚。组蛋白修饰通过改变残基上的修饰状态进而改变特异基因的表达水平,促使细胞发挥特异的功能。研究表明,维持骨骼肌卫星细胞静息状态的生肌调控因子Pax7主要受H3K4me3组蛋白修饰标记调控,在激活的卫星细胞中,MRFs如Myog明显抑制其组蛋白修饰[44],同时Pax7分别受到组蛋白Polycomb(PcG)和Trithorax(TrxG)的拮抗作用,分别抑制或激活其表达。研究还发现,在其启动子添加H3K4me3组蛋白修饰标记后,激活和增殖状态下的骨骼肌卫星细胞中未发现Pax7的表达,而静息状态下的卫星细胞中Pax7表达没有受到干扰[45]。细胞为了维持静息状态,需要表达特异性抑制因子阻止细胞周期的进入。如上所述,细胞周期抑制剂的选择是必不可少的,抑制因子的表达才能使细胞永久性的退出细胞周期。当肌肉受到刺激时,多梳基因家族(PcG)-多梳抑制复合物(PRC)2的催化亚基(Ezh2)可诱导转录因子启动子上的组蛋白修饰,通过抑制组蛋白H3第27位赖氨酸上三甲基化(H3K27me3)修饰,下调Pax7基因表达[41]。通过染色质免疫共沉淀测序(chromatin immunoprecipitation-sequencing,CHIP-Seq)结合转录组数据分析静息和激活状态下骨骼肌卫星细胞表观遗传图谱发现,MRFs如Myf5和Myod的转录启动子上存在大量的H3K27me3修饰标记,并且允许细胞激活。同时,除了MRFs之外,Ezh2癌基因调控组蛋白赖氨酸甲基转移酶在静息期的骨骼肌卫星细胞中表达,确保成年动物机体的肌卫星细胞库保持稳态状态[46]。但是特异性敲除Ezh2会导致骨骼肌卫星细胞增殖能力下降,肌肉受损后骨骼肌再生以及肌卫星细胞自我更新能力降低[18, 46]。
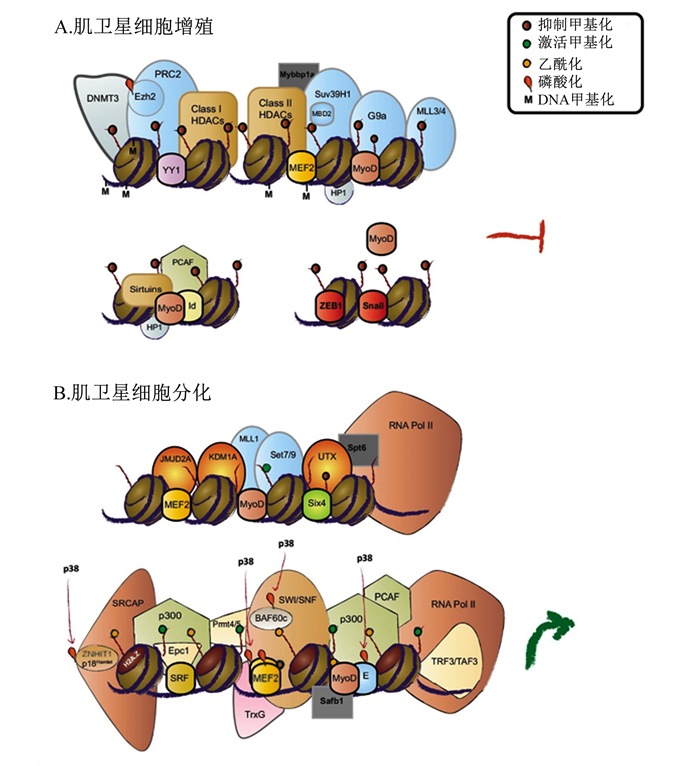
3 组蛋白修饰调控骨骼肌卫星细胞的增殖和分化 3.1 调节骨骼肌卫星细胞的增殖肌肉品质取决于肌肉纤维的特性,纤维数量、类型以及横截面积等形态特征是肌肉品质的主要决定因素[47]。在幼年期,肌纤维增大主要依赖于骨骼肌卫星细胞的增殖、分化和融合,细胞不断增殖受细胞周期蛋白和转录调控因子的共同作用。细胞周期蛋白需要维持在较高表达水平,且MRFs需要维持在较低水平,才能保证肌卫星细胞的增殖状态。Singh等[18]研究发现,Myod1的表达在骨骼肌卫星细胞中起重要作用,它是细胞激活和增殖所必需的,Myod1在细胞增殖过程中抑制分化基因,同时也可在分化期激活促分化功能的基因。Myod1在增殖和分化的过程中看起来是矛盾的,但是它确实既可以抑制特定基因,又可以激活特定基因[48]。在增殖期,Myod1、肌细胞增强因子2D(MEF2D)和转录中介因子1(KAP1)共同作用于细胞的分化,稳定2个共抑制酶(G9A、HDAC1)[49-50]和共激活酶(P300、LSD1)[51-52]之间的关联,这种增强型复合体组装在启动子上建立了一种稳定的染色质状态,当抑制酶占主导作用时即限制基因的表达。组蛋白甲基转移酶(HMT)在Myod1启动子及其远端调控区(DRR)增加了组蛋白H3第27位赖氨酸上二甲基化(H3K27me2)标记,其位点加入H3K27me2标记后可以诱导早期激活的卫星细胞形成异染色质并降低Myod1的表达[40]。这一机制也是在静息期抑制Myod1表达所必需的[42]。虽然调控H3K27me2标记的酶尚不清楚,但是维持这一标记的机制已经被揭开。另一项研究揭示了p38 MAPK信号转导通路受到刺激被激活后,在Myod1丝氨酸(Ser)199和200位点发生蛋白磷酸化,从而允许组蛋白H3K9甲基转移酶(SUV39h1/KMT1a)结合到Myog,减少其在增殖过程中的表达。此外,与组蛋白去乙酰化酶(HDAC)1/2相关的转录抑制因子锌指转录因子(SNAI1)可与分化相关基因(E-box)结合,从而阻止Myod1与靶基因表达的结合[53]。这一机制揭示了SNAI1/HDAC1/2复合体通过阻断Myod1启动分化而促进细胞增殖的作用。研究还发现Deltex2结合蛋白通过导致组蛋白脱甲基酶(JMJD1C)泛素化并抑制其去甲基化酶的活性以维持Myod1启动子和DRR上H3K27me2标记,进而抑制细胞分化[54]。
维持卫星细胞的增殖,仅抑制分化是不够的,还必须维持与细胞周期进程相关基因的表达。组蛋白脱乙酰化和乙酰化对肌源性分化有重要的作用,尤其是在Myod靶基因上,可促进分化进程[5, 55-56](图 2)。在原代小鼠成肌细胞中,E2F1/ p300/CBP相关因子(PCAF)复合物可以增强转录因子E2F1靶基因启动子上的PCAF占有率和组蛋白乙酰化标记,从而允许通过G1/S细胞周期检查点[57]。除了乙酰基转移酶的富集,在许多不同类型细胞研究中表明E2F1可以介导赖氨酸甲基转移酶2(KMT2)家族的组蛋白H3第4位赖氨酸(H3K4)甲基转移酶的富集[58]。在肌细胞系统中,增殖的C2C12细胞通过混合谱系白血病5蛋白(MLL5)在细胞周期基因A2上沉积H3K27me3标记,这是通过G1/S细胞周期检查点所必需的因子[59]。最后C2C12细胞中的组蛋白甲基转移酶Set2通过降低细胞周期蛋白水平而影响G1/S和G2/M期转变。这一结果说明了组蛋白甲基转移酶Set2在维持成肌细胞增殖方面具有重要功能[60]。同时Byrne等[61]发现在刚出生的绵羊骨骼肌基因组中H3K27me3与染色体构型和基因功能密切相关,属于抑制性染色质标记,大量存在于基因启动子中且密度不同,以抑制MRFs基因的表达。以上研究结果均表明组蛋白修饰有助于维持细胞周期进程,同时还能调控骨骼肌卫星细胞的增殖。
|
DNMT3:DNA甲基转移酶3 DNA methyltransferase 3;Class Ⅰ HDACs:Class Ⅰ选择性组蛋白去乙酰化酶class Ⅰ histone deacetylases;Class Ⅱ HDACs:Class Ⅱ选择性组蛋白去乙酰化酶class Ⅱ histone deacetylases;Mybbp1a:Myb结合蛋白1a Myb-binding protein 1a;MEF2:肌细胞增强因子2 myocyte enhancer factor 2;Suv39H1:组蛋白甲基化酶histone deacetylase;MBD2:甲基-CpG结合结构域蛋白2 methyl-CpG-binding domain protein 2;HP1:异染色质蛋白heterochromatin protein 1;G9a:常染色质组蛋白赖氨酸N-甲基转移酶2 euchanromatic histone-lysine N-methyltransferase 2;MLL1/3/4:MLL家族蛋白mixed lineage leukemia;Sirtuins:去乙酰化酶deacetylase;PCAF:P300/CBP相关因子P300/CBP associated factor;Id:分化抑制因子inhibitor of differentiation;ZEB1:E盒结合锌指蛋白1 zinc finger E-box-binding protein;Snail:锌指转录因子zinc-finger transcription factors;JMJD2A:含Jumonji结构域蛋白2A jumonji domain containing 2A;KDM1A:赖氨酸特异性组蛋白去甲基化酶1 lysine-specific histone demethylase;Set7/9:蛋白赖氨酸甲基化转移酶家族成员protein lysine methyltransferases;RNA Pol Ⅱ:RNA聚合酶Ⅱ RNA polymerase Ⅱ;MyoD:生肌决定因子myogenic determination gene;Six4:Six同源盒蛋白4 six homeobox 4;Spt6:转录延长因子suppressor of Ty6;SRCAP:染色质重塑酶snf2-related CBP activator protein;p38:p38丝裂原活化蛋白激酶p38 mitogen-activated protein kinase;p300:E1A相关p300蛋白E1A-associated p300 protein;Epc1:聚梳同源物增强子1 enhancer of polycomb homolog 1;SRF:血清反应因子serum response factor;Prmt4/5:蛋白质精氨酸甲基转移酶4/5 protein arginine methyltransferase 4/5; TrxG:trxG基因trithorax group gene;H2A.Z:组蛋白H2A变体histone H2A variant;Safb1:核基质结合因子scaffold attachment factor b1;TRF3/TAF3:TBP相关因子TBP-related factors。 图 2 组蛋白修饰调控骨骼肌卫星细胞增殖和分化的分子机制 Fig. 2 Molecular mechanism of histone regulation in SCs proliferation an differentiation[5] |
骨骼肌卫星细胞的分化起始于细胞退出细胞周期,由多种抑制细胞周期基因的机制与分化起始机制共同调控。Blais等[62-63]研究发现,骨骼肌卫星细胞分化时细胞抑制基因启动子上有大量H3K27me3富集,H3K27的三甲基化在抑制细胞周期基因表达方面起着重要作用,增殖期H3K27me3可抑制细胞周期蛋白依赖性激酶抑制因子(CDKN2B)基因表达,同时在分化期转录起始区域呈现抑制修饰减少的现象,进而促进细胞退出细胞周期进入分化状态。这些结果揭示了组蛋白修饰调控细胞分化的重要性。
细胞向分化转变的第一步是通过细胞周期阻滞进而阻止卫星细胞的增殖。在此前提下,Pax7的表达势必要下调。PcG-PRC2的催化亚基Ezh2受转录因子Yin-Yang1(YY1)的调节,上调Pax7的启动子区域的H3K27me3修饰,调控成肌分化过程[45]。在参与细胞分化的转录因子中,E2F是一个由8个蛋白组成的家族,包括视网膜母细胞瘤相关蛋白(RB)、视网膜母细胞瘤样蛋白1(RBL1)和视网膜母细胞瘤样蛋白2(RBL2),是细胞分裂和决定细胞命运的中心调控因子,它们的功能是控制蛋白质靶基因的表达,调节细胞周期,促进细胞分化[64-65]。RB可以与HDAC1结合维持细胞增殖,也可以与转录因子E2F4相互作用调节细胞退出细胞周期。同时RB还可以招募半胱氨酸甲基转移酶(HMT)在促进细胞周期相关蛋白靶基因启动子上增加H3K9me3和H3K27me3标记,减少特异基因表达,调节细胞周期的退出并开始分化。Hsu等[66]在小鼠胚胎干细胞中将E2F4特异性敲除后,发现E2F4可与RB家族成员相互作用作为一种与细胞周期停滞有关的转录抑制因子。这种抑制机制依赖于核心蛋白复合体(PRC1和PRC2),其中H3K9me3和H3K27me3的标记建立了一种沉默状态[67],这是由于致死因子恶性脑瘤样蛋白1(L3MBTL1)的催化亚基E2F4与异染色质蛋白(HP1γ)相互作用导致染色质聚合复合体募集到靶基因,进而维持这种沉默状态[68]。
Myod1和Myog的表达对卫星细胞分化也是必不可少的。Myod1表达的增加是由于JMJD1C蛋白驱动H3K9me3标记在其启动子上去甲基化的结果[49]。Myod1高水平表达后,SNAI1/HDAC1/2复合物和Myod1在靶基因E-box处发生转换以促进分化[48]。Myod1、MEF2D和Foxo3等转录因子在MRFs启动子上形成的转录活性状态依赖于多种表观遗传酶的联合作用。激活Myog基因的第1步就是通过赖氨酸去甲基化酶(JMJD2/KDM4A)在启动子上去除H3K9me2和H3K9me3抑制性标记[69]。一旦抑制标记被去除,启动子就可以被修饰并允许转录。第1个标记是启动子上H3R8me2的二甲基化,这是通过一种Ⅱ型精氨酸甲基转移酶5(PRMT5)实现的[70]。H3R8me2标记后,其可以通过染色质重塑酶(BRG1)识别修饰的组蛋白“尾巴”,使染色体重塑复合体(SWI/SNF)稳定地结合。之后SWI/SNF与启动子结合并允许RNA聚合酶结合到DNA上进行转录。此外组蛋白转移酶(SETD7)被Myod1靶向Myog启动子引入H3K4me1标记,SETD7蛋白是分化所必需的,因为其沉默会导致肌管数量减少和阻断Myog的表达[60]。
最后,当完全分化时,成肌细胞在肌肉特异性膜蛋白Myomarker和Myomerger的作用下融合形成多核肌纤维。目前对于Myomarker和Myomerger蛋白的表观遗传调控尚不清楚,但是正向调控的CDON膜蛋白已经被证明受组氨酸甲基转移酶的调节,它在转录起始点沉积了H3K36me2的二甲基化,以防止多梳介导的基因沉默[71]。而且在不影响重链肌球蛋白(MHC)表达的条件下组蛋白甲基化酶(ASH1L)缺失会导致体外培养的小鼠和人的心肌细胞融合能力下降,但不会影响MHC的表达。由此可以想象,表观遗传调节细胞融合的机制将很快被研究。
4 小结哺乳动物骨骼肌发育是一个非常复杂的生物学过程,对于骨骼肌卫星细胞参与修复骨骼肌再生的机制早在很久以前便开始研究了。组蛋白修饰能够通过改变残基上的修饰状态来实现差异基因表达,如H3K27me3,其可以与成肌分化相关基因特异性结合,起到调节骨骼肌增殖分化的作用。MRFs发现以后,对组蛋白修饰调控肌卫星细胞的遗传机制有了更深的了解,同时对在肌肉再生过程中组蛋白转录后调控有了进一步的见解,但其对骨骼肌发育机制的具体调控过程尚不清楚。本文分别阐述了组蛋白修饰在静息、激活、增殖和分化状态下是如何调控骨骼肌再生过程中肌卫星细胞命运的,进一步说明了组蛋白修饰在肌肉再生过程中的重要作用,能够更好地理解组蛋白修饰与骨骼肌卫星细胞增殖分化机制的直接关系,为深入了解骨骼肌卫星细胞命运和肌肉高效再生提供新的研究方向。但是,组蛋白修饰调控肌卫星细胞在肌肉再生过程中的发生机制具有很强的复杂性,这些机制中任何一种发生失调都可能导致生肌程序的异常,从而无法修复受损的肌纤维。因此,组蛋白修饰调控骨骼肌发育机制研究还需进一步深入,为促进哺乳动物个体生长、改善肌肉再生和促进病理状态下肌肉平衡提供解决途径。
| [1] |
SPALDING K L, BHARDWAJ R D, BUCHHOLZ B A, et al. Retrospective birth dating of cells in humans[J]. Cell, 2005, 122(1): 133-143. DOI:10.1016/j.cell.2005.04.028 |
| [2] |
MOSS F P, LEBLOND C P. Satellite cells as the source of nuclei in muscles of growing rats[J]. The Anatomical Record-Advances in Integrative Anatomy and Evolutionary Biology, 1971, 170(4): 421-435. |
| [3] |
SNOW M H. An autoradiographic study of satellite cell differentiation into regenerating myotubes following transplantation of muscles in young rats[J]. Cell and Tissue Research, 1978, 186(3): 535-540. |
| [4] |
SAKUMA K, YAMAGUCHI A. Recent advances in pharmacological, hormonal, and nutritional intervention for sarcopenia[J]. Pflügers Archiv-European Journal of Physiology, 2018, 470(3): 449-460. DOI:10.1007/s00424-017-2077-9 |
| [5] |
SEGALÉS J, PERDIGUERO E, MUÑOZ-CÁNOVES P. Epigenetic control of adult skeletal muscle stem cell functions[J]. The FEBS Journal, 2015, 282(9): 1571-1588. DOI:10.1111/febs.13065 |
| [6] |
BIGOT A, DUDDY W J, OUANDAOGO Z G, et al. Age-associated methylation suppresses SPRY1, leading to a failure of re-quiescence and loss of the reserve stem cell pool in elderly muscle[J]. Cell Reports, 2015, 13(6): 1172-1182. DOI:10.1016/j.celrep.2015.09.067 |
| [7] |
FATICA A, BOZZONI I. Long non-coding RNAs: new players in cell differentiation and development[J]. Nature Reviews Genetics, 2014, 15(1): 7-21. DOI:10.1038/nrg3606 |
| [8] |
CONSALVI S, BRANCACCIO A, DALL'AGNESE A, et al. Praja1 E3 ubiquitin ligase promotes skeletal myogenesis through degradation of EZH2 upon p38α activation[J]. Nature Communications, 2017, 8: 13956. DOI:10.1038/ncomms13956 |
| [9] |
WANG L N, WANG Z, YANG K L, et al. Epigallocatechin gallate reduces slow-twitch muscle fiber formation and mitochondrial biosynthesis in C2C12 cells by repressing AMPK activity and PGC-1α expression[J]. Journal of Agricultural and Food Chemistry, 2016, 64(34): 6517-6523. DOI:10.1021/acs.jafc.6b02193 |
| [10] |
IRAZOQUI A P, BOLAND R L, BUITRAGO C G. Actions of 1, 25(OH)2-vitamin D3 on the cellular cycle depend on VDR and p38 MAPK in skeletal muscle cells[J]. Journal of Molecular Endocrinology, 2014, 53(3): 331-343. DOI:10.1530/JME-14-0102 |
| [11] |
JIN C L, ZHANG Z M, SONG Z W, et al. mTORC1-mediated satellite cell differentiation is required for lysine-induced skeletal muscle growth[J]. Journal of Agricultural and Food Chemistry, 2020, 68(17): 4884-4892. DOI:10.1021/acs.jafc.0c01275 |
| [12] |
CHEN X L, XIANG L, JIA G, et al. Leucine regulates slow-twitch muscle fibers expression and mitochondrial function by Sirt1/AMPK signaling in porcine skeletal muscle satellite cells[J]. Animal Science Journal, 2019, 90(2): 255-263. DOI:10.1111/asj.13146 |
| [13] |
NARLIKAR G J, FAN H Y, KINGSTON R E. Cooperation between complexes that regulate chromatin structure and transcription[J]. Cell, 2002, 108(4): 475-487. DOI:10.1016/S0092-8674(02)00654-2 |
| [14] |
JIN W, PENG J, JIANG S W. The epigenetic regulation of embryonic myogenesis and adult muscle regeneration by histone methylation modification[J]. Biochemistry and Biophysics Reports, 2016, 6: 209-219. DOI:10.1016/j.bbrep.2016.04.009 |
| [15] |
FRONTERA W R, OCHALA J. Skeletal muscle: a brief review of structure and function[J]. Calcified Tissue International, 2015, 96(3): 183-195. DOI:10.1007/s00223-014-9915-y |
| [16] |
TANG Z L, LI Y, WAN P, et al. LongSAGE analysis of skeletal muscle at three prenatal stages in Tongcheng and Landrace pigs[J]. Genome Biology, 2007, 8(6): R115. DOI:10.1186/gb-2007-8-6-r115 |
| [17] |
ALMADA A E, WAGERS A J. Molecular circuitry of stem cell fate in skeletal muscle regeneration, ageing and disease[J]. Nature Reviews Molecular Cell Biology, 2016, 17(5): 267-279. DOI:10.1038/nrm.2016.7 |
| [18] |
SINGH K, DILWORTH F J. Differential modulation of cell cycle progression distinguishes members of the myogenic regulatory factor family of transcription factors[J]. The FEBS Journal, 2013, 280(17): 3991-4003. DOI:10.1111/febs.12188 |
| [19] |
SOLEIMANI V D, PUNCH V G, KAWABE Y I, et al. Transcriptional dominance of Pax7 in adult myogenesis is due to high-affinity recognition of homeodomain motifs[J]. Developmental Cell, 2012, 22(6): 1208-1220. DOI:10.1016/j.devcel.2012.03.014 |
| [20] |
GVNTHER S, KIM J, KOSTIN S, et al. Myf5-positive satellite cells contribute to Pax7-dependent long-term maintenance of adult muscle stem cells[J]. Cell Stem Cell, 2013, 13(5): 590-601. DOI:10.1016/j.stem.2013.07.016 |
| [21] |
GARCÍA-PRAT L, PERDIGUERO E, ALONSO-MARTÍN S, et al. FoxO maintains a genuine muscle stem-cell quiescent state until geriatric age[J]. Nature Cell Biology, 2020, 22(11): 1307-1318. DOI:10.1038/s41556-020-00593-7 |
| [22] |
BEAUCHAMP J R, HESLOP L, YU D S, et al. Expression of CD34 and Myf5 defines the majority of quiescent adult skeletal muscle satellite cells[J]. Journal of Cell Biology, 2000, 151(6): 1221-1234. DOI:10.1083/jcb.151.6.1221 |
| [23] |
YABLONKA-REUVENI Z, RUDNICKI M A, RIVERA A J, et al. The transition from proliferation to differentiation is delayed in satellite cells from mice lacking MyoD[J]. Developmental Biology, 1999, 210(2): 440-455. DOI:10.1006/dbio.1999.9284 |
| [24] |
HINTERBERGER T J, SASSOON D A, RHODES S J, et al. Expression of the muscle regulatory factor MRF4 during somite and skeletal myofiber development[J]. Developmental Biology, 1991, 147(1): 144-156. DOI:10.1016/S0012-1606(05)80014-4 |
| [25] |
LAZURE F, BLACKBURN D M, CORCHADO A H, et al. Myf6/MRF4 is a myogenic niche regulator required for the maintenance of the muscle stem cell pool[J]. EMBO Reports, 2020, 21(12): e49499. |
| [26] |
ZHU Z, MILLER J B. MRF4 can substitute for myogenin during early stages of myogenesis[J]. Developmental Dynamics, 1997, 209(2): 233-241. DOI:10.1002/(SICI)1097-0177(199706)209:2<233::AID-AJA9>3.0.CO;2-J |
| [27] |
CAO Y, YAO Z Z, SARKAR D, et al. Genome-wide MyoD binding in skeletal muscle cells: a potential for broad cellular reprogramming[J]. Developmental Cell, 2010, 18(4): 662-674. DOI:10.1016/j.devcel.2010.02.014 |
| [28] |
BLUM R, VETHANTHAM V, BOWMAN C, et al. Genome-wide identification of enhancers in skeletal muscle: the role of MyoD1[J]. Genes & Development, 2012, 26(24): 2763-2779. |
| [29] |
LLUÍS F, PERDIGUERO E, NEBREDA A R, et al. Regulation of skeletal muscle gene expression by p38 MAP kinases[J]. Trends in Cell Biology, 2006, 16(1): 36-44. DOI:10.1016/j.tcb.2005.11.002 |
| [30] |
MACHADO L, ESTEVES DE LIMA J, FABRE O, et al. In situ fixation redefines quiescence and early activation of skeletal muscle stem cells[J]. Cell Reports, 2017, 21(7): 1982-1993. DOI:10.1016/j.celrep.2017.10.080 |
| [31] |
YUE L, WAN R, LUAN S Y, et al. Dek modulates global intron retention during muscle stem cells quiescence exit[J]. Developmental Cell, 2020, 53(6): 661-676. DOI:10.1016/j.devcel.2020.05.006 |
| [32] |
MASSENET J, GARDNER E, CHAZAUD B, et al. Epigenetic regulation of satellite cell fate during skeletal muscle regeneration[J]. Skeletal Muscle, 2021, 11(1): 4. DOI:10.1186/s13395-020-00259-w |
| [33] |
VAN VELTHOVEN C T J, DE MORREE A, EGNER I M, et al. Transcriptional profiling of quiescent muscle stem cells in vivo[J]. Cell Reports, 2017, 21(7): 1994-2004. DOI:10.1016/j.celrep.2017.10.037 |
| [34] |
LIU L, CHEUNG T H, CHARVILLE G W, et al. Chromatin modifications as determinants of muscle stem cell quiescence and chronological aging[J]. Cell Reports, 2013, 4(1): 189-204. DOI:10.1016/j.celrep.2013.05.043 |
| [35] |
LIU L, CHEUNG T H, CHARVILLE G W, et al. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting[J]. Nature Protocols, 2015, 10(10): 1612-1624. DOI:10.1038/nprot.2015.110 |
| [36] |
SOUSA-VICTOR P, GUTARRA S, GARCÍA-PRAT L, et al. Geriatric muscle stem cells switch reversible quiescence into senescence[J]. Nature, 2014, 506(7488): 316-321. DOI:10.1038/nature13013 |
| [37] |
GOPINATH S D, WEBB A E, BRUNET A, et al. FoxO3 promotes quiescence in adult muscle stem cells during the process of self-renewal[J]. Stem Cell Reports, 2014, 2(4): 414-426. DOI:10.1016/j.stemcr.2014.02.002 |
| [38] |
BJORNSON C R R, CHEUNG T H, LIU L, et al. Notch signaling is necessary to maintain quiescence in adult muscle stem cells[J]. Stem Cells, 2012, 30(2): 232-242. DOI:10.1002/stem.773 |
| [39] |
BRACK A S, RANDO T A. Tissue-specific stem cells: lessons from the skeletal muscle satellite cell[J]. Cell Stem Cell, 2012, 10(5): 504-514. DOI:10.1016/j.stem.2012.04.001 |
| [40] |
BOONSANAY V, ZHANG T, GEORGIEVA A, et al. Regulation of skeletal muscle stem cell quiescence by Suv4-20h1-dependent facultative heterochromatin formation[J]. Cell Stem Cell, 2016, 18(2): 229-242. DOI:10.1016/j.stem.2015.11.002 |
| [41] |
JUAN A H, DERFOUL A, FENG X S, et al. Polycomb EZH2 controls self-renewal and safeguards the transcriptional identity of skeletal muscle stem cells[J]. Genes & Development, 2011, 25(8): 789-794. |
| [42] |
LI Y F, DILWORTH F J. Compacting chromatin to ensure muscle satellite cell quiescence[J]. Cell Stem Cell, 2016, 18(2): 162-164. DOI:10.1016/j.stem.2016.01.009 |
| [43] |
DILWORTH F J, BLAIS A. Epigenetic regulation of satellite cell activation during muscle regeneration[J]. Stem Cell Research & Therapy, 2011, 2(2): 18. |
| [44] |
ADDICKS G C, BRUN C E, SINCENNES M C, et al. MLL1 is required for PAX7 expression and satellite cell self-renewal in mice[J]. Nature Communications, 2019, 10(1): 4256. DOI:10.1038/s41467-019-12086-9 |
| [45] |
PALACIOS D, MOZZETTA C, CONSALVI S, et al. TNF/p38α/polycomb signaling to Pax7 locus in satellite cells links inflammation to the epigenetic control of muscle regeneration[J]. Cell Stem Cell, 2010, 7(4): 455-469. DOI:10.1016/j.stem.2010.08.013 |
| [46] |
WOODHOUSE S, PUGAZHENDHI D, BRIEN P, et al. Ezh2 maintains a key phase of muscle satellite cell expansion but does not regulate terminal differentiation[J]. Journal of Cell Science, 2013, 126(Pt 2): 565-579. |
| [47] |
LEE S H, JOO S T, RYU Y C. Skeletal muscle fiber type and myofibrillar proteins in relation to meat quality[J]. Meat Science, 2010, 86(1): 166-170. DOI:10.1016/j.meatsci.2010.04.040 |
| [48] |
SINGH K, CASSANO M, PLANET E, et al. A KAP1 phosphorylation switch controls MyoD function during skeletal muscle differentiation[J]. Genes & Development, 2015, 29(5): 513-525. |
| [49] |
PURI P L, IEZZI S, STIEGLER P, et al. Class I histone deacetylases sequentially interact with MyoD and pRb during skeletal myogenesis[J]. Molecular Cell, 2001, 8(4): 885-897. DOI:10.1016/S1097-2765(01)00373-2 |
| [50] |
LING B M T, BHARATHY N, CHUNG T K, et al. Lysine methyltransferase G9a methylates the transcription factor MyoD and regulates skeletal muscle differentiation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(3): 841-846. DOI:10.1073/pnas.1111628109 |
| [51] |
DILWORTH F J, SEAVER K J, FISHBURN A L, et al. In vitro transcription system delineates the distinct roles of the coactivators pCAF and p300 during MyoD/E47-dependent transactivation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(32): 11593-11598. DOI:10.1073/pnas.0404192101 |
| [52] |
CHOI J, JANG H, KIM H, et al. Histone demethylase LSD1 is required to induce skeletal muscle differentiation by regulating myogenic factors[J]. Biochemical and Biophysical Research Communications, 2010, 401(3): 327-332. DOI:10.1016/j.bbrc.2010.09.014 |
| [53] |
SOLEIMANI V D, YIN H, JAHANI-ASL A, et al. Snail regulates MyoD binding-site occupancy to direct enhancer switching and differentiation-specific transcription in myogenesis[J]. Molecular Cell, 2012, 47(3): 457-468. DOI:10.1016/j.molcel.2012.05.046 |
| [54] |
LUO D, DE MORREE A, BOUTET S, et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(15): E3071-E3080. |
| [55] |
TAKAHASHI Y, RAYMAN J B, DYNLACHT B D. Analysis of promoter binding by the E2F and pRB families in vivo: distinct E2F proteins mediate activation and repression[J]. Genes & Development, 2000, 14(7): 804-816. |
| [56] |
TAUBERT S, GORRINI C, FRANK S R, et al. E2F-dependent histone acetylation and recruitment of the Tip60 acetyltransferase complex to chromatin in late G1[J]. Molecular and Cellular Biology, 2004, 24(10): 4546-4556. DOI:10.1128/MCB.24.10.4546-4556.2004 |
| [57] |
RAO V K, OW J R, SHANKAR S R, et al. G9a promotes proliferation and inhibits cell cycle exit during myogenic differentiation[J]. Nucleic Acids Research, 2016, 44(17): 8129-8143. DOI:10.1093/nar/gkw483 |
| [58] |
NIGHTINGALE K P, GENDREIZIG S, WHITE D A, et al. Cross-talk between histone modifications in response to histone deacetylase inhibitors: MLL4 links histone H3 acetylation and histone H3K4 methylation[J]. Journal of Biological Chemistry, 2007, 282(7): 4408-4416. DOI:10.1074/jbc.M606773200 |
| [59] |
SEBASTIAN S, SREENIVAS P, SAMBASIVAN R, et al. MLL5, a trithorax homolog, indirectly regulates H3K4 methylation, represses cyclin A2 expression, and promotes myogenic differentiation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(12): 4719-4724. DOI:10.1073/pnas.0807136106 |
| [60] |
YI X, TAO Y, LIN X, et al. Histone methyltransferase Setd2 is critical for the proliferation and differentiation of myoblasts[J]. Biochimica et Biophysica Acta-Molecular Cell Research, 2017, 1864(4): 697-707. DOI:10.1016/j.bbamcr.2017.01.012 |
| [61] |
BYRNE K, MCWILLIAM S, VUOCOLO T, et al. Genomic architecture of histone 3 lysine 27 trimethylation during late ovine skeletal muscle development[J]. Animal Genetics, 2014, 45(3): 427-438. DOI:10.1111/age.12145 |
| [62] |
BLAIS A, DYNLACHT B D. E2F-associated chromatin modifiers and cell cycle control[J]. Current Opinion in Cell Biology, 2007, 19(6): 658-662. DOI:10.1016/j.ceb.2007.10.003 |
| [63] |
BLAIS A, VAN OEVELEN C J C, MARGUERON R, et al. Retinoblastoma tumor suppressor protein-dependent methylation of histone H3 lysine 27 is associated with irreversible cell cycle exit[J]. Journal of Cell Biology, 2007, 179(7): 1399-1412. DOI:10.1083/jcb.200705051 |
| [64] |
BALCIUNAITE E, SPEKTOR A, LENTS N H, et al. Pocket protein complexes are recruited to distinct targets in quiescent and proliferating cells[J]. Molecular and Cellular Biology, 2005, 25(18): 8166-8178. DOI:10.1128/MCB.25.18.8166-8178.2005 |
| [65] |
DIMOVA D K, STEVAUX O, FROLOV M V, et al. Cell cycle-dependent and cell cycle-independent control of transcription by the Drosophila E2F/RB pathway[J]. Genes & Development, 2003, 17(18): 2308-2320. |
| [66] |
HSU J, ARAND J, CHAIKOVSKY A, et al. E2F4 regulates transcriptional activation in mouse embryonic stem cells independently of the RB family[J]. Nature Communications, 2019, 10(1): 2939. DOI:10.1038/s41467-019-10901-x |
| [67] |
SCHWARTZ Y B, PIRROTTA V. Polycomb silencing mechanisms and the management of genomic programmes[J]. Nature Reviews Genetics, 2007, 8(1): 9-22. DOI:10.1038/nrg1981 |
| [68] |
TROJER P, LI G H, SIMS R J, et al. L3MBTL1, a histone-methylation-dependent chromatin lock[J]. Cell, 2007, 129(5): 915-928. DOI:10.1016/j.cell.2007.03.048 |
| [69] |
VERRIER L, ESCAFFIT F, CHAILLEUX C, et al. A new isoform of the histone demethylase JMJD2A/KDM4A is required for skeletal muscle differentiation[J]. Plos Genetics, 2011, 7(6): e1001390. DOI:10.1371/journal.pgen.1001390 |
| [70] |
DACWAG C S, OHKAWA Y, PAL S, et al. The protein arginine methyltransferase Prmt5 is required for myogenesis because it facilitates ATP-dependent chromatin remodeling[J]. Molecular and Cellular Biology, 2007, 27(1): 384-394. DOI:10.1128/MCB.01528-06 |
| [71] |
CASTIGLIONI I, CACCIA R, GARCIA-MANTEIGA J M, et al. The trithorax protein ash1L promotes myoblast fusion by activating Cdon expression[J]. Nature Communications, 2018, 9(1): 5026. DOI:10.1038/s41467-018-07313-8 |




