2. 姜堰区白米镇畜牧兽医站, 泰州 225505;
3. 南京卫岗乳业有限公司, 南京 210008
2. Baimi Animal Husbandry and Veterinary Station in Jiangyan District, Taizhou 225505, China;
3. Nanjing Weigang Dairy Co., Ltd., Nanjing 210008, China
乳脂是牛奶中重要的营养物质之一,含量一般在3%~5%,其主要成分为甘油三酯。乳脂率是评价奶牛生产性能的重要指标,也是影响奶牛产业经济效益的重要因素[1]。乳脂的合成部位是乳腺上皮细胞。用于乳脂合成的脂肪酸主要来源于2种途径:瘤胃内的微生物合成和外源摄取[2]。乙酸和丁酸是瘤胃发酵的产物,其中丁酸通过瘤胃上皮细胞的消化吸收可以转换成β-羟丁酸。乙酸和β-羟丁酸经血液运输至乳腺内,可作为底物合成中短链脂肪酸,而长链脂肪酸则来自于膳食脂质和体脂[3]。乳脂率受到多种因素的影响,如品种、饲粮结构、饲养管理以及奶牛健康状况、胎次、泌乳阶段等[4]。近期研究表明瘤胃微生物及其代谢物对乳脂代谢具有重要的影响[1]。厚壁菌门与拟杆菌门的比例和乳脂产量呈显著正相关,而拟杆菌门中普雷沃氏菌属的比例和乳脂产量呈显著负相关[5-7]。Pitta等[8]发现乳脂率与瘤胃内小杆菌属(Dialister)、巨球型菌属(Megasphaera)、毛螺旋菌属(Lachnospira)、夏普氏菌属(Sharpea)的丰度有关。瘤胃微生物代谢物——共轭亚油酸,可抑制乳腺细胞内脂质合成基因的表达,进而降低乳脂率[9]。脂质组分析显示高乳脂率奶牛和低乳脂率奶牛瘤胃内容物中主要的差异脂质代谢物为甘油三酯和磷脂酰乙醇胺[4]。但调控乳脂代谢的瘤胃微生物、瘤胃代谢物及二者之间潜在的关系鲜有报道。因此,本研究在相同饲粮及饲养管理条件下,采用瘤胃微生物16S rDNA测序技术及代谢组学技术,分析高乳脂率奶牛和低乳脂率奶牛瘤胃微生物及代谢物的不同,探究瘤胃差异微生物及差异代谢物调控乳脂合成的机制,为进一步提升原料乳品质提供理论基础。
1 材料与方法 1.1 试验动物与样品采集基于江苏省某中等规模奶牛场2020年的生产性能测定(DHI)数据,从532头泌乳奶牛中选取1~3胎次、泌乳中后期且产奶量相近的乳脂率长期偏高的奶牛(H组)和乳脂率长期偏低的奶牛(L组)各5头,每日采用全混合日粮(TMR)饲喂2次。参照GB/T 6432—1994方法测定粗蛋白质含量,参照GB/T 6433—2006方法测定粗脂肪含量,参照GB/T 6434—2006方法测定中性洗涤纤维和酸性洗涤纤维含量,参照GB/T 6435—2002方法测定钙含量,参照GB/T 6436—2002方法测定磷含量,饲粮组成及营养水平见表 1。2021年1月16日采集奶样,早晚各取100 mL,混匀后用于乳品质指标的测定。饲喂2 h后固定牛只,通过牛口腔导管采集瘤胃液50 mL,分为2份,一份利用pH计测定瘤胃液pH,另一份立即液氮保存,用于瘤胃微生物组成及代谢组的测定。
|
|
表 1 饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the diet (DM basis) |
利用MilkoScan FT-1型多功能乳品分析仪(multifunctional dairy analyzer,Foss Electric, 丹麦)测定常规乳成分含量。体细胞评分(SCS)则根据以下公式计算:

|
瘤胃液解冻后离心(10 000×g,10 min),取上清液1 mL,加0.2 mL 20%含60 mmol/L巴豆酸的偏磷酸,混匀后在-20 ℃过夜。随后将样品离心(10 000×g,10 min),上清过滤后取1 μL进行气相色谱检测,检测仪器为7890-B气相色谱仪(GC, Agilent, 美国),色谱柱为DB-WAX型毛细管气相色谱柱(30 m×0.25 mm×0.25 μm, Agilent, 美国),具体检测步骤参照文献[10]进行。
1.4 Illumina测序采用HiPure Stool DNA提取试剂盒(广州美基生物科技有限公司)提取瘤胃微生物DNA,具体方法参照说明书。选择特异性引物扩增16S rDNA的V3+V4区。引物序列为:341F,5’-CCTACGGGNGGCWGCAG-3’;806R,5’-GGACTACHVGGGTATCTAAT-3’。PCR扩增条件:95 ℃ 5 min;95 ℃ 1 min,60 ℃ 1 min,72 ℃ 1 min进行30个循环,最后72 ℃ 7 min。将纯化后的扩增产物在ABI StepOnePlus实时PCR系统(Life Technologies, 美国)进行定量分析。纯化后的扩增子根据标准操作在Illumina平台上进行双端测序(PE250),测序服务委托广州基迪奥生物科技有限公司。
1.5 非靶代谢组学分析瘤胃液经预处理后进行液相色谱-质谱(LC-MS)检测,色谱仪为Vanquish UHPLC(Thermo Fisher, 德国),质谱仪为Q ExactiveTM HF-X(Thermo Fisher, 德国),色谱柱为Hypesil Gold column(100 mm×2.1 mm,1.9 μm,Thermo Fisher,美国),质谱扫描范围选择质荷比(m/z) 100~15 00,代谢组测定及分析委托广州基迪奥生物科技有限公司。
1.6 瘤胃液消化酶活性测定瘤胃液解冻后离心,取上清,采用酶联免疫吸附测定试剂盒对4种消化酶的活性进行测定,其中纤维素酶及脂肪酶活性测定试剂盒购自上海邦奕生物科技有限公司,木聚糖酶及果胶酶活性测定试剂盒购自上海劲马实验设备有限公司,具体操作按试剂盒说明书进行。
1.7 数据分析 1.7.1 试验牛基本信息分析采用SPSS 16.0软件中的单因素方差分析(one-way ANOVA)模型分析2组奶牛胎次、泌乳天数、日产奶量、乳成分、瘤胃液VFA浓度及消化酶活性的组间差异,所有数据结果用平均值±标准误表示,P<0.01表示差异极显著,P<0.05表示差异显著,0.05≤P<0.10表示差异有显著趋势。
1.7.2 16S rDNA测序数据分析首先对原始数据进行质控处理,并聚类、去嵌合体,获得操作分类单元(OTU)代表序列和OTU丰度信息,比对SILVA数据库(132)获得物种分类注释信息。根据OTU丰度信息获得各层级的物种丰度信息。基于OTU和物种丰度表,使用R语言VennDiagram包(1.6.16)进行韦恩(Venn)分析;使用R语言ggplot2包(2.2.1)绘制物种丰度堆叠图;使用QIIME(1.9.1)分析α多样性指数,利用R语言Vegan包(2.5.3)分析2组间α多样性指数差异、物种丰度差异并进行主坐标分析(PCoA)。
1.7.3 代谢物鉴定及分析质谱下机后的原始数据经代谢组学处理软件progenesis QI进行基线过滤、峰识别、积分、保留时间校正、峰对齐和归一化,最终得到一个包含保留时间、质荷比和峰强度的数据矩阵。将其与mzCloud、mzVault和Masslist数据库进行比对,最后得到代谢物的名称及定量结果,质荷比质量偏差设置为5 ppm。使用R语言randomForest包进行随机森林分析,确定区分2组样本间差异的关键代谢物,并结合差异倍数(fold change,FC)值和SPSS 16.0中的t检验(P值)来筛选2组间的差异代谢物。应用MetaboAnalyst 5.0分析差异代谢物显著性富集的通路。
1.7.4 瘤胃微生物及代谢组学关联分析利用R语言cor.test函数计算样本中物种和代谢物的Pearson相关系数,筛选出相关系数绝对值大于0.5的微生物与代谢物数据关系对,使用R语言igraph包(1.1.1)绘制网络图。
2 结果与分析 2.1 乳成分分析如表 2所示,2组试验牛的胎次、泌乳天数及日产奶量差异不显著(P>0.05)。2020年2月至2021年1月的全年平均乳脂率差异极显著(P < 0.01),H组全年平均乳脂率为4.55%,极显著高于L组的3.51%(P < 0.01)。在测定日(2021年1月16日),H组的乳脂率(5.82%)极显著高于L组(3.60%),全乳固体含量(16.50%)显著高于L组(13.80%)(P < 0.05),其他乳成分指标在2组间差异不显著(P>0.05)。
|
|
表 2 试验牛基本信息 Table 2 Basic information of experimental dairy cows |
由表 3可知,L组和H组瘤胃液pH分别为6.51±0.16和6.64±0.13,二者差异不显著(P>0.05);与L组相比,H组瘤胃液乙酸/丙酸比值显著升高(P < 0.05);此外,2组间瘤胃液乙酸、丙酸、异丁酸、丁酸、异戊酸、戊酸及总挥发性脂肪酸浓度无显著差异(P>0.05)。
|
|
表 3 不同乳脂率奶牛瘤胃液VFA浓度的比较分析 Table 3 Comparative analysis of volatile fatty acid concentrations in ruminal fluid of dairy cows with different milk fat ratios |
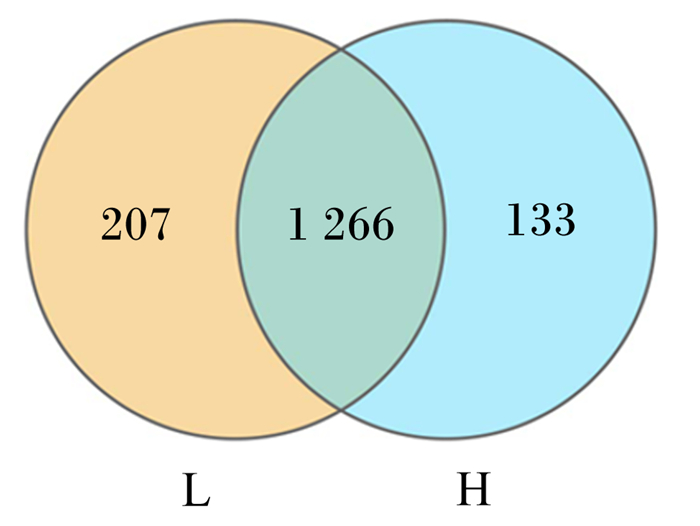
测序数据经质控处理后,对10个瘤胃液样品的有效序列进行OTU聚类分析及去嵌合体处理,共得到1 606个OTU(图 1)。2组共有OTU个数为1 266个,H组奶牛OTU个数要略低于L组奶牛。
|
L:L组;H:H组。下图同。 L: L group; H: H group. The same as below. 图 1 奶牛瘤胃菌群OTU韦恩图 Fig. 1 Venn graph of rumen bacterial community OTU of dairy cows |
基于OTU和物种丰度进行瘤胃菌群的α多样性分析,结果显示2组的α多样性指数(Sobs、Shannon、Simpson、Chao1、Ace指数)之间均不存在显著差异(P>0.05,表 4),表明L组和H组奶牛瘤胃菌群的丰富度与多样性无显著差异。
|
|
表 4 瘤胃菌群α多样性指数 Table 4 α diversity indexes of rumen bacterial community |
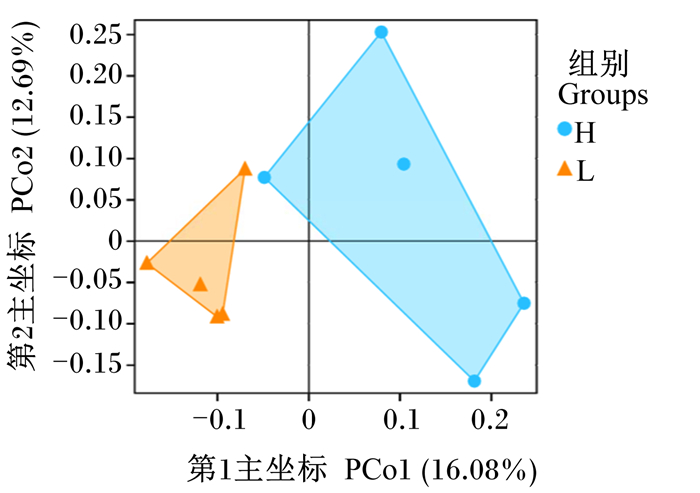
基于Unweighted UniFrac距离的PCoA结果如图 2所示,第1主坐标(PCo1)和第2主坐标(PCo2)的贡献率分别为16.08%和12.69%,且L组和H组奶牛瘤胃菌群被明显区分开,说明L组和H组之间的瘤胃菌群结构有较大差异。PCoA分析中群落结构相似度高的样品倾向于聚集在一起,反之,群落差异越大,则距离较远。由此可知,H组奶牛瘤胃菌群结构差异要大于L组。
|
图 2 奶牛瘤胃菌群PCoA图 Fig. 2 PCoA graph of rumen bacterial community of dairy cows |
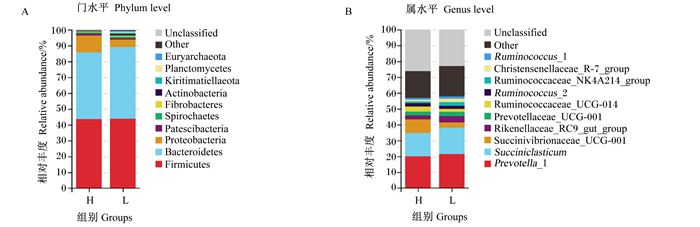
在门水平上,10个瘤胃液样品共检测到22个菌门,其中相对丰度较高的10个菌门如图 3-A所示。厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)相对丰度之和占到总菌的93%以上。与L组相比,H组奶牛瘤胃中Kiritimatiellaeota的相对丰度显著降低(P < 0.05),其余菌门的相对丰度2组间无显著差异(P>0.05)。在属水平上,10个瘤胃液样品共检测到157个菌属,其中相对丰度较高的10个菌属如图 3-B所示。普雷沃氏菌属1(Prevotella_1)的相对丰度最高,可达到总菌的20.66%,其次是解琥珀酸弧菌属(Succiniclasticum,15.69%)和琥珀酸弧菌科UCG-001(Succinivibrionaceae_UCG-001,6.02%)。
|
Firmicutes:厚壁菌门;Bacteroidetes:拟杆菌门;Proteobacteria:变形菌门;Spirochaetes:螺旋体门;Fibrobacteres:纤维杆菌门;Actinobacteria:放线菌门;Planctomycetes:浮霉菌门;Euryarchaeota:广古菌门;Other:其他;Unclassified:未分类;Prevotella_1:普雷沃氏菌属1;Succiniclasticum:解琥珀酸弧菌属;Succinivibrionaceae_UCG-001:琥珀酸弧菌科UCG-001;Rikenellaceae_RC9_gut_group:理研菌科RC9肠道群;Prevotellaceae_UCG-001:普雷沃氏菌科UCG-001;Ruminococcaceae_UCG-014:瘤胃球菌科UCG-014;Ruminococcus_2:瘤胃球菌属2;Ruminococcaceae_NK4A214_group:瘤胃球菌科NK4A214群;Christensenellaceae_R-7_group:克里斯滕森菌科R-7群;Ruminococcus_1:瘤胃球菌属1。 图 3 奶牛瘤胃细菌物种分布堆叠图 Fig. 3 Stack diagram of rumen bacterial species distribution of dairy cows |
如图 4所示,在属水平上,与L组相比,H组奶牛瘤胃中嗜木聚糖真杆菌群(Eubacterium_xylanophilum_group)丰度显著增加(P < 0.05),克里斯滕森菌科R-7群(Christensenellaceae_R-7_group)、产粪甾醇真杆菌群(Eubacterium_coprostanoligenes_group)、瘤胃球菌科UCG-010(Ruminococcaceae_UCG-010)、丁酸弧菌属2(Butyrivibrio_2)、产甲烷短杆菌属(Methanobrevibacter)、p-1088-a5_gut_group、聚乙酸菌属(Acetitomaculum)、毛螺梭菌属10(Lachnoclostridium_10)、毛螺旋菌科ND3007群(Lachnospiraceae_ND3007_group)的丰度显著降低(P < 0.05)。

|
Christensenellaceae_R-7_group:克里斯滕森菌科R-7群;Eubacterium_coprostanoligenes_group:产粪甾醇真杆菌群;Ruminococcaceae_UCG-010:瘤胃球菌科UCG-010;Butyrivibrio_2:丁酸弧菌属2;Methanobrevibacter:产甲烷短杆菌属;Acetitomaculum:聚乙酸菌属;Lachnoclostridium_10:毛螺梭菌属10;Lachnospiraceae_ND3007_group:毛螺旋菌科ND3007群;Eubacterium_xylanophilum_group:嗜木聚糖真杆菌群。 图 4 H组与L组差异微生物 Fig. 4 Differential microbes between H group and L group |
根据差异微生物的种类,进行了瘤胃液部分消化酶活性测定,结果如表 5所示。H组瘤胃液木聚糖酶活性为36.69 U/L,显著高于L组的30.70 U/L(P < 0.05),但瘤胃液脂肪酶活性极显著低于L组(P < 0.01);此外,瘤胃液纤维素酶及果胶酶活性在2组间差异不显著(P>0.05)。
|
|
表 5 不同乳脂率奶牛瘤胃液消化酶的比较分析 Table 5 Comparative analysis of digestive enzyme activities in ruminal fluid of dairy cows with different milk fat ratios |
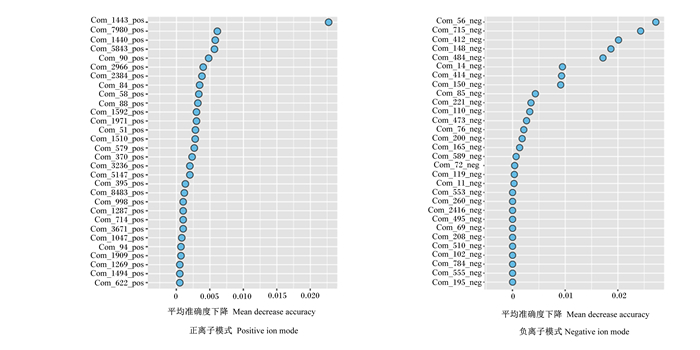
在正、负离子模式检测中,共发现了1 577个正离子,990个负离子。将这些瘤胃代谢物进行统计分析。采用随机森林对瘤胃代谢组学数据进行分类(表 6),正、负离子模式下,对L组和H组的预测准确率分别为40.00%和80.00%,且受试者工作特征曲线(receive operating characteristic curve,ROC曲线)分析显示曲线下面积分别为0.40和0.96,表明正离子模式下模型预测的准确率较低,而负离子模式下模型预测的准确率较高,具有较高的组间分辨能力,能够有效区分L组和H组。图 5显示了对L组与H组间鉴别贡献较大的前30种代谢物,以FC≥1.5且P < 0.05筛选组间差异代谢物,在负离子模式下鉴定出4个差异代谢物。与L组相比,H组内显著上调的差异代谢物有月桂酸、琥珀酸、月桂酸乙酯及12, 13-二羟基十八碳烯酸(P<0.05),这些代谢物主要为脂质及有机酸类,且富集在脂肪酸的生物合成及氨基酸的代谢等途径(表 7)。
|
|
表 6 随机森林对瘤胃代谢组学数据的分类结果 Table 6 Random forest classification results of rumen metabolomics data |
|
图 5 正、负离子模式下瘤胃代谢组学随机森林分析 Fig. 5 Random forest analysis of rumen metabolomics in positive and negative ion modes |
|
|
表 7 负离子模式下的差异代谢物 Table 7 Differential metabolites in negative ion mode |
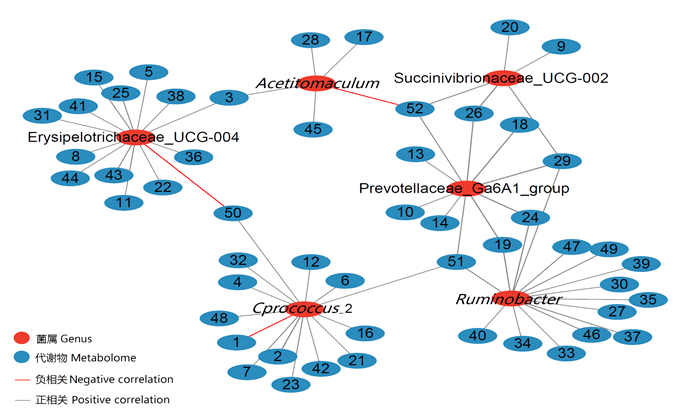
构建瘤胃微生物与其代谢物的网络图,可以展现处于重要关联位置的微生物或代谢物,筛选相关性系数>0.5且P < 0.05的微生物与代谢物数据关系对并绘制网络图(图 6)。月桂酸与粪球菌属2(Coprococcus_2)呈显著正相关(P<0.05),与丹毒丝菌科UCG-004(Erysipelotrichaceae_UCG-004)呈显著负相关(P<0.05)。琥珀酸与琥珀酸弧菌科UCG-002(Succinivibrionaceae_UCG-002)和普雷沃氏菌科Ga6A1群(Prevotellaceae_Ga6A1_group)呈显著正相关(P<0.05),但与Acetitomaculum呈显著负相关(P<0.05)。月桂酸乙酯与Prevotellaceae_Ga6A1_group、Coprococcus_2、瘤胃杆菌属(Ruminobacter)均呈显著正相关(P<0.05)。12, 13-二羟基十八碳烯酸与Coprococcus_2呈显著正相关(P<0.05)。
|
1:(卤素)12(13)-二羟基-9Z-十八烯酸(halogen)12(13)-DiHOME;2:维生素B1 thiamine;3:PC(18:0e/3:0);4:5-[(10Z)-14-(3, 5-二羟基苯基)十四-10-烯-1-基]苯-1, 3-二醇5-[(10Z)-14-(3, 5-dihydroxyphenyl)tetradec-10-en-1-yl]benzene-1, 3-diol;5:山梨糖sorbose,;6:脂多糖27:0 LPS27:0;7:缬氨酸-色氨酸Val-Ser;8:PC(16:0/16:1);9:LPE14:0;10:双环前列腺素E2 bicyclo prostaglandin E2;11:PC(15:0/16:0);12:石油醚(15:0-15:0) PEtOH(15:0-15:0);13:FMH;14:EPK;15:氧化聚乙烯(18:1-18:3+1O) OxPE(18:1-18:3+1O);16:PE(14:1e/10:0);17:PC(22:3e/16:1);18:胞嘧啶cytosine;19:十六烷二酸hexadecanedioic acid;20:7-氨基甲西泮7-aminonimetazepam;21:乙醇石胆酸glycolithocholic acid;22:PI(18:0/22:5);23:地氯雷他定desloratadine;24:N-[(4-羟基-3-甲氧基苯基)甲基]-8-甲基壬酰胺N-[(4-hydroxy-3-methoxyphenyl)methyl]-8-methylnonanamide;25:delta8-THC-d9;26:8-苯基-4, 5-二氢[1, 2, 5]恶二唑并[3, 4-f]肉桂啉8-phenyl-4, 5-dihydro[1, 2, 5]oxadiazolo[3, 4-f]cinnoline;27:新石斛毒素neosaxitoxin;28:4-(2-羟乙基)-2-甲氧基苯基β-D-吡喃葡萄糖苷4-(2-hydroxyethyl)-2-methoxyphenyl β-D-glucopyranoside;29:鸟嘌呤guanine;30:KPH;31:1-(2-呋喃基)-3, 3-二(甲硫基)丙-2-en-1-one 1-(2-furyl)-3, 3-di(methylthio)prop-2-en-1-one;32:粪臭素skatole;33:17β-群勃龙17beta-trenbolone;34:胞苷cytidine;35:PS(14:0/16:0);36:SM(d14:2/24:0);37:BMP(16:0/20:1);38:PE(15:0/18:2);39:HexCer-NDS (d16:0/16:0);40:5α-双氢睾酮5α-dihydrotestosterone;41:石油醚(21:0-20:4) PEtOH(21:0-20:4);42:2-[2-(5-溴-2-吡啶基)重氮-1-烯基]-5-(二乙氨基)苯酚2-[2-(5-bromo-2-pyridyl)diaz-1-enyl]-5-(diethylamino)phenol;43:PC(26:0/18:1);44:PC(14:0e/16:2);45:PC(6:0/22:6);46:BMP(16:0/20:2);47:黄尿酸xanthurenic acid;48:PE(12:0/12:0);49:烟酰胺nicotinamide;50:月桂酸lauric acid;51:月桂酸乙酯lauric acid ethyl ester;52:琥珀酸succinic acid。Coprococcus_2:粪球菌属2;Erysipelotrichaceae_UCG-004:丹毒丝菌科UCG-004;Succinivibrionaceae_UCG-002:琥珀酸弧菌科UCG-002;Prevotellaceae_Ga6A1_group:普雷沃氏菌科Ga6A1群;Acetitomaculum:聚乙酸菌属;Ruminobacter:瘤胃杆菌属。 图 6 瘤胃微生物与代谢物相关网络图 Fig. 6 Network of relationship between rumen microbes and metabolites |
瘤胃发酵过程中可将饲粮中的营养物质分解为乙酸、丙酸、丁酸等,乙酸是合成乳脂的重要前体物,而丙酸作为糖异生的主要底物通过肝脏参与乳糖的合成[11]。近年来,许多研究发现乙酸/丙酸比值可作为评价牛奶质量的综合指标,该比值与乳脂率呈正相关[11-12]。这些报道与本研究结果一致,H组的乙酸/丙酸比值显著高于L组,说明2组间瘤胃发酵类型不同。
3.2 瘤胃微生物对乳脂率的影响不同年龄奶牛瘤胃内优势菌门均为Firmicutes、Bacteroidetes和Proteobacteria,且Firmicutes/Bacteroidetes的比值与产奶性状相关[13]。Jami等[5]发现Firmicutes/Bacteroidetes的比值与乳脂率呈极显著正相关,这与本研究结果相一致,H组Firmicutes/Bacteroidetes的比值为1.04,高于L组的0.97。在门水平上,H组奶牛瘤胃中Kiritimatiellaeota的相对丰度低于L组,Stergiadis等[14]发现牛奶中饱和脂肪酸含量高的奶牛,其瘤胃内Kiritimatiellaeota的相对丰度显著高于饱和脂肪酸含量低的奶牛,由此可见,Kiritimatiellaeota可参与奶牛的乳脂代谢。在属水平上,H组奶牛瘤胃内Eubacterium_xylanophilum_group的丰度显著高于L组,该菌可发酵饲粮中的木聚糖生成甲酸、乙酸和丁酸[15]。瘤胃宏基因组分析表明奶牛瘤胃微生物及其代谢物的协同作用,可以增强半纤维素酶、脂质合成酶及转移酶的基因表达量,进而增强瘤胃降解饲粮营养物质的能力,最终促进乳脂率的提高[4]。由此可见,Eubacterium_xylanophilum_group可降解饲粮中的半纤维素,从而提高瘤胃内乙酸和丁酸的浓度,进而促进乳脂的合成。本研究中发现H组瘤胃液木聚糖酶活性要高于L组,与上述研究结果一致。而在L组丰度显著上升的Eubacterium_coprostanoligenes_group、Ruminococcaceae_UCG-010、Butyrivibrio_2为瘤胃微生物中主要的纤维素降解菌。这些微生物将饲粮中的纤维素类物质逐步降解为单糖供机体利用。
徐晓锋等[16]发现,通过高精料诱导的低乳脂奶牛,瘤胃内Eubacterium_coprostanoligenes_group的丰度较对照组增加了44.78%,但Christensenellaceae_R-7_group、Ruminococcaceae_UCG-010的丰度低于对照组,后2种微生物的变化规律与本研究不同,可能是由于饲粮组成不同导致。饲粮中添加淀粉后,牛奶乳脂率极显著降低,同时瘤胃内毛螺梭菌属-1(Lachnoclostridium-1)、瘤胃球菌属-1(Ruminococcus-1)的丰度极显著增加[17]。此外,Li等[18]发现毛螺梭菌属(Lachnoclostridium)、毛螺旋菌科UCG-006(Lachnospiraceae UCG-006)可抑制短链脂肪酸生成菌,进而抑制乳脂的合成。许多研究显示丁酸弧菌属(Butyrivibrio)参与生物氢化过程,因此可影响乳脂生成[19]。Acetitomaculum是一种产乙酸菌,可以利用瘤胃中产生的氢或者单糖生成乙酸[20]。许多研究发现淀粉和精饲料诱导低乳脂模式下,反刍动物瘤胃中Acetitomaculum的丰度呈上升趋势[21-22]。本研究与这些发现一致,低乳脂率奶牛瘤胃内大部分丰度显著上升的菌群都是产酸菌,由此可见,瘤胃内产酸菌的增殖可改变瘤胃发酵模式,最终影响乳脂率。
3.3 瘤胃代谢物对乳脂率的影响瘤胃代谢组学研究表明,磷脂、氨基酸、无机离子、二羧酸、脂肪酸、碳水化合物、胆固醇酯和甘油酯等是瘤胃内的主要代谢物[4]。Zeng等[23]发现低乳脂饲粮模式下,奶牛瘤胃内23种代谢物含量高于对照组,主要包括氨基酸类、糖类、胺类等。本研究发现,相同饲粮模式下,乳脂率不同的奶牛瘤胃内差异代谢物主要为脂类及有机酸类,H组内月桂酸、琥珀酸、月桂酸乙酯含量高于L组。Sun等[24]发现提高饲粮中中短链脂肪酸与长链脂肪酸的比例,可以促进乳脂的生成,中短链脂肪酸比长链脂肪酸可更快通过肝脏,且长链脂肪酸经过肝脏外组织会导致其浓度下降,进而影响乳脂合成。月桂酸可促进乳腺上皮细胞内脂肪酸的合成,进而促进乳脂的合成。此外,本研究还发现H组瘤胃液中生物活性脂类(12, 13-二羟基十八碳烯酸)含量显著高于L组,这些脂类均与炎症反应有关,其中12, 13-二羟基十八碳烯酸是亚油酸的衍生物,患乳房炎奶牛血浆中12, 13-二羟基十八碳烯酸浓度显著低于健康奶牛[25-26]。
3.4 瘤胃微生物与代谢物关联分析本研究发现琥珀酸与Succinivibrionaceae_UCG-002和Prevotellaceae_Ga6A1_group呈显著正相关。Zeng等[23]也发现了类似的结果,奶牛瘤胃内琥珀酸与普雷沃氏菌属(Prevotella)呈正相关,且琥珀酸可参与丙酸代谢。本研究还发现月桂酸与Coprococcus_2呈显著正相关。在单胃动物中,月桂酸是一种具有抗菌功能的饲料添加剂[27];在反刍动物中,月桂酸可抑制瘤胃内原生动物及甲烷生成,也可以提高瘤胃内的氮利用率及产奶量[28]。瘤胃内粪球菌属(Coprococcus)与益生菌呈正相关,且二者丰度均与脂肪酸合成相关[29]。这表明在奶牛瘤胃内,Coprococcus_2可通过月桂酸发挥其抑菌功效。
4 结论相同饲粮条件下,高乳脂率奶牛瘤胃内乙酸/丙酸的比值显著高于低乳脂率奶牛,高乳脂率奶牛瘤胃内半纤维素降解菌的丰度较高,而低乳脂率奶牛瘤胃内纤维素降解菌及产酸菌的丰度较高,2组奶牛瘤胃内差异代谢物主要为脂类及有机酸类,相关性分析显示月桂酸、月桂酸乙酯、12, 13-二羟基十八碳烯酸均与Coprococcus_2呈显著正相关,琥珀酸与Succinivibrionaceae_UCG-002和Prevotellaceae_Ga6A1_group呈显著正相关,这些微生物的变化可引起瘤胃内脂质及氨基酸代谢通路的改变,最终影响乳脂率。
| [1] |
李鹏飞, 余烨, 鲁琼芬, 等. 奶牛瘤胃微生物及其代谢物与乳脂合成关键酶基因对乳脂率的影响[J]. 饲料研究, 2021, 44(3): 118-121. LI P F, YU Y, LU Q F, et al. Effect of rumen microorganisms, metabolites and key enzyme genes in milk fat synthesis on milk fat rate in dairy cow[J]. Feed Research, 2021, 44(3): 118-121 (in Chinese). |
| [2] |
PARODI P W. Milk fat in human nutrition[J]. Australian Journal of Dairy Technology, 2004, 59(1): 3-59. |
| [3] |
HARVATINE K J, BOISCLAIR Y R, BAUMAN D E. Recent advances in the regulation of milk fat synthesis[J]. Animal, 2009, 3(1): 40-54. DOI:10.1017/S1751731108003133 |
| [4] |
吴建民. 调控奶牛乳脂率的瘤胃微生物和代谢表征物的筛选鉴定[D]. 硕士学位论文. 大庆: 黑龙江八一农垦大学, 2020. WU J M. Screening and identification of rumen microbiome and metabolic markers regulating milk fat content in dairy cows[D]. Master's Thesis. Daqing: Heilongjiang Bayi Agricultural University, 2020. (in Chinese) |
| [5] |
JAMI E, WHITE B A, MIZRAHI I. Potential role of the bovine rumen microbiome in modulating milk composition and feed efficiency[J]. PLoS One, 2014, 9(1): e85423. DOI:10.1371/journal.pone.0085423 |
| [6] |
HASSAN F U, EBEID H M, TANG Z H, et al. A mixed phytogenic modulates the rumen bacteria composition and milk fatty acid profile of water buffaloes[J]. Frontiers in Veterinary Science, 2020, 7: 569. DOI:10.3389/fvets.2020.00569 |
| [7] |
JIANG Y, OGUNADE I M, ARRIOLA K G, et al. Effects of the dose and viability of Saccharomyces cerevisiae.2.Ruminal fermentation, performance of lactating dairy cows, and correlations between ruminal bacteria abundance and performance measures[J]. Journal of Dairy Science, 2017, 100(10): 8102-8118. DOI:10.3168/jds.2016-12371 |
| [8] |
PITTA D W, INDUGU N, VECCHIARELLI B, et al. Effect of 2-hydroxy-4-(methylthio) butanoate (HMTBa) supplementation on rumen bacterial populations in dairy cows when exposed to diets with risk for milk fat depression[J]. Journal of Dairy Science, 2020, 103(3): 2718-2730. DOI:10.3168/jds.2019-17389 |
| [9] |
DEWANCKELE L, TORAL P G, VLAEMINCK B, et al. Invited review: role of rumen biohydrogenation intermediates and rumen microbes in diet-induced milk fat depression: an update[J]. Journal of Dairy Science, 2020, 103(9): 7655-7681. DOI:10.3168/jds.2019-17662 |
| [10] |
张勤. 水牛和娟姗牛对粗饲料消化的差异比较及其差异的微生物机制研究[D]. 硕士学位论文. 南宁: 广西大学, 2016. ZHANG Q. Comparation of forage digestibility between water buffalo and jersey cow and research on ruminal microbial mechanism of the difference[D]. Master's Thesis. Nanning: Guangxi University, 2016. (in Chinese) |
| [11] |
罗治杰, 马露, 卜登攀, 等. 瘤胃发酵产物乙酸和丙酸比对奶牛生产性能及乳成分影响的荟萃分析[J]. 中国畜牧兽医, 2021, 48(5): 1613-1624. LUO Z J, MA L, BU D P, et al. Meta-analysis: how the ruminal acetic acid to propionic acid ratio affect the production and milk composition of dairy cow[J]. China Animal Husbandry & Veterinary Medicine, 2021, 48(5): 1613-1624 (in Chinese). |
| [12] |
SEYMOUR W M, CAMPBELL D R, JOHNSON Z B. Relationships between rumen volatile fatty acid concentrations and milk production in dairy cows: a literature study[J]. Animal Feed Science and Technology, 2005, 119(1/2): 155-169. |
| [13] |
李烨青. 不同采食水平对奶牛生产性能、瘤胃发酵、微生物区系及其代谢组的影响[D]. 硕士学位论文. 南京: 南京农业大学, 2019. LI Y Q. Effects of feed intake levels on performances, rumen fermentation, microflora and metabolome in cows[D]. Master's Thesis. Nanjing: Nanjing Agricultural University, 2019. (in Chinese) |
| [14] |
STERGIADIS S, CABEZA-LUNA I, MORA-ORTIZ M, et al. Unravelling the role of rumen microbial communities, genes, and activities on milk fatty acid profile using a combination of omics approaches[J]. Frontiers in Microbiology, 2021, 11: 590441. DOI:10.3389/fmicb.2020.590441 |
| [15] |
DUNCAN S H, RUSSELL W R, QUARTIERI A, et al. Wheat bran promotes enrichment within the human colonic microbiota of butyrate-producing bacteria that release ferulic acid[J]. Environmental Microbiology, 2016, 18(7): 2214-2225. DOI:10.1111/1462-2920.13158 |
| [16] |
徐晓锋, 郭婷婷, 郭成, 等. 甘露寡糖对高精料诱导的低乳脂奶牛瘤胃细菌菌群调控的研究[J]. 动物营养学报, 2019, 31(11): 5245-5255. XU X F, GUO T T, GUO C, et al. Regulation of mannan oligosaccharides on rumen bacterial flora induced by high concentrate in low milk fat dairy cows[J]. Chinese Journal of Animal Nutrition, 2019, 31(11): 5245-5255 (in Chinese). |
| [17] |
徐晓锋, 郭成. 淀粉诱导奶牛乳脂下降后奶牛瘤胃细菌菌群变化[J]. 浙江农业学报, 2019, 31(10): 1591-1598. XU X F, GUO C. Changes of rumen bacterial flora after starch induced milk fat depression in dairy cows[J]. Acta Agriculturae Zhejiangensis, 2019, 31(10): 1591-1598 (in Chinese). DOI:10.3969/j.issn.1004-1524.2019.10.03 |
| [18] |
LI H Z, LIU F, LU J J, et al. Probiotic mixture of Lactobacillus plantarum strains improves lipid metabolism and gut microbiota structure in high fat diet-fed mice[J]. Frontiers in Microbiology, 2020, 11: 512. DOI:10.3389/fmicb.2020.00512 |
| [19] |
ZHENG L X, WU S R, SHEN J, et al. High rumen degradable starch decreased goat milk fat via trans-10, cis-12 conjugated linoleic acid-mediated downregulation of lipogenesis genes, particularly, INSIG1[J]. Journal of Animal Science and Biotechnology, 2020, 11: 30. DOI:10.1186/s40104-020-00436-3 |
| [20] |
SUN Z Q, YU Z, WANG B. Perilla frutescens leaf alters the rumen microbial community of lactating dairy cows[J]. Microorganisms, 2019, 7(11): 562. DOI:10.3390/microorganisms7110562 |
| [21] |
ZENED A, COMBES S, CAUQUIL L, et al. Microbial ecology of the rumen evaluated by 454 GS FLX pyrosequencing is affected by starch and oil supplementation of diets[J]. FEMS Microbiology Ecology, 2013, 83(2): 504-514. DOI:10.1111/1574-6941.12011 |
| [22] |
MAO S Y, HUO W J, ZHU W Y. Microbiome-metabolome analysis reveals unhealthy alterations in the composition and metabolism of ruminal microbiota with increasing dietary grain in a goat model[J]. Environmental Microbiology, 2016, 18(2): 525-541. DOI:10.1111/1462-2920.12724 |
| [23] |
ZENG H B, GUO C Z, SUN D M, et al. The ruminal microbiome and metabolome alterations associated with diet-induced milk fat depression in dairy cows[J]. Metabolites, 2019, 9(7): 154. DOI:10.3390/metabo9070154 |
| [24] |
SUN Y, BU D P, WANG J Q, et al. Supplementing different ratios of short- and medium-chain fatty acids to long-chain fatty acids in dairy cows: changes of milk fat production and milk fatty acids composition[J]. Journal of Dairy Science, 2013, 96(4): 2366-2373. DOI:10.3168/jds.2012-5356 |
| [25] |
RODRÍGUEZ J P, GUIJAS C, ASTUDILLO A M, et al. Sequestration of 9-hydroxystearic acid in FAHFA (fatty acid esters of hydroxy fatty acids) as a protective mechanism for colon carcinoma cells to avoid apoptotic cell death[J]. Cancers, 2019, 11(4): 524. DOI:10.3390/cancers11040524 |
| [26] |
MAVANGIRA V, GANDY J C, ZHANG C, et al. Polyunsaturated fatty acids influence differential biosynthesis of oxylipids and other lipid mediators during bovine coliform mastitis[J]. Journal of Dairy Science, 2015, 98(9): 6202-6215. DOI:10.3168/jds.2015-9570 |
| [27] |
LÓPEZ-COLOM P, CASTILLEJOS L, RODRÍGUEZ-SORRENTO A, et al. Efficacy of medium-chain fatty acid salts distilled from coconut oil against two enteric pathogen challenges in weanling piglets[J]. Journal of Animal Science and Biotechnology, 2019, 10: 89. DOI:10.1186/s40104-019-0393-y |
| [28] |
BIONAZ M, VARGAS-BELLO-PÉREZ E, BUSATO S. Advances in fatty acids nutrition in dairy cows: from gut to cells and effects on performance[J]. Journal of Animal Science and Biotechnology, 2020, 11(1): 110. |
| [29] |
刘凯珍, 张养东, 黄国欣, 等. 瘤胃微生物调控牛奶品质的研究进展[J]. 饲料工业, 2021, 42(1): 32-37. LIU K Z, ZHANG Y D, HUANG G X, et al. Research progress of rumen microbiota regulating milk quality[J]. Feed Industry, 2021, 42(1): 32-37 (in Chinese). |




