2. 空军军医大学唐都医院, 西安 710038
2. Tangdu Hospital, Air Force Medical University, Xi'an 710038, China
肠毒性大肠杆菌(enterotoxigenic Escherichia coli,ETEC)是导致仔猪腹泻的常见病原微生物之一,约50%的仔猪死亡与ETEC感染有关[1]。2016年我国新生活仔猪8.79亿头,其中1.53亿头因腹泻等疾病在断奶前后死亡,直接导致经济损失高达240亿元[2]。此外,ETEC也是导致发展中国家儿童腹泻和旅行者腹泻最常见的病原体之一[3-5],而全世界有10%~60%的腹泻患者由ETEC感染导致,严重威胁着人体健康[6-7]。
生物被膜(biofilm,BF)是细菌生长过程中的天然保护机制[8],通过增加病原微生物对肠道黏膜表面的黏附从而增强耐受力[9-11]。ETEC感染机体肠道的第1步是在定植因子的介导下黏附于小肠上皮细胞上,而生物被膜无疑大大增强了ETEC的致病性[12-13]。此外,大肠杆菌致病性还受宿主肠道微环境中信号分子的影响[14]。其中,N-乙酰乙醇胺(N-acylethanolamine)和磷脂酰乙醇胺(phosphatidylethanolamine)作为哺乳动物小肠上皮细胞膜和细菌细胞膜的主要成分,可在体内分解为乙醇胺(ethanolamine,EA)而被肠道微生物直接利用。前人研究发现,肠内乙醇胺浓度约为4 mmol/L[15],乙醇胺不仅能够调节出血性大肠杆菌O157 ∶ H7编码志贺毒素和毒力相关基因的表达量[14],也会对鼠伤寒沙门氏菌在宿主肠道内的适应性与毒力表达起到促进作用[16]。而病原菌eut操纵子在其中发挥了重要作用,该操纵子编码了乙醇胺受体eutR基因和参与乙醇胺代谢的eutB等10余个基因[17-20]。
为了研究乙醇胺对ETEC生物被膜的调控作用,本研究首先使用Red重组系统(λ噬菌体γ、β和exo同源重组系统)对eutR和eutB基因进行敲除,然后在体外模拟肠内缺氧条件下,利用乙醇胺对野生型ETEC(WT)和eutR、eutB基因敲除株(△eutR和△eutB)进行干预,通过对ETEC生长情况、生物被膜生成量进行测定,探讨了肠内乙醇胺对ETEC致病表型的可能影响。本研究揭示了乙醇胺影响ETEC生物被膜生成量的一般规律,为预防和控制ETEC对仔猪肠道损伤提供理论依据,也为缓解畜禽腹泻问题提供新的可能突破口。
1 材料与方法 1.1 主要菌株、质粒与试剂本试验所用菌株和质粒信息见表 1[21]。pfu DNA聚合酶、Taq聚合酶、dNTP、切胶纯化试剂盒等分子生物学试剂购自生工生物工程(上海)股份有限公司(以下简称生工)。乙醇胺、L-阿拉伯糖、异丙基-β-D-硫代半乳糖苷(IPTG)、氯霉素(Cm)和卡那霉素(Kn)购自西格玛奥德里奇有限公司(Sigma-Aldrich)。
|
|
表 1 菌株和质粒信息 Table 1 Strains and plasmids information |
伯乐Western blotting电泳转印系统(Bio-Rad Mini-Protean Tetra System)、厌氧培养系统(Gene Science A30)、伯乐电穿孔(Bio-Rad Gene Pluser Xcell)和生化培养箱(Heal Force)、Varioskan LUX多功能酶标仪(Thermo)。
1.3 菌株的培养菌株培养参见文献[22]。试验条件下利用无氧培养系统对ETEC进行培养,其中厌氧气体成分为:80.0%氮气、9.6%二氧化碳及10.4%氢气,重复置换3次后放置在37 ℃生化培养箱内培养。
1.4 打靶片段的设计与制备 1.4.1 eutB基因敲除打靶片段的设计与制备根据ETEC H10407菌株的eutB基因及上、下游序列进行引物设计,通过PCR反应获得外侧50 bp序列为eutB基因的同源重组臂,内侧20~30 bp序列为卡那霉素抗性基因表达框。引物由生工合成。eutB-F、eutB-R引物用于扩增eutB基因两翼的同源序列中间为卡那霉素抗性基因的打靶DNA片段。引物信息参见表 2,其中大写序列为ATCC35401菌株eutB基因的同源重组臂;小写序列为卡那霉素抗性基因的特异引物序列。
|
|
表 2 PCR引物信息 Table 2 PCR primer information |
以pET28a质粒为模板,eutB-F、eutB-R为扩增引物,利用pfu DNA酶进行PCR扩增。反应体系如下:pET28a质粒(10 ng/μL)0.5 μL、10×pfu Buffer 5 μL、dNTP(2.5 mmol/L)4 μL、eutB-F(50 pmol/μL)0.5 μL、eutB-R(50 pmol/μL)0.5 μL、pfu DNA polymerase(MBI, 5 U/μL)0.5 μL,加dH2O至50 μL。PCR扩增条件:首先95 ℃ 5 min;随后95 ℃变性30 s,55 ℃退火(每循环降低0.5 ℃)45 s,72 ℃延伸90 s,重复30个循环;最后72 ℃延伸7 min,4 ℃反应终止。利用生工胶纯化试剂盒进行纯化回收,去除非特异性片段和PCR反应组分并调整浓度为20 ng/μL。
1.4.2 eutR基因敲除打靶片段的设计与制备根据ETEC H10407菌株的eutR基因及上、下游序列进行引物设计,通过PCR反应获得外侧50 bp序列为eutR基因的靶向同源重组臂,内侧20~30 bp序列为氯霉素抗性基因表达框。引物由生工合成。eutR-F、eutR-R引物用于扩增eutR基因两翼的同源序列中间为卡那霉素抗性基因的打靶DNA片段。引物信息参见表 2,其中大写序列为ATCC35401菌株eutR基因的同源重组臂;小写序列为氯霉素抗性基因的特异引物序列。
以pKD3质粒为模板,eutR-F、eutR-R为扩增引物,利用pfu DNA酶扩增。pKD3质粒(10 ng/μL)0.5 μL、10×pfu DNA Buffer 5 μL、dNTP(2.5 mmol/L)4 μL、eutR-F(50 pmol/μL)0.5 μL、eutR-R(50 pmol/μL)0.5 μL、pfu DNA polymerase(MBI, 5 U/μL)0.5 μL,加dH2O至50 μL。PCR扩增条件:同1.4.1。利用生工胶纯化试剂盒进行纯化回收,去除非特异性片段和PCR反应组分并调整浓度为20 ng/μL。
1.5 ETEC H10407菌株感受态细胞的制备及eutB和eutR基因打靶 1.5.1 ETEC H10407菌株感受态细胞的制备及eutB基因打靶首先制备ETEC H10407菌株的电转化感受态细胞,然后将pKD46质粒电转化进入ETEC H10407感受态细胞,将其涂布在含有氨苄青霉素的LB琼脂平板上,30 ℃培养过夜培养。接着挑选H10407/pKD46单克隆,制备成电转化感受态细胞。最后将eutB打靶片段转化进入H10407/pKD46感受态细胞,随后涂布添加有卡那霉素的LB琼脂平板上,37 ℃培养过夜。在上述含有抗生素的LB平板上随机挑取正常生长的淡黄色菌落,各接种至3 mL LB(含50 μg/mL卡那霉素),37 ℃培养过夜。用基因组上同源手臂外侧的1对引物进行PCR检测。引物序列eutB-outF和eutB-outR见表 2;反应体系:菌液0.5 μL、10×Taq Buffer 5 μL、dNTP(2.5 mmol/L)4 μL、eutB-outF(50 pmol/μL)0.5 μL、eutB-outR(50 pmol/μL) 0.5 μL、Taq DNA polymerase(5 U/μL) 0.5 μL、dH2O 39 μL,总共50 μL;扩增条件:95 ℃ 5 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,进行35个循环;最后72 ℃延伸7 min。
1.5.2 ETEC H10407菌株感受态细胞的制备及eutR基因打靶方法同上(1.5.1),最后将eutR打靶片段转化进入H10407/pKD46感受态细胞。转化细胞涂布在含有卡那霉素的LB平板上,37 ℃培养过夜培养。利用eutB-outF/CmSeqR2和CmSeqF2/eutB-outR(具体序列见表 2)引物组进行PCR验证,反应体系:菌液0.5 μL、10×Taq Buffer 5 μL、dNTP (2.5 mmol/L)4 μL、eutR-outF/CmSeqR2 (50 pmol/μL) 0.5 μL、CmSeqF2/eutR-outR (50 pmol/μL)0.5 μL、Taq DNA polymerase(5 U/μL) 0.5 μL、dH2O 39 μL,总共50 μL;扩增条件:95 ℃ 5 min;9 5℃ 30 s,60 ℃ 30 s,72 ℃ 45 s,进行35个循环;最后72 ℃延伸7 min。
1.6 WT和△eutR和△eutB生长曲线的测定为了在体外模拟宿主肠道乙醇胺浓度,本研究选用4 mmol/L作为试验浓度,将活化培养好的WT和△eutR和△eutB按照1 ∶ 100的比例接种至含有乙醇胺或者不含有乙醇胺的LB肉汤培养基中,用Varioskan LUX多测定功能酶标仪测定生长曲线,孵育温度为37 ℃(在此温度下乙醇胺稳定),共测定12 h。
1.7 ETEC的eutR和eutB基因敲除后生物被膜生成量的测定分别挑取WT、ΔeutR和ΔeutB单菌落,至3 mL的LB肉汤培养基中,37 ℃摇床过夜培养。次日将WT、ΔeutR和ΔeutB菌液以1 ∶ 100的比例接种至含有不同浓度乙醇胺的新鲜LB肉汤培养基的样品瓶中,在37 ℃摇床上,160 r/min有氧培养12 h。接着取出样品瓶,倒掉菌液,用磷酸盐缓冲液(PBS)洗去残留菌体,放置晾干。向样品瓶中加入0.5 mL 2.5%的结晶紫溶液,染色1 min,然后用无菌水洗去染液,放置晾干。最后向样品瓶中加入1 mL 33%的冰乙酸,在吸光度(OD)570 nm处测定。
1.8 实时荧光定量PCR(real-time PCR)利用实时荧光定量PCR技术对qesE基因表达水平进行相对定量,首先使用Steadypure Universal RNA提取试剂盒进行总RNA提取,并利用Evo-M-MLV Premix进行反转录,具体操作严格按照试剂盒说明书进行。PCR反应体系如下:2×SYBR Green pro Taq HS Premix 10 μL,50 pmol/μL引物各0.2 μL,模板cDNA 1 μL,dH2O补足20 μL,其中引物序列见表 2。以gyrA基因作为内参,结果采用2-ΔΔCt方法进行计算。
1.9 统计分析数据表示为“平均值±标准差”(means±SD),使用GraphPad Prism 8软件进行数据的处理分析以及图像的绘制,试验数据采用单因素方差分析(one-way ANOVA)进行差异显著性检验。P<0.05表示差异显著。
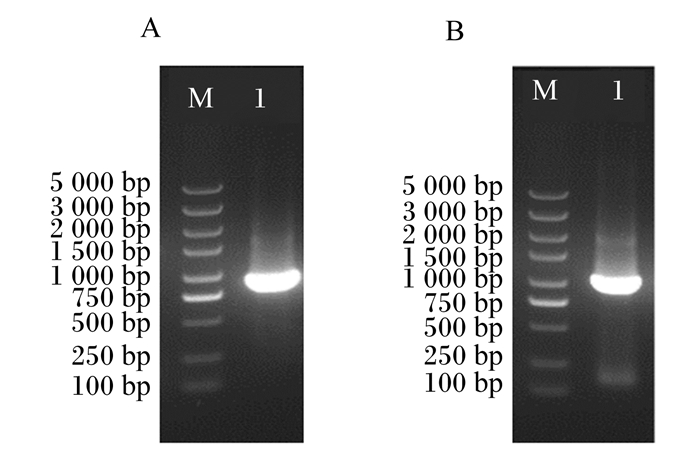
2 结果与分析 2.1 打把片段的制备与纯化利用PCR扩增出了eutB基因打靶片段,其两侧为60~80 bp的eutB基因同源重序列,内侧为卡那霉素抗性基因序列;同理扩增出eutR基因打靶片段,其内侧为氯霉素抗性基因序列。PCR扩增结果见图 1,其中eutR和eutB基因打靶片段长度分别为1 106和967 bp,片段大小与预期相符。随后使用胶回收试剂盒对目标产物进行纯化。
|
图A: 1为eutB基因打靶片段;M为DNA分子质量标准(DL 5 000);图B:1为eutR基因打靶片段;M为DNA分子质量标准(DL 5 000)。 Figure A: 1 was eutB gene targeting fragment; M was DNA marker (DL 5 000); Figure B: 1 was eutR gene targeting fragment; M was DNA marker (DL 5 000). 图 1 eutB和eutR基因的打靶PCR产物琼脂凝胶电泳 Fig. 1 Agarose gel electrophoresis of PCR products of eutB and eutR gene targeting |
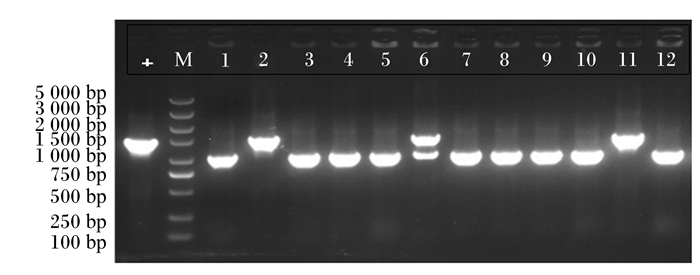
在卡那霉素平板上筛选eutB敲除成功的菌株:随机挑取了12个单菌落,同时用出发菌株(H10407)作为阴性对照,提取DNA后使用eutB-outF和eutB-outR引物进行PCR验证。出发菌株扩增产物长度为1 525 bp,而eutB基因被卡那霉素抗性基因替换后扩增产物长度应为1 045 bp。结果如图 2所示,除了第2、6、11号菌落外,其余扩增产物大小均与预期相符,可能为替换成功的阳性克隆。选择第1号菌落,在含有卡那霉素的LB平板上连续传代3次后随机挑取单菌落,接种至3 mL含卡那霉素的LB肉汤培养基,37 ℃,220 r/min培养过夜,离心后提取菌体DNA,用eutB-outF和eutB-outR引物再次进行PCR扩增,并测序验证。测序结果显示与预期相符,提示eutB基因敲除成功,该菌命名为:Ecoli10407/ΔEutB: : Kn。
|
+: 原始菌株的扩增结果。M: DNA分子质量标准(DL 5 000)。1~12:第1~12号菌落的扩增结果。 +: amplification result of the original strain; M: DNA marker (DL 5 000); 1 to 12: amplification results of clones 1 to 12. 图 2 eutB基因敲除PCR验证的琼脂糖凝胶电泳 Fig. 2 Agarose gel electrophoresis of PCR verification of eutB gene knockout |
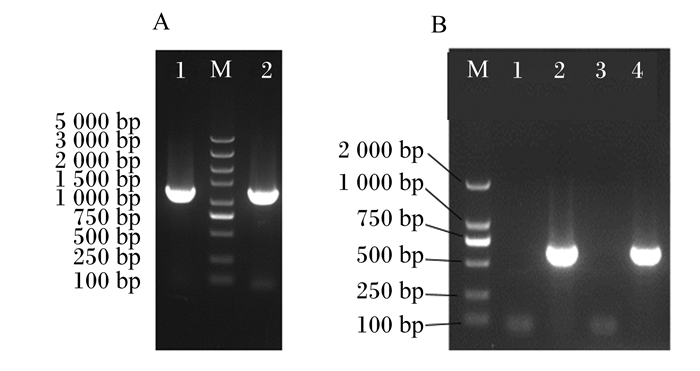
同理,诱导eutR基因敲除后在氯霉素平板筛选,结果仅有单一菌落生长。用于敲除的氯霉素抗性基因长度与目标基因长度一致,因此难以通过eutR-outF和eutR-outR扩增的PCR产物长度进行分辨(图 3-A),因此我们选择eutR-outF与CmSeqF2对氯霉素LB平板上的唯一菌落进行扩增。结果显示(图 3-B)PCR扩增为阳性,且产物长度与预计(638 bp)相符,提示此菌落可能为成功敲除菌株。为进一步验证,同理,将此菌落在氯霉素抗性平板上连续划线传代3次,用双外侧引物对菌体DNA进行PCR扩增,并将产物进行测序验证。测序结果显示与设计一致,提示eutR基因敲除成功,该菌命名为:Ecoli10407/ΔEutR: : Cm。
|
图A为利用外侧引物(eutR-outF和eutR-outR)PCR扩增敲除菌株验证结果。其中,1为阳性克隆PCR扩增结果(理论长度1 250 bp);M为DNA分子质量标准(DL 5 000);2为原始菌株外侧引物PCR扩增结果(理论长度1 267 bp)。Figure A shows the validation results of PCR amplification using external primers (eutR-outF and eutR-outR) to knock out strains. Among them, 1 was PCR amplification results of positive clone (theoretical length: 1 250 bp); M was DNA marker (DL 5 000); 2 was PCR amplification results of primers on the outer side of the original strain (theoretical length: 1 267 bp). 图B为利用交叉引物PCR验证结果。其中,M为DNA分子质量标准(DL 2 000);1为出发菌株使用eutR-outF和CmSeqF2引物进行PCR扩增结果;2为阳性克隆使用eutR-outF和CmSeqF2的PCR扩增结果;3为出发菌株的CmSeqR2和eutR-outR引物进行PCR扩增结果;4为阳性克隆使用CmSeqR2和eutR-outR引物进行PCR扩增结果。Figure B shows the PCR verification results using cross primers. Among them, M was DNA marker (DL 2 000); 1 was PCR amplification results of the starting strain used eutR-outF and CmSeqF2 primers; 2 was PCR amplification results of eutR-outF and CmSeqF2 used for positive clones; 3 was PCR amplification results of the original strain used CmSeqR2 and eutR-outR primers; 4 was PCR amplification results of positive clones used CmSeqR2 and eutR-outR primers. 图 3 eutR基因敲除PCR验证的琼脂糖凝胶电泳结果 Fig. 3 Electrophoresis analysis of PCR verification of eutR gene knockout |
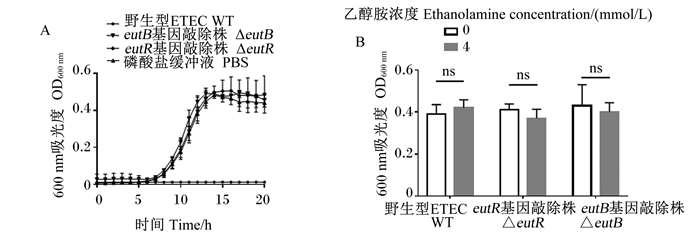
为探讨eutR和eutB基因敲除对ETEC生长繁殖的影响,我们对WT和ΔeutR和ΔeutB进行了生长曲线测定,结果如图 4-A:ΔeutR和ΔeutB在LB培养基中的生长速度与WT相比并未出现明显差异(P>0.05)。接着,使用含有4 mmol/L乙醇胺的LB培养基模拟肠内乙醇胺浓度条件下,检测了上述3株ETEC在37 ℃培养12 h后的OD600值,并以不含乙醇胺的LB培养基作为对照。结果如图 4-B所示,4 mmol/L乙醇胺并不能够改变3株ETEC在LB肉汤培养基中12 h时的生长。
|
图A为WT、ΔeutR和ΔeutB在LB培养基中的生长曲线;图B为4 mmol/L乙醇胺对WT、ΔeutR和ΔeutB在LB培养基中培养12 h时OD600值的影响。ns表示无统计学差异。 Figure A shows the growth curves of WT, ΔeutR and ΔeutB in LB medium. Figure B shows the effects of 4 mmol/L ethanolamine on OD600 value of WT, ΔeutR and ΔeutB when cultured in LB medium for 12 h. ns indicates no statistical difference. 图 4 eutR和eutB基因敲除对ETEC生长繁殖的影响 Fig. 4 Effects of eutR and eutB gene knockout on ETEC growth and reproduction |
虽然ETEC对乙醇胺的感知与代谢并不影响其在LB培养基中的生长繁殖,但为了进一步探究乙醇胺对ETEC致病能力的影响,本研究在4 mmol/L乙醇胺条件下,对WT、ΔeutR和ΔeutB生物被膜生成量进行测定,并以不含乙醇胺的LB培养基作为对照,探讨乙醇胺的识别和利用对ETEC生物被膜生成的影响。结果如图 5所示,相比对照条件,4 mmol/L乙醇胺可以显著提高WT生物被膜生成量(P < 0.05)。然而,当eutB或eutR基因敲除后,4 mmol/L乙醇胺不能够显著改变其生物被膜的生成量(P>0.05)。以上数据提示:1)肠道中乙醇胺浓度可以显著提高野生型ETEC生物被膜的体外生成;2)eutR和eutB基因在乙醇胺促进ETEC生物被膜产生的过程中起到关键作用。
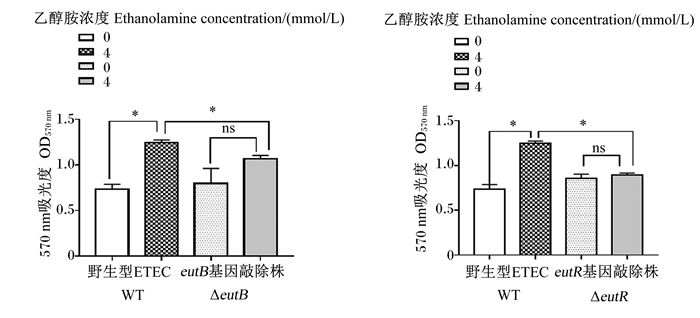
|
ns为无统计学差异;*为差异显著(P<0.05)。下同图。 ns shows no statistical difference; * is significant difference (P < 0.05). The same as below. 图 5 乙醇胺对ΔeutB(A)和ΔeutR(B)生物被膜生成的影响 Fig. 5 Effects of EA on biofilms growth of ΔeutR (A) and ΔeutB (B) |
接下来,为了进一步探讨乙醇胺识别和利用通过何种途径影响了ETEC生物被膜的生成,本研究测定了乙醇胺对生物被膜调控基因qesE表达量的影响,结果如图 6所示,4 mmol/L乙醇胺可以显著提高野生型ETEC的qesE基因表达量(P < 0.05),但无法对ΔeutR和ΔeutB敲除qesE基因表达量产生影响。这一结果提示,环境中乙醇胺对ETEC生物被膜生成量的影响,或许是通过提高qesE基因表达量导致。
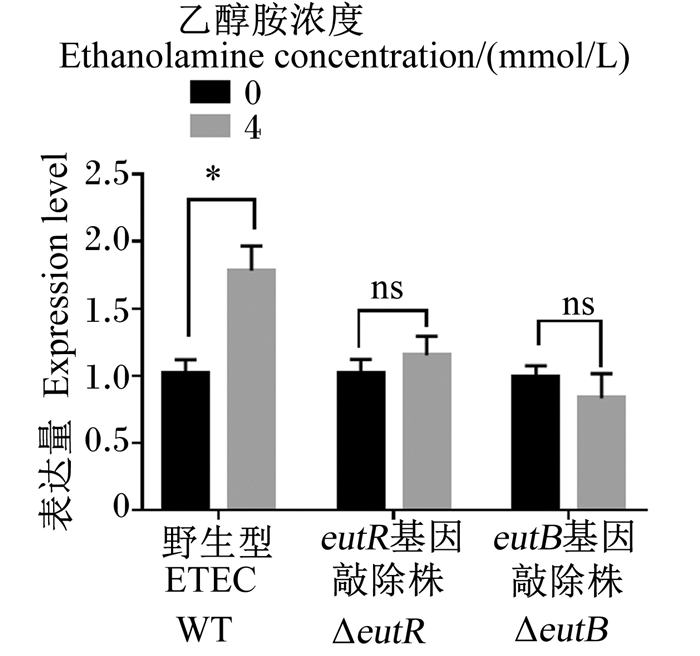
|
图 6 乙醇胺对WT、ΔeutB和ΔeutR qesE基因表达量的影响 Fig. 6 Effects of EA on expression level of qesE gene in WT, ΔeutB andΔeutR |
研究表明,宿主肠道中的营养物质、细胞代谢产物等可以作为信号分子影响肠道病原菌的毒力表达[15, 23]。乙醇胺作为宿主肠道上皮细胞膜和肠道微生物细胞膜的主要成分,对肠道病原菌毒力表达已有相关报道,例如:食源性鼠伤寒沙门氏菌会识别肠道环境中的乙醇胺促进对宿主环境的适应和定植,且eutR基因可以促进乙醇胺的代谢、影响病沙门氏菌毒力表达和传播,而eutB基因在乙醇胺信号传导、影响鼠伤寒沙门氏菌毒力表达中也起到重要作用[14, 16, 24]。此外,在对出血性大肠杆菌(EHEC)H7 ∶ O157的研究中发现:EHEC将乙醇胺作为信号分子在多种环境因素共同作用下激活EHEC的毒力表达。以上研究提示,乙醇胺对肠道致病菌的毒力表达起到关键作用,然而,乙醇胺是如何影响ETEC毒力表型尚不明确。
为了探究乙醇胺对ETEC毒力表型的影响,本研究首先利用λ噬菌体Red重组系统对ETEC编码乙醇胺受体和乙醇胺代谢关键基因eutR和eutB分别敲除成功。该技术在大肠杆菌基因敲除中具有较高的敲除效率[25]。接下来,对生长情况进行测定。结果发现,敲除株与突变株的生长情况并无显著差异,造成这一现象的原因可能是乙醇胺识别和利用功能的缺失,并不会影响ETEC对培养基中其他碳源或者氮源的利用。
环境中的乙醇胺并不会影响ETEC在LB培养基中的生长和繁殖。根据这一结果我们推测:1)4 mmol/L乙醇胺对ETEC致病性的影响可能并非通过促进其生长繁殖的方式来体现;2)从ETEC的营养利用角度看,可能在LB肉汤培养基中的乙醇胺作为碳源或氮源与其他营养物质并无显著区别。可是在后续研究中,可以通过将乙醇胺作为唯一碳源或唯一氮源,在不同浓度条件下进一步深入研究乙醇胺作为营养物质对ETEC生长繁殖的影响。
虽然肠道中乙醇胺浓度并不能显著影响ETEC的生长繁殖,但在接下来的研究中发现,这一浓度的乙醇胺可以显著提高WT生物被膜的生成。对于△eutR和△eutB,由于它们无法识别和利用环境中的乙醇胺,因此不论乙醇胺是否存在,均不会影响敲除株的生物被膜生成量。这一数据提示:1)肠道浓度的乙醇胺可能影响ETEC生物被膜的生成;2)编码乙醇胺受体的eutR基因和编码乙醇胺代谢的关键基因eutB在这一过程中起到了关键作用。众所周知,生物被膜可以增强细菌的抵抗力,从而保护自身免受宿主免疫系统的攻击,而ETEC进入宿主肠道后,首先会在定植因子的介导下黏附在小肠上皮细胞上,然后进行生长繁殖、表达毒力,其生物被膜的产生会进一步增强ETEC对宿主的抵抗力从而增强其对宿主的损伤作用。综合以上数据,由于△eutB的基因组上依然编码有乙醇胺受体基因eutR,因此,△eutB依然能够感知环境中乙醇胺的存在。而eutB敲除却能够抑制乙醇胺对ETEC生物被膜生成的促进作用。这一数据提示,乙醇胺对ETEC对生物被膜生成的影响,可能受到了乙醇胺代谢或其代谢物的影响,而不仅仅是ETEC对乙醇胺信号分子的识别。
虽然,本研究并未揭示乙醇胺促进ETEC生物被膜生成的确切机制,例如:这一促进作用是通过ETEC对乙醇胺代谢后产生的何种物质起到关键作用,但是本研究发现qesE基因在4 mmol/L乙醇胺处理条件下表达量发生显著上调。前人研究也发现,该基因参与生物被膜的生成过程,但乙醇胺对ETEC生物被膜的促进作用是否由qesE基因导致,仍需要进一步研究。另外,本研究也并未对乙醇胺影响ETEC其他毒力表型进行研究,例如,对LT毒素表达、黏附作用以及耐药表型进行研究。但本研究数据足以说明,肠道中乙醇胺浓度在体外厌氧条件下可以提高ETEC生物被膜的生成,应当引起足够重视。最后,通过阻断对乙醇胺的利用,或许能够成为缓解ETEC感染造成仔猪死亡率的靶点之一。
4 结论本研究利用λ噬菌体Red同源重组系统,成功敲除了ETEC基因组中参与乙醇胺识别和利用的eutR和eutB基因。研究发现,eutR和eutB敲除并不会影响ETEC在LB培养基中的正常生长,但4 mmol/L的EA可以显著促进ETEC的生物被膜的生成量,且这一作用可能与eutR和eutB基因密切相关。
| [1] |
刘玉芹, 闫平, 隗金玲, 等. 仔猪源肠毒素性大肠杆菌毒力因子的分布研究[J]. 黑龙江畜牧兽医, 2018(5): 139-142. LIU Y Q, YAN P, KUI J L, et al. Distribution of virulence factors of enterotoxigenic Escherichia coli from piglets[J]. Heilongjiang Animal Science and Veterinary Medicine, 2018(5): 139-142 (in Chinese). |
| [2] |
杨天宁, 刘伟, 涂攀, 等. 产肠毒素大肠杆菌病商用疫苗的研究进展[J]. 猪业科学, 2021, 38(3): 54-55. YANG T N, LIU W, TU P, et al. Progress of commercial vaccines for enterotoxigenic Escherichia coli[J]. Swine Industry Science, 2021, 38(3): 54-55 (in Chinese). DOI:10.3969/j.issn.1673-5358.2021.03.016 |
| [3] |
QADRI F, SVENNERHOLM A M, FARUQUE A S G, et al. Enterotoxigenic Escherichia coli in developing countries: epidemiology, microbiology, clinical features, treatment, and prevention[J]. Clinical Microbiology Reviews, 2005, 18(3): 465-483. DOI:10.1128/CMR.18.3.465-483.2005 |
| [4] |
BOUCKENOOGHE A R, JIANG Z D, DE LA CABADA F J, et al. Enterotoxigenic Escherichia coli as cause of diarrhea among Mexican adults and US travelers in Mexico[J]. Journal of Travel Medicine, 2002, 9(3): 137-140. |
| [5] |
DALTON C B, MINTZ E D, WELLS J G, et al. Outbreaks of enterotoxigenic Escherichia coli infection in American adults: a clinical and epidemiologic profile[J]. Epidemiology and Infection, 1999, 123(1): 9-16. DOI:10.1017/S0950268899002526 |
| [6] |
GASCÓN J, VARGAS M, QUINTÓ L, et al. Enteroaggregative Escherichia coli strains as a cause of traveler's diarrhea: a case-control study[J]. The Journal of Infectious Diseases, 1998, 177(5): 1409-1412. DOI:10.1086/517826 |
| [7] |
BLACK R E. Epidemiology of travelers' diarrhea and relative importance of various pathogens[J]. Reviews of Infectious Diseases, 1990, 12(Suppl.1): S73-S73. |
| [8] |
李婷婷, 国竞文, 励建荣, 等. 细菌生物被膜的研究进展及与群体感应的关系[J]. 中国渔业质量与标准, 2017, 7(1): 1-7. LI T T, GUO J W, LI J R, et al. Research progress of bacterial biofilm and its relationship with quorum sensing[J]. China Fishery Quality and Standards, 2017, 7(1): 1-7 (in Chinese). |
| [9] |
马伊萨兰, 张荣, 王洪志, 等. 食品中金黄色葡萄球菌生物被膜形成基因分析及影响因素研究[J]. 食品工业科技, 2017, 38(15): 129-133, 139. MA Y S L, ZHANG R, WANG H Z, et al. Analysis of biofilm formation related genes and its influence factors for Staphylococcus aures food isolates[J]. Science and Technology of Food Industry, 2017, 38(15): 129-133, 139 (in Chinese). |
| [10] |
王娜. 嗜水气单胞菌浮游态和生物被膜状态比较蛋白质组学及相关蛋白特性分析[D]. 博士学位论文. 南京: 南京农业大学, 2012. WANG N. Comparative proteomic analysis between biofilm and planktonic cells of Aeromonas hydrophila and characteristics of related proteins[D]. Ph. D. Thesis. Nanjing: Nanjing Agricultural University, 2012. (in Chinese) |
| [11] |
DOSLER S, KARAASLAN E. Inhibition and destruction of Pseudomonas aeruginosa biofilms by antibiotics and antimicrobial peptides[J]. Peptides, 2014, 62: 32-37. DOI:10.1016/j.peptides.2014.09.021 |
| [12] |
NAGY B, FEKETE P Z. Enterotoxigenic Escherichia coli in veterinary medicine[J]. International Journal of Medical Microbiology, 2005, 295(6/7): 443-454. |
| [13] |
GAASTRA W, SVENNERHOLM A M. Colonization factors of human enterotoxigenic Escherichia coli (ETEC)[J]. Trends in Microbiology, 1996, 4(11): 444-452. DOI:10.1016/0966-842X(96)10068-8 |
| [14] |
KENDALL M M, GRUBER C C, PARKER C T, et al. Ethanolamine controls expression of genes encoding components involved in interkingdom signaling and virulence in enterohemorrhagic Escherichia coli O157:H7[J]. mBio, 2012, 3(3): e00050-12. |
| [15] |
GONYAR L A, KENDALL M M. Ethanolamine and choline promote expression of putative and characterized fimbriae in enterohemorrhagic Escherichia coli O157 ∶ H7[J]. Infection and Immunity, 2014, 82(1): 193-201. DOI:10.1128/IAI.00980-13 |
| [16] |
ANDERSON C J, CLARK D E, ADLI M, et al. Ethanolamine signaling promotes Salmonella niche recognition and adaptation during infection[J]. PLoS Pathogens, 2015, 11(11): e1005278. DOI:10.1371/journal.ppat.1005278 |
| [17] |
THIENNIMITR P, WINTER S E, WINTER M G, et al. Intestinal inflammation allows Salmonella to use ethanolamine to compete with the microbiota[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(42): 17480-17485. DOI:10.1073/pnas.1107857108 |
| [18] |
KOFOID E, RAPPLEYE C, STOJILJKOVIC I, et al. The 17-gene ethanolamine (eut) operon of Salmonella typhimurium encodes five homologues of carboxysome shell proteins[J]. Journal of Bacteriology, 1999, 181(17): 5317-5329. DOI:10.1128/JB.181.17.5317-5329.1999 |
| [19] |
STOJILJKOVIC I, BÄUMLER A J, HEFFRON F. Ethanolamine utilization in Salmonella typhimurium: nucleotide sequence, protein expression, and mutational analysis of the cchA cchB eutE eutJ eutG eutH gene cluster[J]. Journal of Bacteriology, 1995, 177(5): 1357-1366. |
| [20] |
王洪彬, 朱利霞, 于秀剑, 等. 细菌生物被膜研究进展[J]. 动物医学进展, 2019, 40(9): 74-79. WANG H B, ZHU L X, YU X J, et al. Progress on bacterial biofilm[J]. Progress in Veterinary Medicine, 2019, 40(9): 74-79 (in Chinese). |
| [21] |
DATSENKO K A, WANNER B L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12): 6640-6645. |
| [22] |
鲁曦, 谢永宏, 孙瑞琳, 等. 缺氧条件下肠毒性大肠杆菌hns和stpA基因敲除以及对LT毒素表达调控的研究[J]. 基因组学与应用生物学, 2017, 36(12): 5128-5134. LU X, XIE Y H, SUN R L, et al. Study on hns and stpA gene knockout of enterotoxigenic Escherichia coli and the regulation on LT toxin expression under anaerobic condition[J]. Genomics and Applied Biology, 2017, 36(12): 5128-5134 (in Chinese). |
| [23] |
BAKOVIC M, FULLERTON M D, MICHEL V. Metabolic and molecular aspects of ethanolamine phospholipid biosynthesis: the role of CTP: phosphoethanolamine cytidylyltransferase (Pcyt2)[J]. Biochemistry and Cell Biology, 2017, 85(3): 283-300. |
| [24] |
LUZADER D H, CLARK D E, GONYAR L A, et al. EutR is a direct regulator of genes that contribute to metabolism and virulence in enterohemorrhagic Escherichia coli O157 ∶ H7[J]. Journal of Bacteriology, 2013, 195(21): 4947-4953. |
| [25] |
胡堃, 史兆兴, 赛道建, 等. Red重组系统及在微生物基因敲除中的应用[J]. 遗传, 2003, 25(5): 628-632. HU K, SHI Z X, SAI D J, et al. The Red recombination system and its application to gene knock-out in microorganism[J]. Hereditas, 2003, 25(5): 628-632 (in Chinese). |




