由于抗生素的滥用,使得许多细菌进化为耐药性强的菌株,而抗菌肽(antimicrobial peptides, AMPs)凭借其较难产生耐药性的特点,成为传统抗生素最有潜力的替代品。AMPs多是强阳离子性的小分子多肽,通过结合在带有负电性的细菌细胞膜表面,促使膜破碎致死,而细菌则可以通过修饰细菌表面分子、分泌保护性物质、上调或消除特殊蛋白质以及形成生物膜进而产生对AMPs的抗性[1]。由此,根据耐药性机制,可以将细菌分为单细菌成因的浮游菌和多细菌成因的生物膜2类。近年来AMPs耐药性研究愈发增多,而大量使用或滥用AMPs势必会引起同抗生素一样的严重后果。加深AMPs耐药性研究,有助于设计出更有成效的AMPs,防止AMPs耐药性的产生。本文将着重阐述细菌对AMPs的抗性及机理。
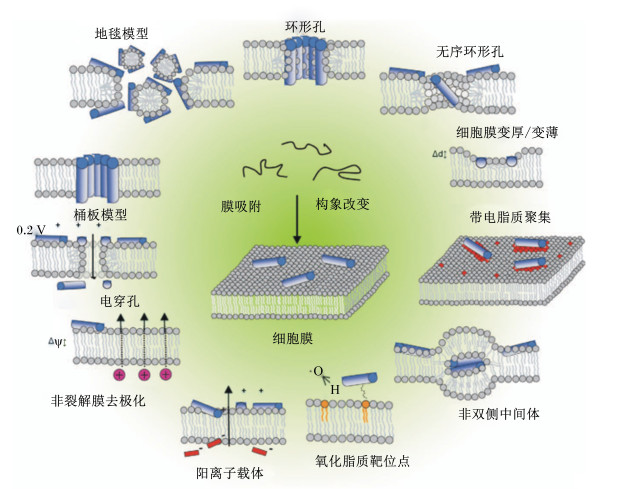
1 浮游菌对AMPs的抗性两亲性结构特点是AMPs抗菌功能的关键,而细菌的细胞膜表面是AMPs最主要的作用位点(图 1),浮游菌正是通过细胞质膜和细胞壁的一系列变化,从而达到抵抗AMPs的目的。细胞质膜是保护细胞内容物的物理屏障,而细胞壁给予细菌结构支持确保大多数细菌的存活。根据细胞壁结构不同可以将细菌分为革兰氏阳性菌和革兰氏阴性菌,其中革兰氏阳性菌的细胞壁包括1个较厚的肽聚糖层和其他糖类,主要由磷壁酸(lipoteichoic acid,LTA)和壁醛酸合成;而革兰氏阴性菌的细胞壁则更为复杂,包括较薄的肽聚糖层、脂蛋白、脂多糖和1个外膜[2]。对于AMPs的抗性也多是来自以上结构的修饰。
|
图 1 抗菌肽作用机理 Figure 1 The action mechanism of AMPs[3] |
胞外多糖是由革兰氏阳/阴性菌合成并分泌的一种多糖聚合物,多呈阴离子性,当共价连接到细胞壁时则被称为荚膜多糖。革兰氏阳性菌胞外多糖大多附着于肽聚糖层,其可以与AMPs结合,阻碍其进一步发挥作用[4]。
革兰氏阴性菌,如克雷白氏菌(K. pneumoniae)、肺炎链球菌(Streptococcus pneumoniae)和铜绿假单胞菌(P. aeruginosa),更倾向于释放胞外多糖到细胞外,以便于诱捕AMPs,而这种分泌胞外多糖的功能有助于减少细菌表面的AMPs数量。此外,有研究表明诱导K. pneumoniae的荚膜多糖变异,会使其AMPs更易感,并且更容易与荚膜多糖结合[5-6]。
胞外蛋白则通过诱捕和降解AMPs使细菌产生对AMPs的抗性。细菌用不同种蛋白酶如金属、丝氨酸、半胱氨酸和天冬氨酸蛋白酶,这些蛋白酶分泌出亦或是固定在细菌表面,用来降解AMPs并保护其靶位点[7]。停乳链球菌类马亚型(Streptococcus dysgalactiae subsp. equisimilisa)可以合成与链球菌补体抑制剂(streptococcal inhibitor of complement, SIC)和链球菌补体抑制剂远亲(distantly related to SIC, DRS)同源的蛋白DrsG,其可以被分泌到细胞外并与LL-37(一种AMPs)结合,从而抑制其活性[8]。
1.2 细胞膜修饰细菌细胞膜包含几种磷脂,如磷脂酰乙醇胺(phosphatidylethanolamine,PE)、磷脂酰甘油(phosphatidyglycerol,PG)和心磷脂(cardiolipin,CL)[9]。调节细胞膜的组分不仅可以改变膜表面电性,还可以改变膜的流动性与刚性。由此可见,细菌细胞膜组分的调节是细菌存活的关键,也是对AMPs抗性的要点。
可将细胞膜分成2种模型:1) 只包含10%CL的膜,该膜可以在AMPs的作用下有效防止膜移位和小孔的形成。对青霉素具有抗性的金黄色葡萄球菌(S. aureus)菌株利用存在于膜内的适应因子,通过多种抗性因子(multiple peptide resistancefactor,mrpF)、心磷脂合酶(cardiolipin synthase,cls)、磷脂酰甘油合酶(phosphatidylglycerolsynthase,pgsA)并在其他机制协同作用下修饰膜表面使其呈现正电性,对AMPs产生抗性[10]。2) 存在氨酰磷脂[赖氨酸(Lys)-PE、谷氨酰胺(Gln)-PE]的膜,这是一种常见的发病菌修饰脂质双分子层从而抵抗AMPs的方式[11]。
可将脂质修饰划分成2类:1) PG修饰。在革兰氏阳性菌中,基因编码大量的膜蛋白mrpF,为了增加赖氨酸和丙氨酸的含量,使PG去合成赖氨酰磷脂酰甘油(lysyl-phosphatidylglycerol,LPG)和丙氨酰磷脂酰甘油(alanyl-phosphatidylglycerol,APG),同时传递这些物质到外部[12]。与易感菌株相比,mrpF功能性突变并且包含大量LPG的S. aureus菌株被发现对AMPs具有更高的抗性,并且其表面电荷降低[10]。2) 脂肪酸成分改变。减少支链脂肪酸含量可能增加粪肠球菌(Enterococcus faecalis)细胞膜的刚性,并伴随正电荷内容物的增加,从而阻止AMPs的穿透[13]。同样,增加S. aureus细胞膜中不饱和、支链和氢化脂肪酸的含量,可以使膜具有一个较高的流动性,可阻止AMPs的聚集[14]。
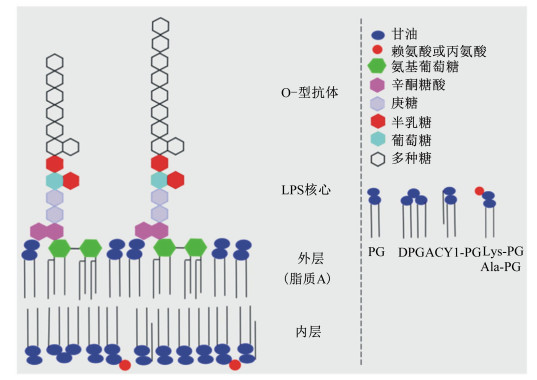
1.3 细胞壁修饰 1.3.1 革兰氏阴性菌在革兰氏阴性菌的表面,主要的修饰场所位于脂多糖(LPS)和脂低聚糖(lipooligosaccharides,LOS),LPS是由脂质A、LPS核心以及O型抗原组成的,带有负电荷(图 2)。LPS的结构不仅可以维持细胞平衡,也可以形成物理屏障以及抵御AMPs,一些LPS的修饰依赖于细菌种类以及环境因素,以此产生对AMPs的抗性,而众多修饰方式中以脂质A的修饰最为主要。
|
PG:磷脂酰甘油phosphatidylglycerol;DPG:双磷脂酰甘油bisphosphatidylglycerol;Acyl-PG:酰基磷脂酰甘油acylphosphatidylglycerol;Lys-PG:赖氨酸磷脂酰甘油lysine phosphatidylglycerol;Ala-PG:丙氨酸磷脂酰甘油alanyl phosphatidylglycerol。 图 2 革兰氏阴性菌外膜(肠道沙门氏菌) Figure 2 Gram negative bacteria outer membrane (S. enterica)[1] |
对于脂质A的修饰种类,可以分为4类:1) 增添氨基糖。氨基糖通常为氨基树胶醛糖、葡糖糖胺和半乳糖胺。如鲍氏不动杆菌(Acinetobacter baumannii)、新凶手弗朗西丝氏菌(Francisella novicida)和百日咳杆菌(Bordetella pertussis)通过添加半乳糖胺和葡糖糖胺对脂质A进行修饰。而在Bordetella pertussis的脂质A中添加葡糖糖胺则可以对多黏菌素B(polymyxin B)、黏杆菌素(colistin)、LL-37、indolicidin、HHC-10和CP28产生抗性[15]。2) 磷酸化和氢化。lpxF基因编码的一种酶可以除去拟杆菌门(Bacteroidetes)LPS的脂质A上的一个负电荷磷酸盐。这种电荷的转变使得正电荷AMPs较难结合到细菌细胞膜上并破坏它。这使得在构建细胞膜时必要的磷酸盐在细菌成熟时变为了多余物,由此产生对AMPs的抗性[16]。3) 酰化和脱酰化。在脂质A上添加酰基被认为是一种有效的对AMPs产生抗性的方式,pagP基因可编码添加酰基到脂质A上,pagP基因突变的副百日咳博德特菌(Bordetella parapertussis)将会比野生菌株对C18G更加易感,而P. aeruginosa可以对C18G产生抗性,但是对polymyxin B并不明显[17-18]。而且pagP基因可以维持膜流动性的平衡,修复被AMPs破坏的膜,这是另一种产生AMPs抗性的方式[19]。4) 增加甘氨酸。almEFG操纵子可以将甘氨酸加入到霍乱弧菌(Vibrio cholerae)的脂质A上,这可以对多黏菌素(polymyxin)产生抗性,而其明确原理尚未清楚[20]。
1.3.1.2 O型抗原当伯克霍尔德菌(Burkholderia)与豆虫肠道内的点蜂缘蝽(Riptortus pedestris)共生繁殖时,失去了LPS的O型抗原会增加宿主AMPs对其的易感程度[21]。肠炎沙门氏菌(Salmonella enterica serovar Enteritidis)中参与编译O型抗原的ΔwbaV基因突变,使得其对AMPs的易感程度提高[22]。然而,就目前的研究来看,并没有确凿的证据说明LPS的O型抗原的存在和长度一定就对AMPs抗性产生影响。
1.3.2 革兰氏阳性菌革兰氏阳性菌没有外膜的保护,所以细胞壁承受全部的外界压力以及抵御如AMPs和抗生素等有害分子的入侵。革兰氏阳性菌细胞壁中含有大量的磷壁酸,磷壁酸又可分为壁磷壁酸(wall teichoicacid,WTA)和脂磷壁酸(lipoteichoic acid,LTA)2种,前者不深入质膜,其末端以磷酸二酯键与肽聚糖的N-乙酰胞壁酸残基相连,而脂磷壁酸横跨过肽聚糖层,以其末端磷酸共价连接于质膜中糖脂的寡糖基部分。
革兰氏阳性菌中LTA的修饰被认为是普遍的对AMPs产生抗性的方式,尤其是LTA的D-丙氨酰化,而其他的LTA的修饰如加入胆碱磷酸和糖基化并未证实可对AMPs产生抗性[23]。dlt操纵子负责编码D-丙氨酸的载体蛋白连接酶,该酶可将D-丙氨酸加到磷壁酸上,这可以增加细菌表面的正电荷,提高对于AMPs的抗性[24]。此外,产单核细胞李斯特菌(Listeria monocytogenes)的WTA中含有L-鼠李糖(L-rhamnosylation),其可以通过形成空间阻碍滞后AMPs与膜之间的作用,促进对AMPs抗性的形成[25]。
2 生物膜对AMPs的抗性 2.1 生物膜的成因与组成细菌已经演化出形成多细胞结构的能力,在临床环境中,细菌被暴露在各种不利条件下,如抗菌剂、营养限制、厌氧和热休克等,这些条件可以刺激细菌的应激反应,这和其他防御机制协同可以形成生物膜,生物膜可以使细菌在恶劣的环境中得以存活。生物膜对于宿主的免疫机制和抗菌剂来说都是很难清除的,正因如此,生物膜是对AMPs产生抗性的主要因素之一[26]。从浮游状态过渡到生物膜是环境变化引起多重调节网络异常的结果,从而在一个传感压力信号作用下,浮游细胞将会接触一个表面形成生物膜,以便于有能力抵抗环境变化带来的影响[27-28]。
生物膜是由细胞外基质、特定的多糖、蛋白质、水通道和胞外DNA(eDNA)组成[29]。其中胞外聚合物(extracellular polymeric substance,EPS)作为生物膜基质,在生物膜内部的通道允许水、空气以及养分通过[30]。EPS的组分:1) 胞外多糖。胞外多糖是从胞内或胞外合成并分泌到外部环境由糖残基组成的高分子聚合物,它们呈线性或支长链连接贴在细胞表面并向外伸出形成巨大的网络,胞外多糖作为骨架使其他碳水化合物、蛋白质、核酸和脂类附着[31]。2) 胞外蛋白(extracellular proteins)。胞外蛋白是另一种主要的EPS组分,其可以附着在细胞和胞外多糖的表面,有助于生物膜的形成和稳固。3) eDNA。eDNA是由溶解细胞(lyzed cells)分泌的,是生物膜附着的关键,它的负电荷在原有的附着物里呈现斥力,但是当细菌和表面之间只有几纳米时,它将与下层表面受体相互作用促进黏附[32]。
2.2 生物膜的形成过程形成生物膜的前提是细菌与表面足够近,当细菌接近表面时具有引力与斥力,距表面10~20 nm时,细菌表面的负电荷与环境表面的负电荷相互排斥,但是这种斥力被细菌和表面之间的范德华力所克服,同时鞭毛和菌毛可以帮助细菌机械的附着于表面[33]。一旦生物膜成熟,随着菌群的发展,一些细胞从原有的固着结构分离出去,这个阶段被称为传播,它是生物膜完成循环的关键[34]。生物膜的形成可以分为3个阶段:附着、成熟和传播。
附着可以分为2个阶段,不可逆附着与可逆附着。不可逆附着的生物膜可以抵抗更强烈的物理和化学的侵袭,在可逆附着中,鞭毛和菌毛介导的机动性十分重要,鞭毛是细菌和表面最初接触的关键,而菌毛则可以使细菌聚集在一起形成菌落[35]。
成熟的生物膜由于种种原因引发生物膜的分离传播,原因可能是缺乏营养、激烈的竞争和过度生长的菌群等,传播可能发生在部分或整个生物膜上,释放浮游菌促进其在其他位点上形成新的生物膜[34]。
2.3 生物膜对AMPs的抗性机理AMPs抑制或清除生物膜的方式可以分为以下2类[36]。1) 当AMPs浓度等于或高于最小抑菌浓度(minimal inhibitory concentration,MIC)时,AMPs可以通过杀死浮游菌阻止生物膜形成,也可杀死生物膜分离的细菌,同样可以通过杀死生物膜内部细菌减少或根除生物膜。2) 当AMPs浓度低于MIC时:a.干扰生物膜附着。可通过AMPs结合在材料表面、细菌表面或EPS组分来抑制或阻止生物膜的生成;b.干扰基因表达。通过干扰修饰、下调参与基质合成的基因表达、其他基因调节异常控制生物膜生活类型,以及针对信号分子如严禁反应的ppGpp。而生物膜则通过结构障碍和诱导阻碍对AMPs产生抗性。
生物膜比浮游菌具有对抗菌剂和宿主免疫系统分泌的活性因子更强的抗性,据估计,生物膜对AMPs的抗性是浮游菌的10 000倍[29]。然而细菌对于AMPs的抗性机理并没有像其他抗菌剂那样研究的那么透彻,此外,也没有特定的细菌对于AMPs抗性的机理被发现,仅仅是一些假设的机理被提出(图 3)。生物膜对AMPs产生抗性的途径可分为以下3类。1) 结构障碍:包括胞外基质、不均质性菌群和外排泵;2) 基因控制:包括群体感应(quorum sensing,QS)、环二鸟苷酸[bis-(3’—5’)-cyclic diguanosine monophosphate,c-di-GMP]和小RNAs(small RNAs, sRNAs);3) 信号调节:包括双组分调节系统(two-component regulatory system,TCS)。其中基因控制与信号调节可视之为诱导阻碍。
2.3.1 结构障碍EPS:以P. aeruginosa为例,P. aeruginosa会合成并释放3种胞外多糖,分别命名为藻酸盐(alginate)、富葡萄糖多糖(glucose rich polysaccharide,Pel)以及戊多糖(pentasaccharide,Psl),它们可以使得生物膜更加稳固,藻酸盐可以和养分与水相互作用并且提供养分到生物膜,而Pel和Psl则起到支撑生物膜结构的功能[38]。而P. aeruginosa的eDNA则可以通过螯合阳离子对AMPs产生抗性,否则将会对生物膜的结构功能以及PhoPQ和PmrAB调节因子的诱导作用产生影响[39]。由此可见,胞外多糖多以隔离的方式抵抗AMPs,而eDNA则是通过与其电性不同的AMPs结合抵抗AMPs。
不均质性菌群:生物膜菌群包括处在不同生长时期的不均质性细菌,而这些细菌的差异将会按照氧气和养分的梯度划分,以此判定细菌应该存在于生物膜的表面还是深层。新陈代谢旺盛的细菌处于生物膜的表面,而养分与氧气被掠夺的细菌则处于生物膜的深层,它们在暴露于不同种AMPs时产生不同的严禁反应[26]。在不均质性菌群里,有一类可以产生显著抗性的细胞就是持留细胞,持留细胞在生物膜内的数量远多于浮游菌。持留细胞生长速度几近于零,AMPs对其的效果也几乎没有,因此持留细胞可以作为细菌的存储器,一旦抗菌压力消失又可以重新生长。TN-5可以有效抵抗P. aeruginosa PDO300的持留细胞,而不是其生物膜[40]。持留细胞是否能转移AMPs对于生物膜的杀伤还需进一步研究。
外排泵:外排泵对于AMPs的抗性可以分为浮游菌与生物膜2个时期,但是外排泵在生物膜中比在浮游菌中起更大作用,排外泵可以将AMPs等抗菌剂排出细菌以及生物膜。排外泵有6个家族,分别为ABC、MFS、MATE、SMR、RND和DMT[41]。其中只有ABC利用ATP作为能源将AMPs排出,其他排外泵则依靠电化学性质将AMPs排出。
2.3.2 基因控制生物膜的生成与传播由基因与环境信号所调控。现在所知道的生物膜主要的调节因子有QS、c-di-GMP和sRNAs[42]。1) QS:当细菌密度达到一定程度时,细菌会分泌出一种自产的信号小分子,称之为自诱导物(autoinducers),当自诱导物浓度达到阈值时,靶基因将会对生物膜进行调控,而且QS还可以调控eDNA、凝集素和生物表面活性剂的生成。研究发现,椰稙伯克霍尔德菌(B. cenocepacia)的QS调控大量的表面蛋白、凝集素和eDNA的表达[43]。2) c-di-GMP:作为一种第二信使调控胞外多糖、表面蛋白的合成以及生物膜流动性,有2类酶控制c-di-GMP在细菌中的含量,分别是DGGs和CGDEF[44]。P. aeruginosa的c-di-GMP通常包括5个部分,分别是环境信号感受器、涉及c-di-GMP合成与分解的酶、具体效应(由c-di-GMP调控蛋白或核糖开关变构)、靶位点(DNA、酶和分子结构)以及合成输出[45]。3) sRNAs:是非编码小分子RNA包括核糖开关,参与细菌基因转录后调控,参与一部分代谢途径、压力适应以及发病机理[46]。现在研究较为清晰的是P. aeruginosa生物膜中的基因控制,详见表 1。
|
|
表 1 P. aeruginosa生物膜中的基因控制 Table 1 Genetic control of P. aeruginosa biofilm[42] |
TCS包括组氨酸蛋白激酶(histidine kinase,HK)和应答调节蛋白(response regulator,RR)。HK是一个传感器蛋白,通常由N端的配体结合域和C端的激酶结构域组成。信号传递是通过磷酰基的转移,即一个特定的组氨酸残基从ATP转移到HK上,随后HK将磷酰基从组氨酸残基转移到RR上的天冬氨酸残基,这种磷酸激活RR的活动被视为转录调节[47]。
参与AMPs抗性的TCS可以分为4类。1) PhoPQ TCS:普遍存在于鼠伤寒沙门氏菌(S. typhimurium)中,PhoQ作为感受蛋白激酶,用于识别一些低水平的刺激如镁离子、酸性pH和AMPs(polymyxin B、C18G、LL-37和protegrin),每一个刺激都会促进不同水平PhoP调控基因的表达[48]。在S. thyphimurium中PhoPQ TCS可控制PgtE的分泌,从而对AMPs产生抗性[49]。2) PmrAB TCS:多存在于S. thyphimurium、大肠埃希氏菌(E. coli)、K. pneumoniae、鼠疫耶尔森菌(Y. pestis)、鼠类柠檬酸杆菌(C. rodentium)和P. aeruginosa中,PmrB可以感知到高浓度的铁离子、镁离子、弱酸pH和阳离子AMPs[50]。PmrAB在抵抗polymyxin B有着较好的效果,易获得黏菌素抗性菌株[51]。3) CsrRS TCS:存在于A群链球菌(group A Streptococcus)中,CraS作为感受蛋白激酶可以识别LL-37、RP-1和polymyxin B[52]。此外,CsrRS TCS还可以通过增加甘氨酸控制Vibrio cholerae的LPS修饰,CraS通过直接结合在almEFG操纵子的调节区域促进其调控[53]。4) GacSA TCS:存在于P. aeruginosa、S. thyphimurium,可促进生物膜的生成[38]。
TCS往往还存在交联对话(cross-talk),在E. coli中由PrmD介导的PhoPQ TCS和PrmAB TCS发生交联对话,但是活化PrmD不依赖PhoP/PhoQ,这个展现了TCS的复杂过程[54]。此外,TCS可以与外排泵协同作用,革兰氏阳性细菌中有一个特殊的结构称之为肽感应与排毒分子(peptide sensing and detoxificationmodules,PSD),这是由TCS识别AMPs和ABC外排AMPs协同的结果[55]。
在信号调节还包括如下3个部分。1) 严禁反应:是细菌在贫养环境(如缺少氨基酸)中生长时,细胞的蛋白质合成及其他一些代谢活性被关闭的现象,其中ppGpp是介导严禁反应的关键,其可以诱导毒素抗毒素反应(toxin-antitoxin,TA)来控制细菌持留现象[56]。(p)ppGpp通过Obg与毒素HokB的转录激活相联系,Obg通过诱导HokB的表达可以在P. aeruginosa生物膜中控制持留,同时HokB水平的提高会引起生物膜的两极化与休眠[57]。2) TA模型:TA模型是诱导持留现象的关键,而第2类TA模型通常由2种蛋白组成,可以抑制细胞重要功能的毒素和由毒素组成并使毒素失活的抗毒素,在应激反应中抗毒素被降解成游离的毒素,而毒素可以阻碍DNA转录翻译、AMPs或细胞壁的合成[41]。3) SOS反应:可以修复受损的DNA,防止AMPs使细菌关键基因发生突变,以此对AMPs产生抗性。
4 小结革兰氏阳性菌多以较厚的肽聚糖层和改变表面电性抵抗AMPs的侵入,还会分泌一些蛋白酶或分子附着在细菌表面消除或减少AMPs。革兰氏阴性菌主要通过外膜修饰产生对AMPs的抗性。此外,细菌还可以分泌囊泡或胞外多糖、胞外蛋白来诱捕AMPs,外排泵同样会阻止AMPs接近细菌细胞膜。浮游菌达到一定菌群数时将会形成生物膜,所产生的诱导阻碍与结构障碍将会对AMPs产生更大的抗性。目前细菌对AMPs抗性的研究还只停留在实验室阶段,而大量临床试验证明AMPs所引起的细菌耐受性与传统抗生素相比轻微的多。因此AMPs作为新一代抗菌药物仍具有广阔的前景,但对其将来出现耐药性的可能性也应引起足够的重视。只有从AMPs抑菌机理和细菌对AMPs抗性机理2个方面综合考虑,才能设计出高效抗菌的AMPs,在投入生产时要严加控制大规模使用与滥用,这样才能扬长避短造福人类。
| [1] | NURI R, SHPRUNG T, SHAI Y. Defensive remodeling:how bacterial surface properties and biofilm formation promote resistance to antimicrobial peptides[J]. Biochimica et Biophysica Acta:Biomembranes, 2015, 1848(11): 3089–3100. DOI: 10.1016/j.bbamem.2015.05.022 |
| [2] | MARIA-NETO S, DE ALMEIDA K C, MACEDO M L R, et al. Understanding bacterial resistance to antimicrobial peptides:from the surface to deep inside[J]. Biochimica et Biophysica Acta:Biomembranes, 2015, 1848(11): 3078–3088. DOI: 10.1016/j.bbamem.2015.02.017 |
| [3] | NGUYEN L T, HANEY E F, VOGEL H J. The expanding scope of antimicrobial peptide structures and their modes of action[J]. Trends in Biotechnology, 2011, 29(9): 464–472. DOI: 10.1016/j.tibtech.2011.05.001 |
| [4] | OKUMURA C Y M, NIZET V. Subterfuge and sabotage:evasion of host innate defenses by invasive gram-positive bacterial pathogens[J]. Annual Review of Microbiology, 2014, 68(1): 439–458. DOI: 10.1146/annurev-micro-092412-155711 |
| [5] | LLOBET E, TOMÁS J M, BENGOECHEA J A. Capsule polysaccharide is a bacterial decoy for antimicrobial peptides[J]. Microbiology, 2008, 154(12): 3877–3886. DOI: 10.1099/mic.0.2008/022301-0 |
| [6] | CAMPOS M A, VARGAS M A, REGUEIRO V, et al. Capsule polysaccharide mediates bacterial resistance to antimicrobial peptides[J]. Infection and Immunity, 2004, 72(12): 7107–7114. DOI: 10.1128/IAI.72.12.7107-7114.2004 |
| [7] | FLEITAS O, AGBALE C M, FRANCO O L. Bacterial resistance to antimicrobial peptides:an evolving phenomenon[J]. Frontiers in Bioscience, 2016, 21(5): 1013–1038. DOI: 10.2741/4438 |
| [8] | SMYTH D, CAMERON A, DAVIES M R, et al. DrsG from Streptococcus dysgalactiae subsp.equisimilis inhibits the antimicrobial peptide LL-37[J]. Infection and Immunity, 2014, 82(6): 2337–2344. DOI: 10.1128/IAI.01411-13 |
| [9] | ROLIN O, MUSE S J, SAFI C, et al. Enzymatic modification of lipid A by ArnT protects Bordetella bronchiseptica against cationic peptides and is required for transmission[J]. Infection and Immunity, 2014, 82(2): 491–499. DOI: 10.1128/IAI.01260-12 |
| [10] | BAYER A S, MISHRA N N, SAKOULAS G, et al. Heterogeneity of mprF sequences in methicillin-resistant Staphylococcus aureus clinical isolates:role in cross-resistance between daptomycin and host defense antimicrobial peptides[J]. Antimicrobial Agents and Chemotherapy, 2014, 58(12): 7462–7467. DOI: 10.1128/AAC.03422-14 |
| [11] | COX E, MICHALAK A, PAGENTINE S, et al. Lysylated phospholipids stabilize models of bacterial lipid bilayers and protect against antimicrobial peptides[J]. Biochimica et Biophysica Acta:Biomembrane, 2014, 1838(9): 2198–2204. DOI: 10.1016/j.bbamem.2014.04.018 |
| [12] | ERNST C M, KUHN S, SLAVETINSKY C J, et al. The lipid-modifying multiple peptide resistance factor is an oligomer consisting of distinct interacting synthase and flippase subunits[J]. mBio, 2015, 6(1): e02340–14. |
| [13] | KUMARIYA R, SOOD S K, RAJPUT Y S, et al. Increased membrane surface positive charge and altered membrane fluidity leads to cationic antimicrobial peptide resistance in Enterococcus faecalis[J]. Biochimica et Biophysica Acta:Biomembrane, 2015, 1848(6): 1367–1375. DOI: 10.1016/j.bbamem.2015.03.007 |
| [14] | LATHER P, MOHANTY A K, JHA P, et al. Changes associated with cell membrane composition of Staphylococcus aureus on acquisition of resistance against class Ⅱa bacteriocin and its in vitro substantiation[J]. European Food Research and Technology, 2015, 240(1): 101–107. DOI: 10.1007/s00217-014-2311-z |
| [15] | SHAH N R, HANCOCK R E W, FERNANDEZ R C. Bordetella pertussis lipid A glucosamine modification confers resistance to cationic antimicrobial peptides and increases resistance to outer membrane perturbation[J]. Antimicrobial Agents and Chemotherapy, 2014, 58(8): 4931–4934. DOI: 10.1128/AAC.02590-14 |
| [16] | CULLEN T W, SCHOFIELD W B, BARRY N A, et al. Antimicrobial peptide resistance mediates resilience of prominent gut commensals during inflammation[J]. Science, 2015, 347(6218): 170–175. DOI: 10.1126/science.1260580 |
| [17] | HITTLE L E, JONES J W, HAJJAR A M, et al. Bordetella parapertussis PagP mediates the addition of two palmitates to the lipopolysaccharide lipid A[J]. Journal of Bacteriology, 2015, 197(3): 572–580. DOI: 10.1128/JB.02236-14 |
| [18] | THAIPISUTTIKUL I, HITTLE L E, CHANDRA R, et al. A divergent Pseudomonas aeruginosa palmitoyltransferase essential for cystic fibrosis-specific lipid A[J]. Molecular Microbiology, 2014, 91(1): 158–174. DOI: 10.1111/mmi.2014.91.issue-1 |
| [19] | BAND V I, WEISS D S. Mechanisms of antimicrobial peptide resistance in gram-negative bacteria[J]. Antibiotics, 2015, 4(1): 18–41. |
| [20] | HENDERSON J C, FAGE C D, CANNON J R, et al. Antimicrobial peptide resistance of vibrio cholerae results from an LPS modification pathway related to nonribosomal peptide synthetases[J]. ACS Chemical Biology, 2014, 9(10): 2382–2392. DOI: 10.1021/cb500438x |
| [21] | KIM J K, SON D W, KIM C H, et al. Insect gut symbiont susceptibility to host antimicrobial peptides caused by alteration of the bacterial cell envelope[J]. The Journal of Biological Chemistry, 2015, 290(34): 21042–21053. DOI: 10.1074/jbc.M115.651158 |
| [22] | JAISWAL S, PATI N B, DUBEY M, et al. The O-antigen negative △wbaV mutant of Salmonella enterica serovar Enteritidis shows adaptive resistance to antimicrobial peptides and elicits colitis in streptomycin pretreated mouse model[J]. Gut Pathogens, 2015, 7(1): 24. DOI: 10.1186/s13099-015-0070-4 |
| [23] | PERCY M G, GRVNDLING A. Lipoteichoic acid synthesis and function in gram-positive bacteria[J]. Annual Review of Microbiology, 2014, 68(1): 81–100. DOI: 10.1146/annurev-micro-091213-112949 |
| [24] | NEUHAUS F C, BADDILEY J. A continuum of anionic charge:structures and functions of D-alanyl-teichoic acids in gram-positive bacteria[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 686–723. DOI: 10.1128/MMBR.67.4.686-723.2003 |
| [25] | CARVALHO F, ATILANO M L, POMBINHO R, et al. L-rhamnosylation of Listeria monocytogenes wall teichoic acids promotes resistance to antimicrobial peptides by delaying interaction with the membrane[J]. PLoS Pathogens, 2015, 11(5): e1004919. DOI: 10.1371/journal.ppat.1004919 |
| [26] | DE LA FUENTE-NÙÑEZ C, REFFUVEILLE F, FERNÁNDEZ L, et al. Bacterial biofilm development as a multicellular adaptation:antibiotic resistance and new therapeutic strategies[J]. Current Opinion in Microbiology, 2013, 16(5): 580–589. DOI: 10.1016/j.mib.2013.06.013 |
| [27] | HALL-STOODLEY L, COSTERTON J W, STOODLEY P. Bacterial biofilms:from the natural environment to infectious diseases[J]. Nature Reviews Microbiology, 2004, 2(2): 95–108. DOI: 10.1038/nrmicro821 |
| [28] | COSTERTON J W, STEWART P S, GREENBERG E P. Bacterial biofilms:a common cause of persistent infections[J]. Science, 1999, 284(5418): 1318–1322. DOI: 10.1126/science.284.5418.1318 |
| [29] | RABIN N, ZHENG Y, OPOKU-TEMENG C, et al. Biofilm formation mechanisms and targets for developing antibiofilm agents[J]. Future Medicinal Chemistry, 2015, 7(4): 493–512. DOI: 10.4155/fmc.15.6 |
| [30] | FLEMMING H C, NEU T R, WOZNIAK D J. The EPS matrix:the "house of biofilm cells"[J]. Journal of Bacteriology, 2007, 189(22): 7945–7947. DOI: 10.1128/JB.00858-07 |
| [31] | NWODO U U, GREEN E, OKOH A I. Bacterial exopolysaccharides:functionality and prospects[J]. International Journal of Molecular Sciences, 2012, 13(11): 14002–14015. |
| [32] | DAS T, SHARMA P K, BUSSCHER H J, et al. Role of extracellular DNA in initial bacterial adhesion and surface aggregation[J]. Applied and Environmental Microbiology, 2010, 76(10): 3405–3408. DOI: 10.1128/AEM.03119-09 |
| [33] | PALMER J, FLINT S, BROOKS J. Bacterial cell attachment, the beginning of a biofilm[J]. Journal of Industrial Microbiology & Biotechnology, 2007, 34(9): 577–588. |
| [34] | O'TOOLE G, KAPLAN H B, KOLTER R. Biofilm formation as microbial development[J]. Annual Review of Microbiology, 2000, 54(1): 49–79. DOI: 10.1146/annurev.micro.54.1.49 |
| [35] | RENNER L D, WEIBEL D B. Physicochemical regulation of biofilm formation[J]. MRS Bulletin, 2011, 36(5): 347–355. DOI: 10.1557/mrs.2011.65 |
| [36] | BATONI G, MAISETTA G, ESIN S. Antimicrobial peptides and their interaction with biofilms of medically relevant bacteria[J]. Biochimica et Biophysica Acta:Biomembrane, 2016, 1858(5): 1044–1060. DOI: 10.1016/j.bbamem.2015.10.013 |
| [37] | RIBEIRO S M, FELÍCIO M R, BOAS E V, et al. New frontiers for anti-biofilm drug development[J]. Pharmacology & Therapeutics, 2016, 160: 133–144. |
| [38] | RASAMIRAVAKA T, LABTANI Q, DUEZ P, et al. The formation of biofilms by Pseudomonas aeruginosa:a review of the natural and synthetic compounds interfering with control mechanisms[J]. Biomed Research International, 2015, 2015: 759348. |
| [39] | RODRÍGUEZ-ROJAS A, MAKAROVA O, MÜLLER U, et al. Cationic peptides facilitate iron-induced mutagenesis in bacteria[J]. PLoS Genetics, 2015, 11(10): e1005546. DOI: 10.1371/journal.pgen.1005546 |
| [40] | BAHAR A A, LIU Z G, GARAFALO M, et al. Controlling persister and biofilm cells of gram-negative bacteria with a new 1, 3, 5-triazine derivative[J]. Pharmaceuticals, 2015, 8(4): 696–710. DOI: 10.3390/ph8040696 |
| [41] | VAN ACKER H, COENYE T. The role of efflux and physiological adaptation in biofilm tolerance and resistance[J]. The Journal of Biological Chemistry, 2016, 291(24): 12565–12572. DOI: 10.1074/jbc.R115.707257 |
| [42] | WOLSKA K I, GRUDNIAK A M, RUDNICKA Z, et al. Genetic control of bacterial biofilms[J]. Journal of Applied Genetics, 2016, 57(2): 225–238. DOI: 10.1007/s13353-015-0309-2 |
| [43] | FAZLI M, ALMBLAD H, RYBTKE M L, et al. Regulation of biofilm formation in P seudomonas and B urkholderia species[J]. Environmental Microbiology, 2014, 16(7): 1961–1981. DOI: 10.1111/emi.2014.16.issue-7 |
| [44] | RÖMLING U, GALPERIN M Y, GOMELSKY M. Cyclic di-GMP:the first 25 years of a universal bacterial second messenger[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 1–52. DOI: 10.1128/MMBR.00043-12 |
| [45] | HENGGE R. Principles of c-di-GMP signalling in bacteria[J]. Nature Reviews Microbiology, 2009, 7(4): 263–273. DOI: 10.1038/nrmicro2109 |
| [46] | KALIA V C, WOOD T K, KUMAR P. Evolution of resistance to quorum-sensing inhibitors[J]. Microbial Ecology, 2014, 68(1): 13–23. DOI: 10.1007/s00248-013-0316-y |
| [47] | BOLES B R, THOENDEL M, ROTH A J, et al. Identification of genes involved in polysaccharide-independent Staphylococcus aureus biofilm formation[J]. PLoS One, 2010, 5(4): e10146. DOI: 10.1371/journal.pone.0010146 |
| [48] | GROISMAN E A, MOUSLIM C. Sensing by bacterial regulatory systems in host and non-host environments[J]. Nature Reviews Microbiology, 2006, 4(9): 705–709. DOI: 10.1038/nrmicro1478 |
| [49] | LIU Y H, ZHANG Q F, HU M, et al. Proteomic analyses of intracellular Salmonella enterica serovar typhimurium reveal extensive bacterial adaptations to infected host epithelial cells[J]. Infection and Immunity, 2015, 83(7): 2897–2906. DOI: 10.1128/IAI.02882-14 |
| [50] | CHEN H D, GROISMAN E A. The biology of the PmrA/PmrB two-component system:the major regulator of lipopolysaccharide modifications[J]. Annual Review of Microbiology, 2013, 67(1): 83–112. DOI: 10.1146/annurev-micro-092412-155751 |
| [51] | 葛琳, 郭大伟, 何方, 等. PmrA-PmrB二元调控系统介导大肠杆菌对黏杆菌素耐药的机制研究[J]. 畜牧兽医学报, 2016, 47(4) :812–819. |
| [52] | CHEUNG A L, BAYER A S, YEAMAN M R, et al. Site-specific mutation of the sensor kinase GraS in Staphylococcus aureus alters the adaptive response to distinct cationic antimicrobial peptides[J]. Infection and Immunity, 2014, 82(12): 5336–5345. DOI: 10.1128/IAI.02480-14 |
| [53] | BILECEN K, FONG J C N, CHENG A, et al. Polymyxin B resistance and biofilm formation in Vibrio cholerae are controlled by the response regulator CarR[J]. Infection and Immunity, 2015, 83(3): 1199–1209. DOI: 10.1128/IAI.02700-14 |
| [54] | RUBIN E J, HERRERA C M, CROFTS A A, et al. PmrD is required for modifications to Escherichia coli endotoxin that promote antimicrobial resistance[J]. Antimicrobial Agents and Chemotherapy, 2015, 59(4): 2051–2061. DOI: 10.1128/AAC.05052-14 |
| [55] | DINTNER S, HEERMANN R, FANG C, et al. A sensory complex consisting of an ATP-binding cassette transporter and a two-component regulatory system controls bacitracin resistance in Bacillus subtilis[J]. The Journal of Biological Chemistry, 2014, 289(40): 27899–27910. DOI: 10.1074/jbc.M114.596221 |
| [56] | MAISONNEUVE E, CASTRO-CAMARGO M, GERDES K. (p)ppGpp controls bacterial persistence by stochastic induction of toxin-antitoxin activity[J]. Cell, 2013, 154(5): 1140–1150. DOI: 10.1016/j.cell.2013.07.048 |
| [57] | VERSTRAETEN N, KNAPEN W J, KINT C I, et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance[J]. Molecular Cell, 2015, 59(1): 9–21. DOI: 10.1016/j.molcel.2015.05.011 |