2. 太仓市金诸农业发展有限公司, 太仓 215400;
3. 江苏徐淮地区淮阴农业科学研究所, 淮安 223001
2. Taicang JinChu Agricultural Development Co., Ltd., Taicang 215400, China;
3. Huaiyin Agricultural Science Research Institute of Jiangsu Xuhuai Region, Huai'an 223001, China
在过去的几十年,抗生素在畜牧业中对于治疗疾病、预防疾病、促进生长、提高饲料转化效率等方面效果显著[1-2],对养殖业的发展起到巨大的推动作用。然而近年有关抗生素的副作用,例如长期饲喂抗生素会导致抗生素耐药基因、耐药菌株的增加,抗生素在亚抑菌浓度下会调控细菌基因表达以及畜禽内源性感染和二重感染等问题引起了人们对抗生素使用的重视[3]。美国传染病学会(infectious disease society of America)已经建议美国政府限制抗生素在农业方面的使用[4],欧盟也已经禁止用抗生素促进动物生长[5]。然而在国内,在现代以及传统农业结合的生产方式下由抗生素带来的成本效益,使得众多国内的畜牧行业不得不坚持使用抗生素。
抗生素的作用效果明显,目前人们认可的其抗菌机制主要是抗生素能抑制细菌胞壁合成、损害细菌胞浆膜、抑制细菌蛋白质合成、抑制核酸合成等。有关抗生素促生长的作用机制研究也很多,Francois[6]和Visek[7]认为,抗生素可以控制动物的亚临床感染,降低对动物生长有抑制作用的微生物代谢产物,降低微生物对宿主动物的养分竞争,使动物肠壁变薄,促进营养物质吸收,从而促进生长。也有研究表明,抗生素可以减少胃肠道乳酸杆菌的数量从而促进动物对营养物质的消化吸收[8]。Rosen[9]总结认为,抗生素有可能促进有益菌的生长,从而抑制有害菌的生长。Dibner等[10]研究认为,肠道微生物可竞争抵御致病菌和其他微生物在肠道中的定植,激活宿主肠道防御体系,对宿主既具有营养作用,也会与宿主争夺营养物质。因此不难看出,抗生素对肠道微生物的影响在促生长机制中占有重要地位,许多学者的研究也证明了这一点。然而目前国内有关抗生素对肠道微生物的影响研究大多使用平板计数法,只能对极少数的微生物或者某些感兴趣的微生物进行检测。随着PCR技术的出现及核酸研究技术的不断完善,16S rRNA基因检测技术已成为病原菌检测和鉴定的一种强有力工具。
肠道微生物是一个庞大且动态的生态系统,随着研究的深入,肠道微生物不仅可以产生个体生命活动中必不可少的产物,对病原体形成一个屏障,而且在肠道形态、免疫、消化、调控宿主基因表达方面发挥着重要的作用[11-12]。肠道微生物可以协助宿主消化代谢、合成维生素、病原体异位以及促进免疫系统成熟[13-14]。已经有报导证实,人类疾病,如肥胖、糖尿病、肠道炎症疾病等,都与肠道微生物的变化密切相关[15-18]。对于猪而言,肠道微生物对猪胃肠道免疫系统的发展有重要的促进作用,同时也和腹泻密切相关[19-20]。养分有效性、pH、氧化还原电位以及肠道的蠕动都会影响动物肠道微生物的组成[21],对有益肠道微生物群的干扰是抗生素的一个潜在的作用。研究抗生素对微生物群落的影响对全面了解饲用抗生素带来的好处和后果十分重要。因此,本文利用16S rRNA技术对比添加抗生素和不添加抗生素的仔猪肠道菌群组成,旨在从肠道微生物的角度再次探究抗生素的作用机制,为绿色抗生素替代品的研究提供理论基础和参考。
1 材料与方法 1.1 试验动物与试验设计试验所用抗生素为复合抗生素,即杆菌肽锌与黏杆菌素以5 : 1混合。
试验在江苏省江阴定山猪场进行。试验选取6头30日龄的杜×长×大三元杂交去势公猪,随机分为2组,分别为基础饲粮组(不添加抗生素)和抗生素组(基础饲粮中添加0.12%复合抗生素),每组3个重复,每个重复1头猪。试验期为35 d,每天08:00和16:00各饲喂1次,自由采食和饮水。每头猪按猪场规定接种常规疫苗,猪舍每天打扫2次,定期消毒。基础饲粮为粉状配合饲料,参照NRC(2012) 猪营养需要量配制。基础饲粮组成及营养水平见表 1。
|
|
表 1 基础饲粮组成及营养水平(风干基础) Table 1 Composition and nutrient levels of the basic diet (air-dry basis) |
在试验的最后1天(仔猪65日龄),将6头试验猪全部屠宰,打开腹腔,分离结肠,采取结肠食糜于1.5 mL灭菌冻存管,迅速置于液氮保存,用来测定微生物组成。
1.3 结肠食糜微生物区系分析采用微生物DNA抽提试剂盒(德国,QIAGEN公司)提取100 mg样品基因组DNA,具体步骤按说明书操作。利用Thermo NanoDrop 2000紫外微量分光光度计(美国)和1%琼脂糖凝胶电泳进行总DNA质检。将提取的DNA作为模板,PCR扩增16S rDNA V3~V4可变区。引物序列为上游:338F 5′-ACTCCTACGGGAGGCAGCA-3′;下游:806R 5′-GGACTACHVGGGTWTCTAAT-3′。在通用引物的5′端加上适合HiSeq2500 PE250测序的index序列和接头序列,完成特异性引物的设计。PCR扩增体系(20 μL):5×FastPfu Buffer 4 μL,2.5 mmol/L dNTPs 2 μL,上、下游引物(5 μmol/L)各0.8 μL,FastPfu Polymerase 0.4 μL,DNA模板10 ng。PCR扩增条件为:95 ℃ 2 min,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,25个循环,最后72 ℃延伸5 min。用2%琼脂糖凝胶电泳检测PCR产物,并用AxyPrep DNA凝胶回收试剂盒(美国,AXYGEN公司)切胶回收PCR产物。回收后,利用Thermo NanoDrop 2000紫外微量分光光度计(美国)和2%琼脂糖凝胶电泳进行文库质检。根据标准协议使用Illumina MiSeq平台进行上机测序。
测序后首先对原始数据进行质控得到Clean reads,用Usearch(v7.0.1090) 软件对数据进行去嵌合体和聚类的操作,Usearch聚类时,先将Reads按照丰度从大到小排序,通过97%相似度的标准聚类,得到操作分类单元(operational taxonomic units,OTU),每个OTU被认为可代表 1个物种。接下来对每个样品的Reads进行随机抽平处理,并提取对应的OTU序列。然后使用Qiime软件,做Alpha多样性指数的稀释曲线,根据稀释曲线选择合理的抽平参数,利用Qiime软件对得到的抽平后的OTU进行分析,首先从OTU中分别提取1条Read作为代表序列,使用RDP方法,将该代表序列与16S数据库比对,从而对每个OTU进行物种分类。归类后,根据每个OTU中序列的条数,从而得到OTU丰度表,最后根据该OTU丰度表进行后续分析。
1.4 数据统计与分析本研究中组间显著性差异分析是通过统计学的方法检验2组样品间微生物群落丰度的差异,并使用伪发现率(false discovery rate,FDR)评估差异的显著性。从检验结果中,可以筛选出导致2组样品组成差异的物种。本分析分别在门、纲、目、科、属、种分类等级进行组间显著性差异分析。使用Metastats(http://metastats.cbcb.umd.edu/)软件或者R(v3.0.3) 软件(秩和检验、Fisher’s精确检验、卡方检验、t检验、方差检验)进行组间显著性差异分析。P值校正通过R(v3.0.3) 软件包中的p.adjust进行,校正方法为“BH”(即Benjamini-CHochberg)。
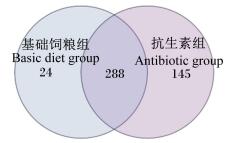
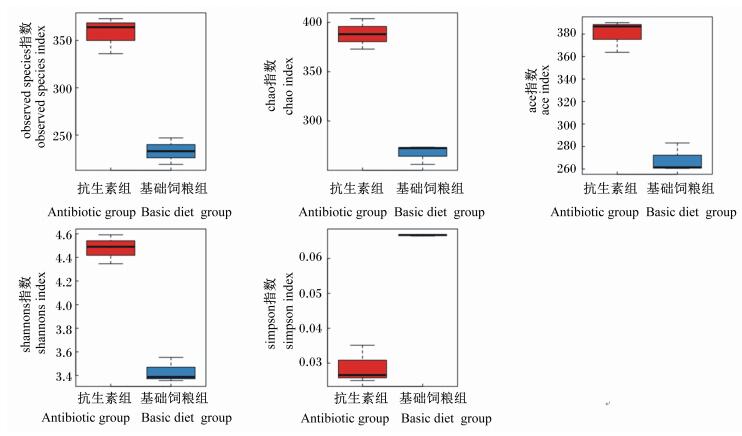
2 结果与分析 2.1 OTU及其丰度分析拼接的Clean reads经过优化后,在97%相似度下将其聚类为用于物种分类的OTU,26个样品共产生457个OTU,个体样本OTU统计见表 2,将每个样品的OTU个数进行Venn分析,展示OTU的重叠情况,如图 1所示,基础饲粮组和抗生素组共有288个OTU,抗生素组的特有OTU有145个,而基础饲粮组的特有OTU只有24个。对2组中的OTU进行Alpha多样性分析,图 2中分别描述了observed species指数、chao指数、ace指数、shannon指数以及simpson指数,抗生素组在前面4个指数中均明显高于基础饲粮组,在最后一个指数中明显低于基础饲粮组,说明抗生素组中的物种更丰富。这也再次验证了图 1 Venn分析中,抗生素组的特有OTU更多的结果。
|
|
表 2 样品OTU统计 Table 2 The sample's OTU statistics |
|
图 1 OTU Venn分析 Figure 1 The Venn analysis of OTU |
|
盒形图可以显示5个统计量(最小值,第1个四分位数,中位数,第3个中位数和最大值,即由下到上的5条线)。 Five statistics can be displayed in the box diagram (minimum value, the first quartile, median, the third median and maximum value, they are five lines from bottom to top). 图 2 组间Alpha多样性盒形图 Figure 2 Alpha diversity box shape among groups |
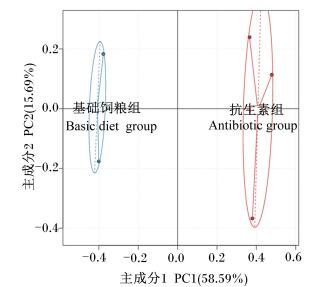
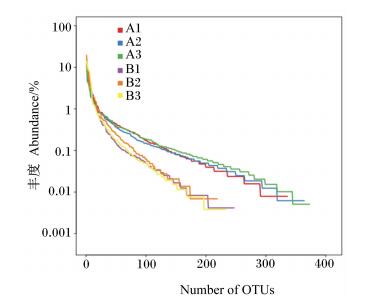
物种的多样性包含2个方面,即样品所含物种的丰富程度和均匀度。我们利用每个OTU在每个样品的相对丰度,进行OTU的主成分分析(principal component analysis,PCA)。在PCA中如果2个样品距离越近,则表示这2个样品的组成越相似,如图 3所示,在PCA中,2组样品很明显的被区分开,说明2组样品的微生物结构及丰度明显不同。OTU Rank曲线也说明了这个结果,如图 4所示,抗生素组3个个体的横轴长度较基础饲粮组更宽,曲线也较基础饲粮组更平坦,说明抗生素样品中物种组成越丰富,物种均匀度也越高。
|
图中每个点分别表示每个样品,不同颜色代表样品属于不同的分组。 Each point in the graph represents each sample, and the different colors represent the different samples. 图 3 主成分分析 Figure 3 Principal component analysis |
|
A1:抗生素1 Antibiotic 1, A2:抗生素2 Antibiotic 2, A3:抗生素3 Antibiotic 3, B1:基础饲粮1 Basic diet 1, B2:基础饲粮2 Basic diet 2, B3:基础饲粮3 Basic diet 3。图 5同。The same as Fig. 5. 图 4 OTU Rank曲线图 Figure 4 The graph of OTU rank |
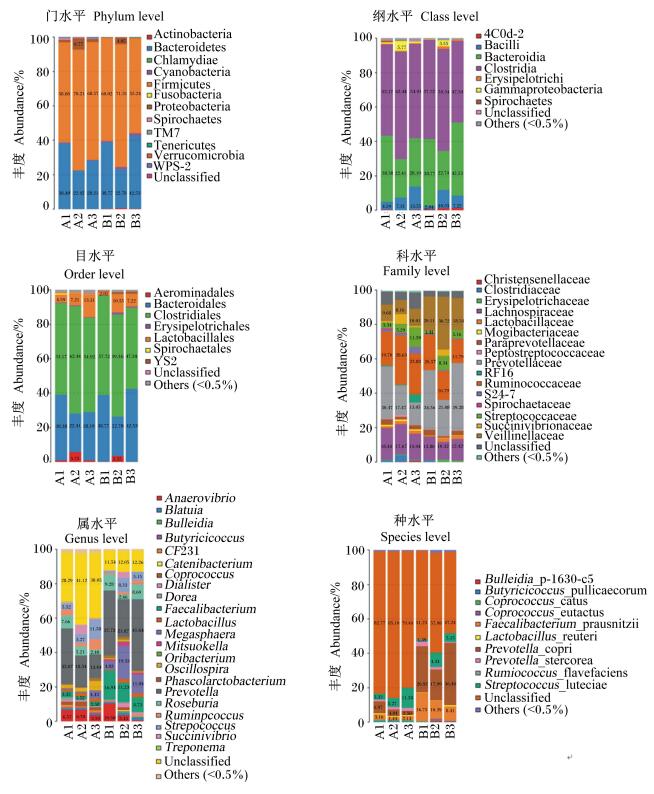
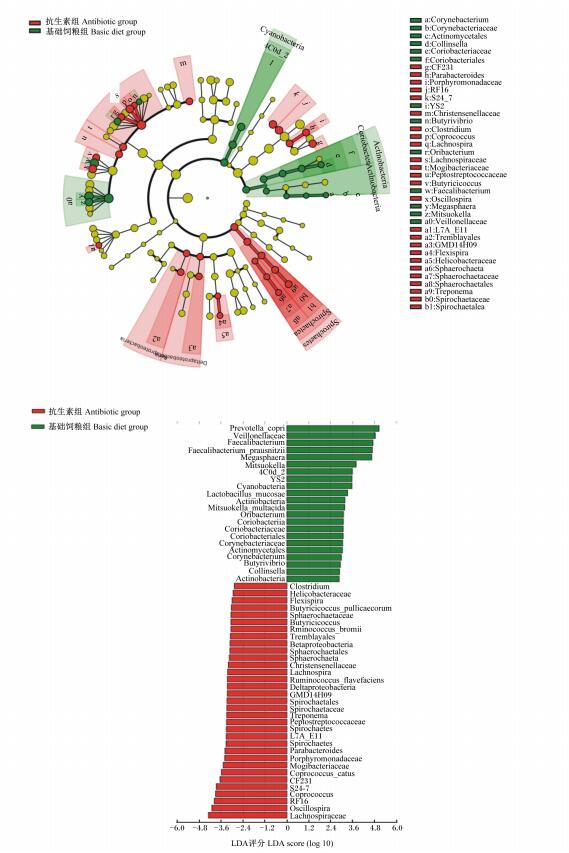
通过与数据库进行比对,对OTU进行物种分类并分别在门、纲、目、科、属、种几个分类等级对各个样品作物种profiling柱状图(图 5),结合LEFSE分析图(图 6),在门水平上,2组的微生物组成结构如图 6所示。根据测序结果,在门水平上,占主导作用的主要是放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、蓝藻门(Cyanobacteria)、厚壁菌门(Firmicutes)、梭杆菌门(Fusobacteria)、变形菌门(Proteobacteria)及螺旋体门(Spirochaetes)等(丰度大于0.5%)。抗生素组的Spirochaetes、δ-变形菌纲(Deltaproteobacteria)、β-变形菌(Betaproteobacteria)的丰度显著高于基础饲粮组(P<0.05),Actinobacteria和Cyanobacteria的丰度显著低于基础饲粮组(P<0.05)。其中Spirochaetes、Actinobacteria和Cyanobacteria的丰度超过0.5%。另外在抗生素组中多出了衣原体门(Chlamydiae)、疣微菌门(Verrucomicrobia)和WPS-2等。在属水平上,2组的微生物组成结构如图 6所示,排在前5名的是普雷沃菌属(Prevotella)、巨球型菌属(Megasphaera)、链球菌属(Streptococcus)、柔嫩菌属(Faecalibacterium)和厌氧弧菌属(Anaerovibrio)。抗生素组的L7A_E11、Flexispira、粪球菌属(Coprococcus)、毛螺菌属(Lachnospira)、Sphaerochaeta、密螺旋体属(Treponema)、CF231、Parabacteroides、梭状芽孢杆菌(Clostridium)、Butyricicoccus及颤螺菌属(Oscillospira)显著高于基础饲粮组(P<0.05),而Mitsuokella、Megasphaera、Faecalibacterium、Oribacterium、丁酸弧菌属(Butyrivibrio)、Collinseella及棒状杆菌(Corynebacterium)显著低于基础饲粮组(P<0.05)。其中Faecalibacterium、巨型球属(Megasphaera)、光岗菌属(Mitsuokella)、Oribacterium、CF231、Coprococcus及Treponema的丰度超过0.5%。
|
门水平为所有物种的柱状图,从纲水平开始,物种丰度均高于0.5%其余全部合并成Others。 Phylum level for all species, starting from the class level, the species richness was higher than 0.5%, which less than 0.5% were all merged into Others. 图 5 样品在不同分类水平中物种profiling柱状图 Figure 5 Species profiling histogram of samples in different classification level |
|
左图中为LEfSE聚类树,不同颜色表示不同分组,不同颜色的节点表示在该颜色所代表的分组中起到重要作用的微生物群,一个颜色圈点代表一个biomarker(组间在丰度上存在显著差异的物种),中间图例为biomarker名称。黄色节点表示的是在不同分组中没有起到重要作用的微生物类群。右图为差异显著物种集合(P<0.05)。 The left figure is LEfSE cluster tree, different colors represent different groups, and node with different colors represents the microbiota which plays an important role in the group. A color dots represents a biomarker, the middle legend is the name to biomarker. The yellow node represents microorganism that do not play an important role in different groups. The right figure is the significant different species (P < 0.05). 图 6 LEFSE分析图 Figure 6 LEFSE analysis chart |
除此之外,本研究还筛选出在其他水平上既满足丰度大于0.5%,又在2组中差异显著的菌种(P<0.05),如在纲水平上只有4C0d_2在基础饲粮组显著高于抗生素组(P<0.05);目水平上基础饲粮组的标记微生物为YS2,抗生素组为螺旋体目(Spirochaetales);在科水平上基础饲粮组的标记微生物为韦荣球菌科(Veillonellaceae)、抗生素组为Christensenellaceae、毛螺菌科(Lachnospiraceae)、消化链球菌科(Peptostreptococcaceae)、RF16、S24_7、螺旋体科(Spirochaetaceae);在种水平上基础饲粮组的标记微生物为柔嫩梭菌群(Faecalibacterium_prausnitzii)、人体普氏菌(prevotella_copri),而抗生素组为Butyricicoccus_pullicaecorum、Coprococcus_catus、生黄瘤胃球菌(Ruminococcus_flavefaciens)。
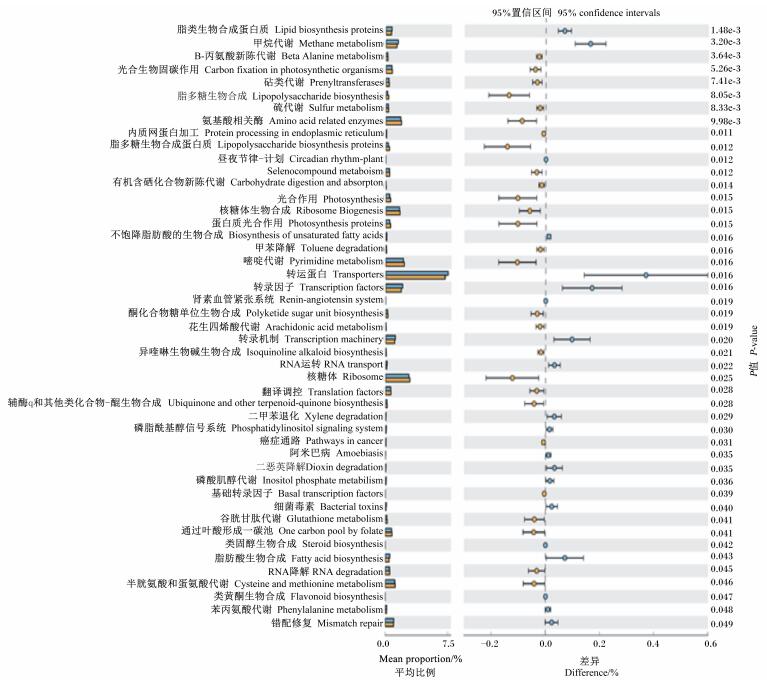
2.3 抗生素组对比基础饲粮组16S功能预测为了比较抗生素组和基础饲粮组中微生物的功能,使用PICRUSt对其进行功能预测。如图 7所示,抗生素组在脂类生物合成蛋白质、甲烷代谢、转运蛋白、转录因子、转录机制、RNA转运、脂肪酸生物合成等通路中发挥更大的功能。
|
图 7 16S功能预测 Figure 7 16S function prediction |
肠道是一个复杂的微生态系统,在肠道黏膜表面有大量的微生物菌群定植。动物在健康状态下,其肠道微生物处于动态平衡状态,这些长期定植在动物肠道的微生物作为肠道内环境的重要组成部分而存在[22]。肠道微生物菌群通过定植肠道,增加抵抗力,帮助机体防御病原菌的入侵[23]。也可通过增强机体对病原菌的免疫应答和阻止病原菌在肠道的附着,帮助机体免受外来病原菌感染[24]。研究证实,无菌动物比肠道内微生物菌群正常的动物更容易遭受病原菌感染[25-27]。本研究中,肠道微生物的结构确实因为抗生素的添加与否发生了改变,抗生素组的肠道微生物多样性明显高于基础饲粮组。长期以来,人们一直认为饲用抗生素的促生长作用是源于其对肠道微生物的抑制作用。从这里我们可以推测,抗生素可能是通过抑制某些有害菌,从而给更多有益菌增加了繁殖的空间,最终发挥作用。
本试验结果表明,无论在何水平,热图的聚众分析均将3个抗生素个体聚在一起,将3个基础饲粮个体聚在一起,说明2个组内的菌群整体不存在明显差异,而组间差异显著,试验分组合理。在门水平上,无论是基础饲粮组还是抗生素组肠道中的优势菌群都是Firmicutes和Bacteroidetes,值得关注的是Firmicutes和Bacteroidetes都是发酵多糖的主力军,Ley等[28]和Turnbaugh等[11]比较了胖鼠和瘦鼠盲肠内容物的菌群,Ley等[15]还比较了胖人和瘦人粪便的菌群,结果一致表明胖个体的Bacteroidetes丰度较低,而Firmicutes的丰度较高,且Firmicute/Bacteroidetes(F/B)比例在胖个体中偏高。特别是当肠道内F/B比例较高,宿主摄取食物中能量的能力会加强,体内脂肪的贮存也会加剧[28]。Bckhed等[29]用发现无菌鼠接种多形Bacteroidetes后其体脂肪显著增加了23%,但增加的幅度小于接种盲肠混合微生物的(含有高比例的Bacteroidetes),这说明Bacteroidetes有促进脂肪沉积的作用,这可能与Bacteroidetes能分解植物多糖有关,但促进脂肪沉积的效果不如Firmicutes高的细菌群体。Firmicutes与Bacteroidetes两者可能存在一种协同的共生关系,高F/B比例可能更易促进宿主吸收/储存能量。国内郭秀兰[30]也进一步证实猪脂肪沉积与肠道的Bacteroidetes及F/B比例相关,他们认为调控肠道微生物可作为控制猪脂肪沉积的途径之一。根据本试验数据,抗生素组的F/B比例为2.21,基础饲粮组的F/B比例为1.79,因此本研究推测添加抗生素有利于仔猪体内脂肪的贮存。
在属水平上,抗生素组的L7A_E11、Flexispira、Coprococcus、Lachnospira、Sphaerochaeta、Treponema、CF231、Parabacteroides、Clostridium、Butyricicoccus和Oscillospira显著高于基础饲粮组,而抗生素组的Mitsuokella、Megasphaera、Faecalibacterium、Oribacterium、Butyrivibrio、Collinseella、Corynebacterium显著低于基础饲粮组。其中Treponema、Coprococcus、Lachnospira和L7A_E11与粗饲料中果胶降解密切相关,能通过促进瘤胃微生物蛋白质合成,进而提高动物生产性能[31]。Clostridium是丁酸产生的主要菌,微生物发酵产生的短链脂肪酸,特别是丁酸,在维持宿主健康和疾病预防上发挥着重要的作用,它可以向宿主结肠上皮细胞提供比葡萄糖更优化的碳源和氮源,不仅能促进肠上皮细胞的生长,加速受损肠黏膜的修复,还能生理性地调控肠上皮细胞的基因表达,有效地抑制肠炎和结肠直肠癌的发生[32]。Oscillospira是一种较难培养的厌氧菌,常见于草食动物肠道中[33],姬玉娇等[34]发现,Oscillospira为环江香猪结肠内容物中的优势菌属,这与其耐粗饲有很大关系。相反动植物致病性Corynebacterium会引起家畜各种器官部位的化脓性感染和小麦等农作物的溃疡、萎蔫疾病等[35]。因此抗生素可能是通过对部分有益菌的促进作用,抑制部分有害菌从而发挥自身的作用。
3.2 抗生素组引起的功能通路变化本研究中用到的抗生素为杆菌肽锌和黏杆菌素。杆菌肽锌属多肽类抗生素,对革兰阳性菌具有杀菌作用,其机制主要为抑制细菌的细菌壁合成,也能与敏感细菌的细胞膜结合,损伤细胞膜的完整性,导致细胞内重要物质外流。它可以促进畜禽生长,提高饲料转换率。黏杆菌素为抗革兰阴性杆菌抗生素,具有杀菌作用,对大多数革兰阴性杆菌有较强抗菌作用,杆菌肽锌与黏杆菌素有协同抗菌作用,作用于仔猪具有预防疾病、提高成活率、促进生长的作用。本研究中,通过16S功能预测,发现抗生素组在脂类生物合成蛋白质、甲烷代谢、转运蛋白、转录因子、转录机制、RNA转运、脂肪酸生物合成等通路中发挥更大的功能。猪肉作为人类最常见的肉类食品之一,脂肪是必不可少的组成成分,皮下脂肪也是肉品加工的重要原料,可以为人类提供必需脂肪酸,并协助脂溶性维生素和胡萝卜素的吸收[36]。对脂类生物合成蛋白质及脂肪酸生物合成等通路的促进作用,有益于仔猪脂肪的合成。甲烷是重要的温室气体,在集约化饲养的猪舍,猪本身的呼吸和舍内粪便会排放大量的温室气体,如何科学有效地处理利用猪场粪便污水,控制环境污染和粪便管理过程中产生的甲烷等温室气体排放,已成为社会关注的问题[37]。加速甲烷气体的代谢可能也是添加抗生素的一个潜在作用。
4 结论① 抗生素组肠道微生物物种多样性较基础饲粮组更丰富,且F/B比例较高,因此添加抗生素有利于仔猪体内脂肪的贮存。
② 抗生素可能是通过增加L7A_E11、Flexispira、Coprococcus、Lachnospira、Sphaerochaeta、Treponema、CF231、Parabacteroides、Clostridium、Butyricicoccus和Oscillospira等有益菌属,抑制Mitsuokella、Megasphaera、Faecalibacterium、Oribacterium、Butyrivibrio、Collinseella、Corynebacterium等菌属,从而发挥预防疾病、提高成活率、促进生长的作用。
③ 通过16S功能预测,发现抗生素在脂类生物合成蛋白质、甲烷代谢、转运蛋白、转录因子、转录机制、RNA转运和脂肪酸生物合成等通路中发挥了重要的功能。
| [1] | CROMWELL G L. Why and how antibiotics are used in swine production[J]. Animal Biotechnology, 2002, 13(1): 7–27. DOI: 10.1081/ABIO-120005767 |
| [2] | 曾明富. 抗生素在兽医临床上的科学使用[J]. 乡村科技, 2016, 6 :32–33. |
| [3] | 史自涛, 姚焰础, 江山, 等. 粪肠球菌替代抗生素对断奶仔猪生长性能、腹泻率、血液生化指标和免疫器官的影响[J]. 动物营养学报, 2015, 27(6) :1832–1840. |
| [4] | Infectious Diseases Society of America.The Infectious Diseases Society of America's (IDSA) statement on antibiotic resistance:promoting judicious use of medically important antibiotics in animal agriculture.Before the House Committee on Energy and Commerce Subcommittee on Health[R]. Washington, D.C.:House Committee on Energy and Commerce, 2010. |
| [5] | US Department of Health and Human Services Food and Drug Administration Center for Veterinary Medicine.The judicious use of medically important antimicrobial drugs in food-producing animals.Draft guidance No.209[R]. Rockville, MD:US Department of Health and Human Services Food and Drug Administration Center for Veterinary Medicine, 2010. |
| [6] | FRANCOIS A C. Mode of action of antibiotics on growth[J]. World Review of Nutrition and Dietetics, 1961, 3: 21–64. |
| [7] | VISEK W J. The mode of growth promotion by antibiotics[J]. Journal of Animal Science, 1978, 46(5): 1447–1469. DOI: 10.2527/jas1978.4651447x |
| [8] | 李同洲, 臧素敏, 李德发. 饲用抗生素对仔猪肠道菌群及肠道物质代谢影响的研究[J]. 饲料研究, 1999(5) :3–5. |
| [9] | ROSEN G D.Antibacterials in poultry and pig nutrition[M]//WALLACE R J, CHESSON A.Biotechnology in Animal Feeds and Animal Feeding.Weinheim, Germany:VCH Verlagsgesellschaft mbH, 1995:143-172. |
| [10] | DIBNER J J, RICHARDS J D. Antibiotic growth promoters in agriculture:history and mode of action[J]. Poultry Science, 2005, 84(4): 634–643. DOI: 10.1093/ps/84.4.634 |
| [11] | TURNBAUGH P J, LEY R E, MAHOWALD M A, et al. An obesity-associated gut microbiome with increased capacity for energy harvest[J]. Nature, 2006, 444(7122): 1027–1031. DOI: 10.1038/nature05414 |
| [12] | GUO X, XIA X, TANG R, et al. Development of a real-time PCR method for Firmicutes and Bacteroidetes in faeces and its application to quantify intestinal population of obese and lean pigs[J]. Letters in Applied Microbiology, 2008, 47(5): 367–373. DOI: 10.1111/lam.2008.47.issue-5 |
| [13] | GREEN G L, BROSTOFF J, HUDSPITH B, et al. Molecular characterization of the bacteria adherent to human colorectal mucosa[J]. Journal of Applied Microbiology, 2006, 100(3): 460–469. DOI: 10.1111/jam.2006.100.issue-3 |
| [14] | BIRD A R, CONLON M A, CHRISTOPHERSEN C T, et al. Resistant starch, large bowel fermentation and a broader perspective of prebiotics and probiotics[J]. Beneficial Microbes, 2010, 1(4): 423–431. DOI: 10.3920/BM2010.0041 |
| [15] | LEY R E, TURNBAUGH P J, KLEIN S, et al. Microbial ecology:human gut microbes associated with obesity[J]. Nature, 2006, 444(7122): 1022–1023. DOI: 10.1038/4441022a |
| [16] | SEKIROV I, RUSSELL S L, ANTUNES C M, et al. Gut microbiota in health and disease[J]. Physiological Reviews, 2010, 90(3): 859–904. DOI: 10.1152/physrev.00045.2009 |
| [17] | QIN J J, LI Y R, CAI Z M, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes[J]. Nature, 2012, 490(7418): 55–60. DOI: 10.1038/nature11450 |
| [18] | CONTE M P, SCHIPPA S, ZAMBONI I, et al. Gut-associated bacterial microbiota in paediatric patients with inflammatory bowel disease[J]. Gut, 2006, 55(12): 1760–1767. DOI: 10.1136/gut.2005.078824 |
| [19] | LI X Q, ZHU Y H, ZHANG H F, et al. Risks associated with high-dose Lactobacillus rhamnosus in an Escherichia coli model of piglet diarrhoea:intestinal microbiota and immune imbalances[J]. PLoS One, 2012, 7(7): e40666. DOI: 10.1371/journal.pone.0040666 |
| [20] | BAUER E, WILLIAMS B A, SMIDT H, et al. Influence of the gastrointestinal microbiota on development of the immune system in young animals[J]. Current Issues in Intestinal Microbiology, 2006, 7(2): 35–51. |
| [21] | HAO W L, LEE Y K. Microflora of the gastrointestinal tract:a review[J]. Methods in Molecular Biology, 2004, 268: 491–502. |
| [22] | SCHIFFRIN E J, BLUM S. Interactions between the microbiota and the intestinal mucosa[J]. European Journal of Clinical Nutrition, 2002, 56(Suppl.3): S60–S64. |
| [23] | MAY K D.Granulated lysozyme as an alternative to antibiotics and the use of rice based diets to improve growth performance and immune response in young pigs[D]. Master thesis.Arkansas:University of Arkansas, 2011. |
| [24] | INOUE R, TSUKAHARA T, NAKANISHI N, et al. Development of the intestinal microbiota in the piglet[J]. The Journal of general and applied micribiology, 2005, 51(4): 257–265. DOI: 10.2323/jgam.51.257 |
| [25] | ROTHKOTTER H J, PABST R. Lymphocyte subsets in jejunal and ileal Peyer's patches of normal and gnotobiotic minipigs[J]. Immunology, 1989, 67(1): 103–108. |
| [26] | ROTHKÖTTER H J, KIRCHHOFF T, PABST R. Lymphoid and non-lymphoid cells in the epithelium and lamina propria of intestinal mucosa of pigs[J]. Gut, 1994, 35(11): 1582–1589. DOI: 10.1136/gut.35.11.1582 |
| [27] | ROTHKÖTTER H J, MÖLLHOFF S, PABST R. The influence of age and breeding conditions on the number and proliferation of intraepithelial lymphocytes in pigs[J]. Scandinavian Journal of Immunology, 1999, 50(1): 31–38. DOI: 10.1046/j.1365-3083.1999.00557.x |
| [28] | LEY R E, BÄCKHED F, TURNBAUGH P, et al. Obesity alters gut microbial ecology[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(31): 11070–11075. DOI: 10.1073/pnas.0504978102 |
| [29] | BÄCKHED F, DING H, WANG T, et al. The gut microbiota as an environmental factor that regulates fat storage[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(44): 15718–15723. DOI: 10.1073/pnas.0407076101 |
| [30] | 郭秀兰. 猪肠道硬壁菌门和拟杆菌门数量的检测及其相对丰度与脂肪沉积的相关性研究[D]. 博士学位论文. 雅安: 四川农业大学, 2008. |
| [31] | 刘晶. 饲料果胶对瘤胃微生物菌群结构和微生物蛋白合成影响的研究[D]. 博士学位论文. 杭州: 浙江大学, 2014. |
| [32] | 刘威. 一株猪源性乳酸利用、丁酸产生菌的分离和鉴定及其体外代谢特性的初步研究[D]. 硕士学位论文. 南京: 南京农业大学, 2007. |
| [33] | MACKIE R I, AMINOV R I, HU W P, et al. Ecology of uncultivated Oscillospira species in the rumen of cattle, sheep, and reindeer as assessed by microscopy and molecular approaches[J]. Applied and Environmental Microbiology, 2003, 69(11): 6808–6815. DOI: 10.1128/AEM.69.11.6808-6815.2003 |
| [34] | 姬玉娇, 祝倩, 耿梅梅, 等. 高、低营养水平饲粮对环江香猪结肠菌群结构及代谢物的影响[J]. 微生物学通报, 2016, 43(7) :1650–1659. |
| [35] | 仝佳. 产丙酮酸棒状杆菌的发现、生物学特性及应用性研究[D]. 博士学位论文. 镇江: 江苏大学, 2010. |
| [36] | 房正国. 不同脂肪来源的日粮对仔猪肠道发育及肠道微生物区系的影响[D]. 硕士学位论文. 南京: 南京农业大学, 2012. |
| [37] | 张路寒, 韩冶. 浅谈畜禽粪便污染的危害与处理[J]. 中国畜禽种业, 2010(6) :40–41. |




