2. 四川农业大学动物科技学院, 成都 611130
2. College of Animal Science and Technology, Sichuan Agricultural University, Chengdu 611130, China
哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)是雷帕霉素在动物体内的一种靶蛋白,而雷帕霉素是一种吸水链霉菌产出的大环内酯类化合物。20世纪70年代,Vézina等[1]在智利的复活节岛的土壤中分离出这种细菌并报道了雷帕霉素。1991年Heitman等筛选出抗雷帕霉素的啤酒酵母突变株,对比发现了3个与此抗性有关的基因,其中2个基因以其发现地建筑Spalentor命名为雷帕霉素靶蛋白1(TOR1)和雷帕霉素靶蛋白2(TOR2)[2-3]。1994年,Sabatini首次在哺乳动物——大鼠中发现了与雷帕霉素结合的蛋白质,而编码该蛋白质的基因与酵母TOR1和TOR2同源,该作者就将其正式命名为“mammalian target of rapamycin(mTOR)”,即哺乳动物雷帕霉素靶蛋白[4]。除哺乳动物外,mTOR在其他物种中也广泛分布,如拟南芥、果蝇和鲤鱼等[5-7]。后来研究证实,mTOR是一种结构与功能高度保守的丝氨酸/苏氨酸蛋白激酶,可通过整合来自上游的信号因子进而发挥生物学功能。由于广泛参与机体的生物学功能,mTOR现已成为生物科学相关领域的研究热点。为此,本文着重阐述了mTOR的主要生物学作用及其机制,以期为各同行在研究过程中提供一定的参考。
1 mTOR结构 1.1 mTOR的基因结构目前,已在315种真核生物中鉴定出mTOR基因。一些代表种,如酵母mTOR基因长7 413 bp,编码2 470个氨基酸残基长度的蛋白质;其旁系同源基因mTOR2长7 425 bp,编码2 474个氨基酸残基长度的蛋白质。人类mTOR基因位于1号染色体短臂(1p36.2),其开放阅读框为7 650 bp,编码2 549个氨基酸,蛋白质分子质量为288.95 ku。大鼠mTOR基因位于5号染色体长臂(5q36),开放阅读框为8 554 bp,编码2 549个氨基酸,蛋白质分子质量为288.85 ku。由此可见,mTOR基因结构十分保守,各物种的mTOR基因片段大小以及编码mTOR的氨基酸片段大小近似。
1.2 mTOR的蛋白质结构mTOR是一种非典型丝氨酸/苏氨酸蛋白激酶,属于磷脂酰肌醇激酶相关激酶(phosphatidylinositol kinase-related kinase,PIKK)家族。mTOR由多个结构域组成,从N端到C端依次是20个HEAT重复域、FAT域、FRB域、激酶域(kinase domain,KD)和C末端FAT域。HEAT域是在一些细胞质蛋白质中发现的α螺旋结构串联的模体,是4种细胞质蛋白质[亨廷顿蛋白(huntingtin)、延长因子3(elongation factor 3)、蛋白磷酸酶2A(PP2A)亚单元和雷帕霉素靶蛋白(TOR)]的简写,该区域与蛋白质间相互作用及胞内转运相关[8]。FAT域是一段PIKK家族共有的激酶域,由PIKK家族的3个蛋白质(FRAP、ATM和TRRAP)简写构成,与mTOR的活性有关。FRB域为FK506结合蛋白雷帕霉素结合域(FKBP12-rapamycin binding domain)的简写,它为mTOR与雷帕霉素提供结合的位点(图 1)[9]。
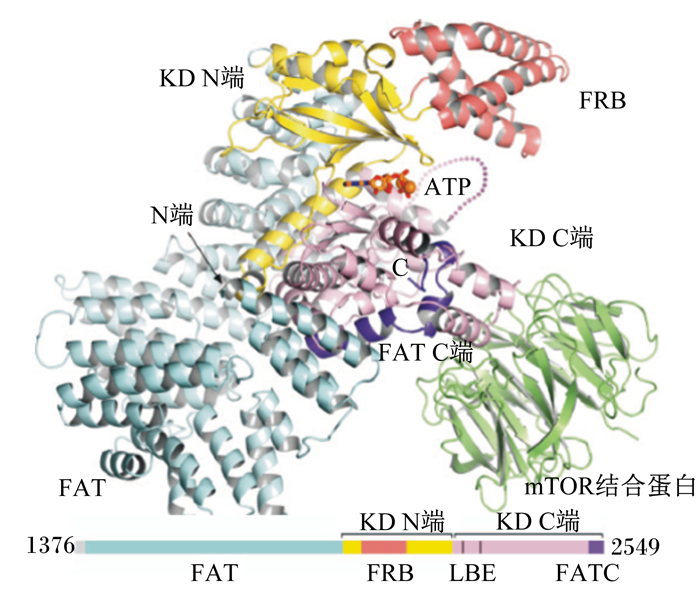
|
图 1 mTOR的蛋白质结构 Figure 1 The protein structure of mTOR[9] |
mTOR上还有4个磷酸化位点,分别是丝氨酸1261、苏氨酸2446、丝氨酸2448和丝氨酸2481位点,这些磷酸化位点与mTOR的功能密切相关。HEAT域的丝氨酸1261位点受到胰岛素信号作用而磷酸化,丝氨酸1261位点的磷酸化能进一步促进mTOR下游核糖体蛋白S6激酶(ribosomal protein S6 kinase,S6K)和真核翻译起始因子4E-结合蛋白1(eukaryotic translation initiation factor 4E-binding protein 1,4E-BP1)发生磷酸化;苏氨酸2446位点受营养物质的调控,通过腺苷酸激酶(adenosine monophosphate kinase,AMPK)途径完成磷酸化作用;丝氨酸2448位点受S6K磷酸化作用;丝氨酸2481是一个对雷帕霉素敏感且具有自发磷酸化的位点[10-11]。
2 mTOR的组织分布mTOR广泛分布于动物的中枢神经系统和外周组织。Cota等[12]利用免疫组织化学的方法研究报道了mTOR在大鼠脑中的分布情况,结果显示mTOR广泛分布在大鼠各脑区,在下丘脑区域内的室旁核以及弓状核还发现了大量磷酸化(丝氨酸2448位点)的mTOR,而下丘脑外侧区域磷酸化的mTOR较少。检测山羊各组织中mTOR mRNA水平时发现,mTOR mRNA在脑、睾丸、脾脏、肾脏和心脏中含量较高,而在肝脏和肺脏中含量较少[13]。Makky等[14]用原位杂交技术研究对比了斑马鱼mTOR的时空分布情况,结果显示斑马鱼的脑中最先表达mTOR mRNA,随后在肠道中检测到mTOR mRNA的表达。Jiang等[7]对比了鲤鱼mTOR的时空分布情况,结果显示鲤鱼的心脏、肌肉、头肾、脾脏、鳃和肠道均有mTOR mRNA表达,但表达量存在着时空差异。
3 mTOR的生物学功能 3.1 调节摄食mTOR不具有直接调控摄食的作用,而是受机体能量水平等因素作用,调控处于其下游的食欲调节因子的表达,进而参与摄食调控。已有研究表明,mTOR与中枢食欲调节因子神经肽Y(neuropeptide Y,NPY)、刺鼠相关蛋白(agouti-related protein,AgRP)、黑皮素原(proopiomelanocortin,POMC)和可卡因-安非他明调节转录肽(cocaine-and amphetamine-regulated transcript,CART)以及外周食欲调节因子瘦素(leptin)、胃饥饿素(ghrelin)和nesfatin-1(一种饱食分子蛋白)相互关联,在食欲调节中发挥重要作用。NPY[15]、AgRP[16]、POMC[17]、CART[18]神经核团均存在于下丘脑这一摄食调节中枢,发挥着增食欲或抑制食欲的作用。leptin[19]、ghrelin[20]和nesfatin-1[21]是存在于外周组织调节食欲的因子,也具有调节食欲的作用。
Cota等[12]的研究首次证实mTOR对动物摄食具有调节作用。中枢系统mTOR信号通路可过感应细胞外营养物质和激素水平调控大鼠摄食。大鼠进食后弓状核mTOR及下游靶点S6K1磷酸化水平增加;当禁食48 h后,mTOR和S6K1的磷酸化水平降低,而大鼠禁食后再喂食,使其磷酸化水平再次增加,从而进一步明确了下丘脑mTOR在机体能量感知方面的重要作用。食物中的蛋白质含量也能影响mTOR信号通路的表达,投喂高蛋白质的饲粮能促使长白猪mTOR的磷酸化[22]。此外,mTOR还可以调节其他食欲肽的表达量。研究发现,给大鼠脑室注射leptin后,下丘脑mTOR的磷酸化水平增加,而注射mTOR的抑制剂雷帕霉素后,leptin的生理效应明显减弱。此后的研究表明,mTOR对诸多食欲因子都具有相似的调节方式。mTOR信号通路介导了外周的ghrelin在中枢神经系统中的增食欲作用。Martins等发现,脑室注射ghrelin 2 h后的大鼠摄食量显著增加,同时mTOR和S6K1磷酸化水平也显著增加[23]。下丘脑中的mTOR在转导甲状腺激素信号的摄食调控功能方面也具有相似的作用机制[24],即受机体能量水平等因素作用,调控其下游的食欲调节因子的表达继而参与摄食调控。甲状腺激素是响应并调节机体能量状态重要因子,大鼠下丘脑弓状核注射甲状腺激素后增食欲因子NPY和AgRP基因的表达量显著增加,厌食因子POMC基因的表达量显著减少,大鼠摄食量增加。与此同时,下丘脑mTOR及其下游因子的磷酸化水平显著增加,而肝脏中mTOR及其下游因子的磷酸化水平显著降低。相应地,脑室注射雷帕霉素阻断mTOR信号后,增食欲因子的基因表达量不再因甲状腺激素增加而升高,大鼠体重也随之下降。mTOR在摄食调控过程中的这种信号转导作用还见于其他食欲因子的研究[25-27]。
3.2 调节脂质合成mTOR参与了脂质合成、脂质β氧化和脂质分解等脂质代谢过程。在脂质合成中,脂肪酸及甘油三酯的合成是关键环节,其间主要包括脂质合成酶以及脂质合成酶转录调控因子,如乙酰辅酶A羧化酶(acetyl CoA carboxylase,ACC)、脂肪酸合成酶(fatty acid synthase,FAS)、硬脂酰辅酶A去饱和酶(stearoyl-coenzyme A desaturase,SCD)、lipin 1、固醇调节元件结合蛋白(sterol regulatory element binding protein,SREBP)和过氧化物酶体增殖物激活受体-γ(peroxisome proliferator activated receptor-γ,PPAR-γ)等。此外,脂肪氧化分解相关酶的活性与脂质的合成效率紧密相关,如三酰甘油脂肪酶(adipose triglyceride lipase,ATGL)、激素敏感酯酶(hormone-sensitive triglyceride lipase,HSL)和单酰甘油脂肪酶(monoacylglycerol lipase,MGL)等。受上游营养因子或胰岛素等信号激发,mTOR能增加脂质合成最为关键的转录调控因子——SREBP-1c基因的表达[28],继而使机体脂质合成作用加快。2008年首次报道了mTOR具有调节SREBP-1c表达,影响脂质合成的作用。用4-羟基他莫昔芬激活蛋白激酶B(protein kinase B,PKB/AKT)处理人类视网膜色素上皮细胞4 h后,细胞核内的SREBP-1c水平显著升高。由于核内SREBP-1c能增加脂质合成相关酶基因表达,脂肪酸合成酶(fatty acid synthetase,FASN)mRNA水平也在4 h后显著增加;当用雷帕霉素阻断mTOR的信号传递后,核内SREBP-1c不再因AKT的激活而增加,FASN和ATP-柠檬酸裂合酶(ATP citrate lyase,ACLY)的mRNA水平也因此受阻[29]。
mTOR对SREBP-1c的调节是多方面的,主要是mRNA转录和蛋白质加工。在SREBP-1c的脂质合成作用研究中,进食或胰岛素刺激均能增加动物肝脏细胞SREBP-1c基因的表达量[30-31]。Owen等[32]发现大鼠肝细胞在100 nmol/L胰岛素浸润15 min后,细胞核内SREBP-1c水平达到峰值并持续至6 h;磷脂酰肌醇3-激酶(phosphoinositide 3-kinase,PI3K)、mTOR和S6K的拮抗剂:渥曼青霉素、雷帕霉素和LYS6K2均可阻断胰岛素对细胞核内SREBP-1c的作用,表明胰岛素-PI3K-mTOR-S6K通路调节SREBP-1c的蛋白质加工;然而雷帕霉素预处理30 min抑制胰岛素浸润对SREBP-1c有升高作用,LYS6K2未能降低SREBP-1c mRNA表达量。这些研究结果表明,mTOR对SREBP-1c的mRNA转录与SREBP-1c的蛋白质加工的调节通路不尽相同,转录和加工的具体调控途径仍不清楚。近来有研究表明,mTOR调控了SREBP-1c由内质网向高尔基体的转运过程。Han等[33]研究发现,抑制mTOR会导致SREBP-1c向高尔基体的转运受阻。环腺苷酸(cyclic adenosine monophosphate,cAMP)响应元件结合蛋白的共激活因子——转录激活因子2(CREB regulated transcription coactivator 2,CRTC2)基因突变会引发小鼠肝细胞中甘油三酯增多,甘油三酯合成相关基因mRNA表达量升高,高脂饲喂能进一步增强这种作用;与此同时,细胞核外未成熟的SREBP-1c没有变化,SREBP-1c mRNA表达量未出现显著差异,而细胞核内SREBP-1c增多,深入研究表明,mTOR在这一过程中发挥重要作用;mTOR受机体能量水平升高而激活,使得CRTC2的136位氨基酸残基发生磷酸化,导致CRTC2与运输蛋白质COPⅡ的亚基Sec31分离,分离的Sec31得以与Sec23结合,从而将内质网上未成熟的SREBP-1c运输至高尔基体。
3.3 调节蛋白质合成mTOR通过磷酸化下游的4E-BP1和S6K,从而刺激相关基因的转录水平提高,mTOR促进蛋白质合成的作用对于细胞的生长和增殖有重要作用[34]。4E-BP1是与真核翻译起始因子4E(eukaryotic translation initiation factor 4E, eIF4E)结合的蛋白,而eIF4E是真核翻译起始因子4F(eukaryotic translation initiation factor 4F, eIF4F)翻译起始复合物的帽子结合亚单位。真核细胞mRNA的5'端7-甲基鸟苷“帽子”是调控其转录效率、剪切形式和核外运输的结构,几乎所有真核细胞都需要eIF4F将核糖体导向mRNA的这种帽子结构,从而开启mRNA的翻译过程,而4E-BP1通过与eIF4E结合抑制翻译的起始。当mTOR信号因上游一些刺激而激活以后,4E-BP1作为其下游因子发生磷酸化,致使4E-BP1构象改变并与eIF4E脱离,mRNA翻译效率提升[33, 35]。已知一些支链氨基酸能激活mTOR信号[36]。在奶牛上的研究还发现,L-亮氨酸和L-异亮氨酸都能够增强mTOR和S6K1的磷酸化水平及促进乳腺细胞中的蛋白质合成[37]。新生仔猪禁食24 h后,分别灌喂低蛋白质(LP组)、低蛋白质+亮氨酸(LP+L组)和高蛋白质(HP组)的饲粮后,LP+L和HP组的mTOR/S6K/4E-BP1磷酸化水平及蛋白质合成水平显著高于LP组[38]。Columbus等[39]在研究亮氨酸对仔猪蛋白质合成的作用时做了类似的处理,LP+L或HP组仔猪血浆中的亮氨酸、异亮氨酸和颉氨酸含量显著增加,LP+L组的mTOR信号通路中的4E-BP1和eIF4G·eIF4E的磷酸化水平均显著高于LP组;与此同时,HP组仔猪的体重和瘦肉量均显著增加。此外,Yao等[40]发现精氨酸也可以激活mTOR信号通路,并促进仔猪肌肉蛋白质的合成和体重的增加。同样的结果在原鸡上也得到了证实,在含有精氨酸的培养液中,mTOR、4E-BP1和S6K1 mRNA表达量得到了显著的增加,且鸡肠上皮细胞的蛋白质合成增强,蛋白质分解减弱[41]。以上研究结果表明,亮氨酸等氨基酸激活了mTOR的信号通路,4E-BP1的磷酸化水平也随着增加,使得蛋白质合成相关的基因表达量增加,最终使得机体的蛋白质合成增多。
3.4 调节细胞自噬自噬是细胞内的物质成分利用溶酶体被降解过程的统称。细胞在外界因素(营养成分、缺血和缺氧、生长因子浓度等)及内在因素(代谢压力、破坏的细胞器等)的诱发下,通过对受损细胞器等大分子物质进行降解,细胞自噬为合成新的蛋白质提供所需的原料,有利于维持蛋白质代谢平衡及细胞内环境稳定。
mTOR调节自噬已有广泛的研究,其调节机制是调控ULK1-Atg13-RB1CC1-Atg101复合体的磷酸化状态。在营养丰富的条件下,mTOR磷酸化Atg13,后者在高磷酸化状态下,与Atg1的亲和力下降,使Atg1激酶活性降低,细胞自噬水平下降。相反,在饥饿条件下,mTOR活性被抑制,Atg13去磷酸化并与Atg1激酶紧密结合,导致Atg1激酶激活从而启动自噬,雷帕霉素可抑制mTOR的活性,有助于Atg13去磷酸化和Atg1活化,诱导自噬[42-43]。有研究发现,丝氨酸/苏氨酸蛋白激酶ATM的C端序列与PI3K催化区同源, 能够刺激LBK/AMPK/TSC2通路下游信号, 从而抑制mTOR。mTOR被抑制后可激活ULK1,ULK1通过与UVRAG结合再使beclin 1(丝氨酸14位点)磷酸化, 从而增强beclin 1-Vps34-Atg14L复合体的活性,启动自噬[44-45]。
3.5 mTOR调节衰老衰老是随着时间的推进在机体内出现的细胞以及组织器官的生理功能的衰退现象。一直以来,衰老都是研究的焦点,mTOR也因其参与衰老进程的调控作用而得到大量的研究。其中,2009年,首次在《Nature》上报道了mTOR信号与衰老有密切的关系。该研究发现,600日龄的雌雄小鼠饲粮中添加mTOR的抑制剂雷帕霉素,分别提高了雌雄小鼠13%和9%的寿命[46]。随后,有学者关注了mTOR下游信号与衰老之间的关系。在mTOR-S6K1信号通路中,限制能量摄入的小鼠或敲除S6K1基因的小鼠同样表现出了寿命延长,衰老相关疾病减轻[47]。以上研究表明了mTOR信号通路参与了机体衰老,但具体机制现仍未明确。
4 小结mTOR是一种结构与功能高度保守的蛋白质,是能量代谢调控的中枢传感器,在摄食调控、脂类代谢、蛋白质合成等方面都起到了重要的作用。mTOR信号通路可以在转录和翻译水平上调节许多基因的表达,若其调控机制失调将会引起机体紊乱。已有的研究报道发现,mTOR与代谢综合征、癌症等疾病的发生密切相关。因此,对mTOR生理功能的深入研究,将有助于疾病的治疗和新药的开发。目前,有关mTOR的研究主要集中在人类、小鼠上,而在其他哺乳动物上的研究还相对较少。鉴于mTOR强大的生理功能,如何通过mTOR信号通路来调节动物生产性能将是我们今后的一个研究重点。如mTOR在蛋白质和脂质合成上发挥了重要的调控作用,那么是否可通过调节mTOR信号通路来提高动物饲料的蛋白质利用率及减少动物体内脂肪沉积。这些都是未来值得我们去研究的方向,可以预期mTOR在动物生产中将有广泛的应用前景。
| [1] |
VÉZINA C, KUDELSKI A, SEHGAL S. Rapamycin (AY-22, 989), a new antifungal antibiotic:Ⅰ.taxonomy of the producing streptomycete and isolation of the active principle[J]. The Journal of Antibiotics, 1975, 28(10): 721-726. DOI:10.7164/antibiotics.28.721 |
| [2] |
HEITMAN J, MOVVA N R, HALL M N. Targets for cell cycle arrest by the immunosuppressant rapamycin in yeast[J]. Science, 1991, 253(5022): 905-909. DOI:10.1126/science.1715094 |
| [3] |
HEITMAN J. On the discovery of TOR as the target of rapamycin[J]. PLoS Pathogens, 2015, 11(11): e1005245. DOI:10.1371/journal.ppat.1005245 |
| [4] |
SABATINI D M, ERJIUMENT-BROMAGE H, LUI M, et al. RAFT1:a mammalian protein that binds to FKBP12 in a rapamycin-dependent fashion and is homologous to yeast TORs[J]. Cell, 1994, 78(1): 35-43. DOI:10.1016/0092-8674(94)90570-3 |
| [5] |
MENAND B, DESNOS T, NUSSAUME L, et al. Expression and disruption of the Arabidopsis TOR (target of rapamycin) gene[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(9): 6422-6427. DOI:10.1073/pnas.092141899 |
| [6] |
OLDHAM S, MONTAGNE J, RADIMERSKI T, et al. Genetic and biochemical characterization of dTOR, the Drosophila homolog of the target of rapamycin[J]. Genes & development, 2000, 14(21): 2689-2694. |
| [7] |
JIANG J, FENG L, LIU Y, et al. Mechanistic target of rapamycin in common carp:cDNA cloning, characterization, and tissue expression[J]. Gene, 2013, 512(2): 566-572. DOI:10.1016/j.gene.2012.08.006 |
| [8] |
ANFRADE M A, BORK P. Heat repeats in the Huntington's disease protein[J]. Nature Genetics, 1995, 11(2): 115-116. DOI:10.1038/ng1095-115 |
| [9] |
YANG H J, RUDGE D G, Koos J D, et al. mTOR kinase structure, mechanism and regulation[J]. Nature, 2013, 497(7448): 217-223. DOI:10.1038/nature12122 |
| [10] |
ACOSTA-JAQUEZ H A, KELLER J A, FOSTER K G, et al. Site-specific mTOR phosphorylation promotes mTORC1-mediated signaling and cell growth[J]. Molecular and Cellular Biology, 2009, 29(15): 4308-4324. DOI:10.1128/MCB.01665-08 |
| [11] |
WATANABE R, WEI L, HUANG J. mTOR signaling, function, novel inhibitors, and therapeutic targets[J]. Journal of Nuclear Medicine, 2011, 52(4): 497-500. |
| [12] |
COTA D, PROULX K, SMITH K A B, et al. Hypothalamic mTOR signaling regulates food intake[J]. Science, 2006, 312(5775): 927-930. DOI:10.1126/science.1124147 |
| [13] |
LIANG Y, BAO W L, CHEBRI, et al. Molecular characterization and functional analysis of cashmere goat mammalian target of rapamycin[J]. DNA and Cell Biology, 2012, 31(5): 839-844. DOI:10.1089/dna.2011.1393 |
| [14] |
MAKKY K, TEKIELA J, MAYER A N. Target of rapamycin (TOR) signaling controls epithelial morphogenesis in the vertebrate intestine[J]. Developmental Biology, 2007, 303(2): 501-513. DOI:10.1016/j.ydbio.2006.11.030 |
| [15] |
NEWMYER B A, NANDAR W, WEBSTER R I, et al. Neuropeptide Y is associated with changes in appetite-associated hypothalamic nuclei but not food intake in a hypophagic avian model[J]. Behavioural Brain Research, 2013, 236: 327-331. DOI:10.1016/j.bbr.2012.08.015 |
| [16] |
KRASHES M J, SHAH B P, KODA S, et al. Rapid versus delayed stimulation of feeding by the endogenously released AgRP neuron mediators GABA, NPY, and AgRP[J]. Cell Metabolism, 2013, 18(4): 588-595. DOI:10.1016/j.cmet.2013.09.009 |
| [17] |
OH T S, ChO H, CHO J H, et al. Hypothalamic AMPK-induced autophagy increases food intake by regulating NPY and POMC expression[J]. Autophagy, 2016, 12(11): 2009-2025. DOI:10.1080/15548627.2016.1215382 |
| [18] |
SINGH O, KUMAR S, SINGH U, et al. Cocaine-and amphetamine-regulated transcript peptide (CART) in the brain of zebra finch, Taeniopygia guttata:organization, interaction with neuropeptide Y, and response to changes in energy status[J]. The Journal of Comparative Neurology, 2016, 524(15): 3014-3041. DOI:10.1002/cne.v524.15 |
| [19] |
ZHAO S, KANOSKI S E, YAN J, et al. Hindbrain leptin and glucagon-like-peptide-1 receptor signaling interact to suppress food intake in an additive fashion[J]. International Journal of Obesity, 2012, 36(12): 1522-1528. DOI:10.1038/ijo.2011.265 |
| [20] |
SENIN L L, AL-MASSADI O, FOLGUEIRA C, et al. The gastric CB1 receptor modulates ghrelin production through the mTOR pathway to regulate food intake[J]. PLoS One, 2013, 8(11): e80339. DOI:10.1371/journal.pone.0080339 |
| [21] |
WERNECKE K, LAMPRECHT I, JOHREN O, et al. Nesfatin-1 increases energy expenditure and reduces food intake in rats[J]. Obesity, 2014, 22(7): 1662-1668. DOI:10.1002/oby.20736 |
| [22] |
LIU Y, LI F, KONG X, et al. Signaling pathways related to protein synthesis and amino acid concentration in pig skeletal muscles depend on the dietary protein level, genotype and developmental stages[J]. PLoS One, 2015, 10(9): e0138277. DOI:10.1371/journal.pone.0138277 |
| [23] |
MARTINS L, FERNÁNDEZ-MALLO D, NOVELLE M G, et al. Hypothalamic mTOR signaling mediates the orexigenic action of ghrelin[J]. PLoS One, 2012, 7(10): e46923. DOI:10.1371/journal.pone.0046923 |
| [24] |
VARELA L, MARTÍNEZ-SÁNCHEZ N, GALLEGO R, et al. Hypothalamic mTOR pathway mediates thyroid hormone-induced hyperphagia in hyperthyroidism[J]. The Journal of Pathology, 2012, 227(2): 209-222. DOI:10.1002/path.v227.2 |
| [25] |
SENIN L L, AL-MASSADI O, FOLGUEIRA C, et al. The gastric CB1 receptor modulates ghrelin production through the mTOR pathway to regulate food intake[J]. PLoS One, 2013, 8(11): e80339. DOI:10.1371/journal.pone.0080339 |
| [26] |
TOWNSEND K L, SUZUKI R, HUANG T L, et al. Bone morphogenetic protein 7(BMP7) reverses obesity and regulates appetite through a central mTOR pathway[J]. The FASEB Journal, 2012, 26(5): 2187-2196. DOI:10.1096/fj.11-199067 |
| [27] |
YANG M L, ZHANG Z H, WANG C, et al. Nesfatin-1 action in the brain increases insulin sensitivity through Akt/AMPK/TORC2 pathway in diet-induced insulin resistance[J]. Diabetes, 2012, 61(8): 1959-1968. DOI:10.2337/db11-1755 |
| [28] |
EBERLÉ D, HEGARTY B, BOSSARD P, et al. SREBP transcription factors:master regulators of lipid homeostasis[J]. Biochimie, 2004, 86(11): 839-848. DOI:10.1016/j.biochi.2004.09.018 |
| [29] |
PORSTMANN T, SANTOS C R, GRIFFITHS B, et al. SREBP activity is regulated by mTORC1 and contributes to Akt-dependent cell growth[J]. Cell Metabolism, 2008, 8(3): 224-236. DOI:10.1016/j.cmet.2008.07.007 |
| [30] |
TIAN J, GOLDSTEIN J L, BROWN M S. Insulin induction of SREBP-1c in rodent liver requires LXRα-C/EBPβ complex[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(29): 8182-8187. DOI:10.1073/pnas.1608987113 |
| [31] |
HAAS J T, MIAO J, CHANDA D, et al. Hepatic insulin signaling is required for obesity-dependent expression of SREBP-1c mRNA but not for feeding-dependent expression[J]. Cell Metabolism, 2012, 15(6): 873-884. DOI:10.1016/j.cmet.2012.05.002 |
| [32] |
OWEN J L, ZHANG Y, BAE S H, et al. Insulin stimulation of SREBP-1c processing in transgenic rat hepatocytes requires p70 S6-kinase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(40): 16184-16189. DOI:10.1073/pnas.1213343109 |
| [33] |
HAN J B, LI E W, CHEN L Q, et al. The CREB coactivator CRTC2 controls hepatic lipid metabolism by regulating SREBP1[J]. Nature, 2015, 524(7564): 243-246. DOI:10.1038/nature14557 |
| [34] |
LAPLANTE M, SABATINI D M. mTOR signaling at a glance[J]. Journal of Cell Science, 2009, 122(20): 3589-3594. DOI:10.1242/jcs.051011 |
| [35] |
YANAGIYA A, SUYAMA E, ADACHI H, et al. Translational homeostasis via the mRNA cap-binding protein, eIF4E[J]. Molecular Cell, 2012, 46(6): 847-858. DOI:10.1016/j.molcel.2012.04.004 |
| [36] |
JEWELL J L, RUSSELL R C, GUAN K L. Amino acid signalling upstream of mTOR[J]. Nature Reviews Molecular Cell Biology, 2013, 14(3): 133-139. DOI:10.1038/nrm3522 |
| [37] |
ZHANG X, ZHAO F, SI Y, et al. GSK3β regulates milk synthesis in and proliferation of dairy cow mammary epithelial cells via the mTOR/S6K1 signaling pathway[J]. Molecules, 2014, 19(7): 9435-9452. |
| [38] |
TORRAZZA R M, SURYAWAN A, GAZZANEO M C, et al. Leucine supplementation of a low-protein meal increases skeletal muscle and visceral tissue protein synthesis in neonatal pigs by stimulating mTOR-dependent translation initiation[J]. The Journal of Nutrition, 2010, 140(12): 2145-2152. DOI:10.3945/jn.110.128421 |
| [39] |
COLUMBUS D A, STEINHOFF-WAGNER J, SURYAWAN A, et al. Impact of prolonged leucine supplementation on protein synthesis and lean growth in neonatal pigs[J]. American Journal of Physiology-Endocrinology and Metabolism, 2015, 309(6): E601-E610. DOI:10.1152/ajpendo.00089.2015 |
| [40] |
YAO K, YIN Y L, CHU W Y, et al. Dietary arginine supplementation increases mTOR signaling activity in skeletal muscle of neonatal pigs[J]. The Journal of Nutrition, 2008, 138(5): 867-872. DOI:10.1093/jn/138.5.867 |
| [41] |
YUAN C, DING Y, HE Q, et al. L-arginine upregulates the gene expression of target of rapamycin signaling pathway and stimulates protein synthesis in chicken intestinal epithelial cells[J]. Poultry Science, 2015, 94(5): 1043-1051. DOI:10.3382/ps/pev051 |
| [42] |
HOSOKAWA N, HARA T, KAIZUKA T, et al. Nutrient-dependent mTORC1 association with the ULK1-Atg13-FIP200 complex required for autophagy[J]. Molecular Biology of the Cell, 2009, 20(7): 1981-1991. DOI:10.1091/mbc.E08-12-1248 |
| [43] |
JUNG C H, JUN C B, RO S H, et al. ULK-Atg13-FIP200 complexes mediate mTOR signaling to the autophagy machinery[J]. Molecular Biology of the Cell, 2009, 20(7): 1992-2003. DOI:10.1091/mbc.E08-12-1249 |
| [44] |
MANZONI C, MAMAIS A, ROOSEN D A, et al. mTOR independent regulation of macroautophagy by leucine rich repeat kinase 2 via Beclin-1[J]. Scientific Reports, 2016, 6: 35106. DOI:10.1038/srep35106 |
| [45] |
RUSSELL R C, TIAN Y, YUAN H X, et al. ULK1 induces autophagy by phosphorylating Beclin-1 and activating VPS34 lipid kinase[J]. Nature Cell Biology, 2013, 15(7): 741-750. DOI:10.1038/ncb2757 |
| [46] |
HARRISON D E, STRONG R, SHARP Z D, et al. Rapamycin fed late in life extends lifespan in genetically heterogeneous mice[J]. Nature, 2009, 460(7253): 392-395. |
| [47] |
SELMAN C, TULLET J M, WIESER D, et al. Ribosomal protein S6 kinase 1 signaling regulates mammalian life span[J]. Science, 2009, 326(5949): 140-144. DOI:10.1126/science.1177221 |




