乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACC)是脂肪酸合成的限速酶,它以生物素为辅酶,催化乙酰辅酶A生成丙二酸单酰辅酶A(malonyl CoA, MA),为脂肪酸的合成提供底物[1-2]。原核生物ACC由3个基因编码的亚基组成,它们分别是生物素羧化酶(biotin carboxylase, BC)、生物素羧基载体蛋白(biotin carboxyl carrier protein, BCCP)和羧基转移酶(Carboxyltransferase, CT),真核生物ACC由1个基因编码,并同时具有上述3个结构域[3]。目前ACC有2种亚型,即ACC1(或ACCα)和ACC2(或ACCβ),分别由ACACA和ACACB基因编码。ACC 2种亚型的组织分布不同,ACC1主要在脂肪生成活跃的组织如肝脏、脂肪和乳腺中表达,为脂肪酸合成提供原料,而ACC2主要在脂肪分解活跃的组织如肌肉和心脏中表达,通过调节肉碱棕榈酰转移酶-Ⅰ(carnitine palmitoyltransferase-Ⅰ, CPT-Ⅰ)发挥生物学功能[4]。在恒温动物中,已经克隆出鼠[5]、牛[6]等ACC1基因和人[7]ACC2基因的全长cDNA,人的ACC2基因含1个长度为7 449 bp的开放阅读框,编码2 483个氨基酸,定位在染色体12q23上[7]。ACC2与ACC1的氨基酸序列的相似性高达75%,两者的主要区别在N端,哺乳动物的ACC2比ACC1多约140个氨基酸[8-9],多出区域主要作用是将ACC2定位在线粒体外膜上。关于鱼类ACC基因的研究较少,目前已经克隆出草鱼(Ctenopharyngodon idella)ACC1基因全长cDNA序列[10]。
草鱼属鲤形目鲤科,因其生长迅速、肉味鲜美、饲料来源广和经济效益好而成为我国优良养殖鱼类。由于规模化养殖的不断扩大,鱼类出现肝脂质过度蓄积,所引起的代谢紊乱等问题也日趋严重。随着分子生物学技术的快速发展,从分子层面上探究营养调控的机理是营养学研究的新趋势。本试验拟克隆草鱼ACC2基因全长cDNA序列,分析其在不同组织中表达情况,研究饲喂不同脂肪源饲料后草鱼肌肉和肝胰脏中及饥饿再投喂后草鱼肝胰脏中ACC2 mRNA的表达变化,探讨该基因在草鱼脂肪代谢调控中的作用,为优化草鱼饲料配方提供参考,同时为解决养殖鱼类营养性脂肪肝等问题提供理论支撑。
1 材料与方法 1.1 草鱼ACC2基因全长cDNA分子克隆 1.1.1 试验用鱼试验用鱼购自连云港市赣榆区欢墩渔场,规格为(56.4±1.8) g/尾。
1.1.2 试验引物本试验所用引物见表 1。本试验共设计了3对ACC2实时荧光定量PCR引物,候选内参基因β-肌动蛋白(ACTB)、eEF1A、甘油醛-3-磷酸脱氢酶(GAPDH)1、GAPDH2、RPL13A和TUBB2分别设计2对引物,筛选扩增效率在0.90~1.05的引物为最终定量引物。
|
|
表 1 试验所用引物序列及预期产物大小 Table 1 Primer sequences and expected product sizes for the experiment |
取6尾喂食后6 h的草鱼活体解剖,快速分离肝胰脏、脾脏、肾脏、前肠、中肠、后肠、肠系膜脂肪、大脑、白肌和心脏,液氮研磨,采用QIAGEN公司的RNA提取试剂盒(RNeasy Lipid Tissue Mini Kit)按推荐的方法提取各个组织的总RNA,1%的琼脂糖凝胶电泳检测RNA完整性,核酸蛋白定量仪测定RNA的浓度。采用QIAGEN反转录试剂盒,以Oligo(dT)16AP为引物,按推荐的方法去除DNA污染并反转录合成cDNA第1链。
1.1.4 ACC2基因cDNA的克隆根据斑马鱼(Danio rerio)(XM_009301377)和草鱼已扩增序列,设计4对简并引物和4对特异性引物,PCR扩增ACC2基因8段核心序列,25 μL反应体系如下:TaKaRa公司的Sapphire Amp Fast PCR Master Mix 12.5 μL,肝胰脏和心脏2种组织混合cDNA第1链1 μL,上、下游引物各0.5 μL,双蒸水(ddH2O)10.5 μL。扩增条件为:94 ℃变性40 s、52 ℃退火30 s、72 ℃延伸1 min,共35个循环,反应前95 ℃预变性3 min,反应后72 ℃充分延伸7 min。1%的琼脂糖凝胶电泳检测,切胶回收,克隆测序。
1.1.5 草鱼ACC2基因3′和5′cDNA末端快速扩增(RACE)根据ACC2基因核心序列设计3′和5′RACE特异性引物(表 1)。按照Cheng等[10]的方法克隆3′和5′末端序列。
1.1.6 序列分析用DNAstar 7.1软件包中SeqMan软件将核心序列及3′和5′末端序列进行组装,得到ACC2基因全长cDNA序列。用EditSeq对序列进行编辑和分析,寻找开放阅读框,并翻译成氨基酸序列。通过http://www.cbs.dtu.dk/services/SignalP/预测信号肽,通过http://smart.embl-heidelberg.de/分析蛋白质的结构域。
1.2 草鱼ACC2基因组织表达试验用鱼购自连云港市赣榆区欢墩渔场,规格为(56.4±1.8) g/尾。取6尾草鱼活体解剖,快速分离肝胰脏、脾脏、肾脏、前肠、中肠、后肠、肠系膜脂肪、大脑、白肌和心脏,液氮研磨,提取各个组织的总RNA,以随机引物按推荐的方法去除DNA污染并反转录合成cDNA第1链,采用SYBR Green RT-qPCR方法,反应在Step One Plus PCR仪(ABI)上进行。采用QIAGEN的QuantiNova SYBR Green PCR Kit定量试剂盒,每个样品设置3个重复,候选内参基因分别为ACTB、eEF1A、GAPDH1、GAPDH2、RPL13A和TUBB2,利用软件GenEx 6.0.1中geNorm算法筛选内参基因,对草鱼ACC2基因组织表达进行定量分析。
1.3 不同脂肪源的饲料对草鱼ACC2基因表达的影响 1.3.1 试验饲料试验共设3组,3组饲料的脂肪源分别为5%鱼油(鱼油组)、5%猪油(猪油组)和5%豆油(豆油组),饲料中其他组分完全相同(表 2)。制作饲料的鱼粉、鱼油和预混料等主要原料由岳泰集团提供。称量各种原料,充分混匀10 min,每千克原料加水500 mL,揉拌15 min,利用便携式绞肉机进行制粒,放入干燥箱内55 ℃烘3 h,室温放置1 h,装袋密封保存,编号,待用。
|
|
表 2 试验饲料组成及营养水平(干物质基础) Table 2 Composition and nutrient levels of experimental diets (DM basis) |
饲养试验在单循环可控试验生态水槽系统的9个水缸内完成,每个水缸体积240 L。试验用鱼购自连云港市赣榆区欢墩渔场,共180尾,规格为(56.4±1.8) g/尾,随机分为3组,每组3个重复,每个水缸20尾,养殖周期12周,养殖水温25~28 ℃。放养后用商品饲料喂养1周,驯化草鱼上浮抢食,然后逐步换用试验饲料。试验期间,每天08:00、11:00、14:00和17:00各投饲1次,初期根据试验鱼的初始体重,按2%的日投饵率确定日投饵量为25 g/缸,如当日各缸饲料有剩余,称重扣除,根据各缸草鱼的吃食情况及时调整投饵量,每天草鱼的采食量和采食情况及时记录,后期日投饵量增加到35 g/缸。
1.3.3 不同脂肪源饲料对草鱼ACC2基因表达的影响饲养试验结束后,统计各组剩余鱼尾数,逐尾称重。每缸取3尾,每个组共取9尾解剖,取内脏称重,计算脏体比,取肝胰脏和肌肉提取总RNA,用于ACC2基因表达定量研究。
1.3.4 饥饿再投喂对草鱼ACC2基因表达的影响饲养试验结束后,禁食24 h后再次投喂,分别在投喂后3、6、12和24 h时每缸取3尾,每个组共取9尾解剖,取肝胰脏,用于总RNA提取和ACC2基因表达定量研究。
1.3.5 相关指标计算公式

|
采用软件GenEx 6.0.1中geNorm算法,筛选内参基因,采用2-ΔΔCt法计算ACC2基因的相对表达量,结果采用平均值±标准差表示。采用SPSS 17.0软件统计分析,各组数据均通过了正态性和方差齐性检验,因此,组间差异采用单因素方差分析(one-way ANOVA),组间多重比较采用最小显著差数(LSD)检验,P<0.05表示差异显著。
2 结果与分析 2.1 草鱼ACC2基因全长cDNA分子特征草鱼ACC2基因cDNA全长7 533 bp,含1个7 149 bp的开放阅读框,编码2 382个氨基酸,ACC2蛋白计算分子质量为268.34 ku,等电点为6.13,命名为ACCβ-1(GenBank登录号:MF611923)。此外,由于可变剪接,还发现另外1个分子质量为267.53 ku的草鱼ACC2蛋白的同工型(isoforms),命名为ACCβ-2(GenBank登录号:MF611924),比ACCβ-1少8个氨基酸。
2.2 草鱼ACC2基因的组织表达经筛选,以RPL13A和eEF1A基因组合为内参基因,采用RT-qPCR方法对草鱼ACC2基因在肝胰脏、脾脏、肾脏、前肠、中肠、后肠、肠系膜脂肪、大脑、白肌和心脏等组织中的表达进行了研究。结果表明,ACC2基因在所有检测组织中均有表达,ACC2 mRNA的相对表达量在肌肉中最高,为29.13;在大脑、前肠和心脏中次之,分别为19.45、15.33和14.40;在肝胰脏中最低,仅为1.90。肌肉中ACC2 mRNA的相对表达量与大脑、前肠和心脏差异不显著(P>0.05),但显著高于其他组织(P<0.05,图 1)
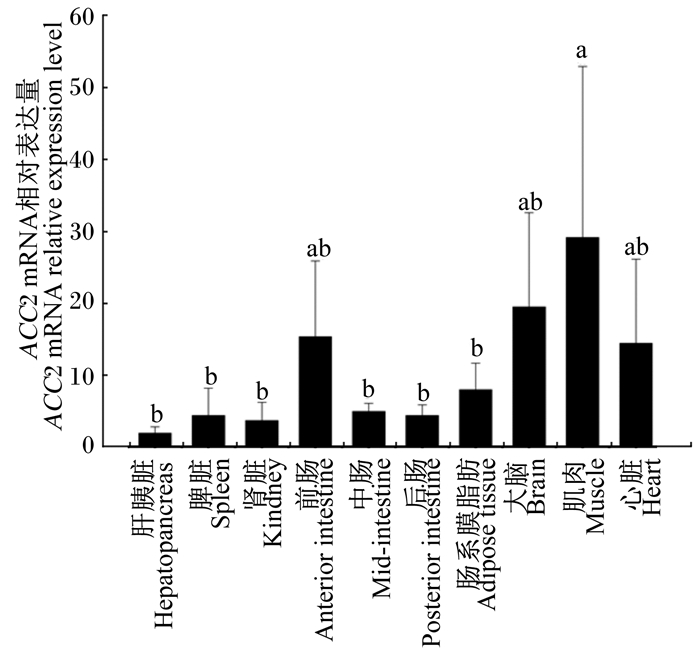
|
数据柱上标注不同字母表示差异显著(P<0.05)。下图同。 Value columns with different letters mean significant difference (P < 0.05). The same as below. 图 1 ACC2基因在草鱼不同组织中的表达分析 Figure 1 Expression analysis of ACC2 gene in different tissues of grass carp |
饲养试验结束后对草鱼的生长情况进行测量,计算增重率、饲料系数和脏体比等生长指标。由表 3可以看出,鱼油组和豆油组增重率分别为139.6%和138.8%,显著高于猪油组(P<0.05),猪油组的增重率只有128.8%,说明鱼油和豆油是草鱼良好的脂肪源,猪油效果较差;鱼油组和豆油组的饲料系数分别为1.14和1.17,显著低于猪油组(P<0.05),表明猪油不适合单独作为草鱼的脂肪源。
|
|
表 3 不同脂肪源饲料对草鱼生长指标的影响 Table 3 Effects of diets with different lipid sources on growth indexes of grass carp |
将6个内参基因分别和不同脂肪源饲料组以及饥饿再投喂后ACC2的Ct值和扩增效率输入软件,最后软件自动筛选,RPL13A和GAPDH2为不同脂肪源肝胰脏及肌肉中目的基因表达的内参基因,ACTB和GAPDH1为饥饿再投喂目的基因表达的内参基因。结果显示,投喂不同脂肪源饲料对草鱼肝胰脏及肌肉中ACC2 mRNA的相对表达量均无显著影响(P>0.05,图 2);饥饿再投喂后,草鱼肝胰脏中ACC2 mRNA的相对表达量在投喂后12 h时显著升高(P<0.05),且达到高峰值6.17,之后出现显著下降(P<0.05),24 h时仅为2.84(图 3)。
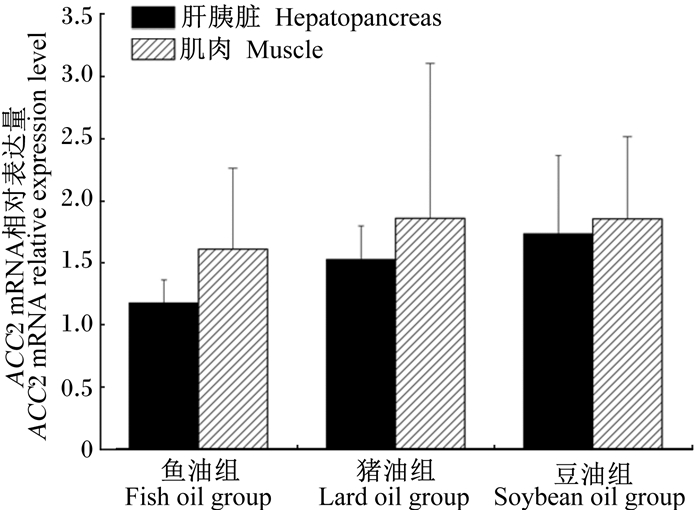
|
图 2 投喂不同脂肪源饲料的草鱼肝胰脏和肌肉中ACC2 mRNA的相对表达量 Figure 2 Relative expression level of ACC2 mRNA in hepatopancreas and muscle of grass carp fed diets with different lipid sources |
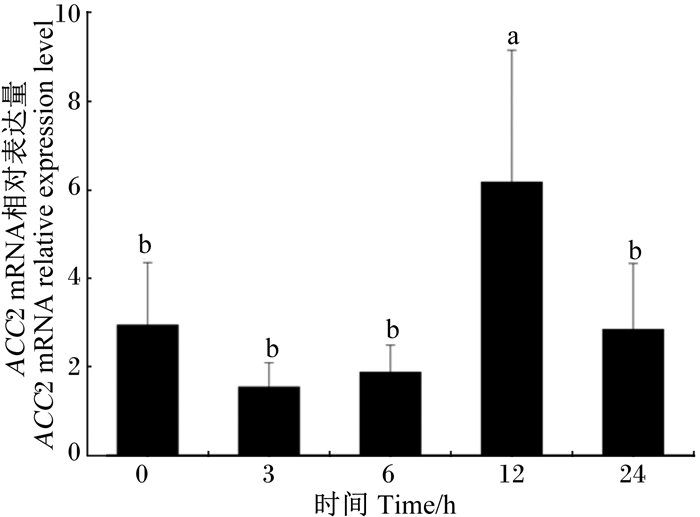
|
图 3 饥饿再投喂后草鱼肝胰脏ACC2 mRNA的相对表达量 Figure 3 Relative expression level of ACC2 mRNA in hepatopancreas of grass carp after starvation and refeeding |
对草鱼ACC2氨基酸功能性结构域进行了分析,其结果见图 4。
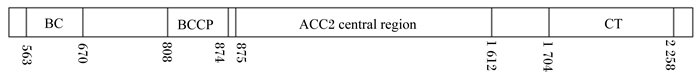
|
BC:生物素羧化酶biotin carboxylase;BCCP:生物素羧基载体蛋白biotin carboxyl carrier protein;CT:羧基转移酶carboxyltransferase;ACC2 central region:ACC2中心区域。 图 4 草鱼ACC2蛋白的3个结构域 Figure 4 Three domains of ACC2 protein in grass carp |
由图 4可知,草鱼ACC2由1个基因编码,具有3个结构域,分别为BC、BCCP和CT,它们组成ACC2的3个亚基。甘氨酸富集区(GGGGKG)被认为是ATP的结合域[11],草鱼ACC2的BC域包含1个ATP结合位点是Gly371~376,与草鱼[10]ACC1、半滑舌鳎[12]和人类[7, 13]ATP结合域完全相同,说明这段序列高度保守。保守的Met-Lys-Met序列被认为是生物素结合位点[14]。草鱼ACC2的BCCP域有2个4肽序列Val-Met-Lys-Met和Arg-Met-Lys-Met分别位于氨基酸残基的第840~843位点和第1 698~1 701位点。相关研究表明生物素会上调ACC2基因在下丘脑的表达,对小鼠给予过量生物素会抑制其食物摄入量[15]。
3.2 草鱼ACC2基因的组织表达草鱼ACC2基因在检测的10种组织中均有表达,其中肌肉中相对表达量最高,肝胰脏中相对表达量最低,白肌中的相对表达量是肝胰脏中的15.3倍(图 1)。据Abu-Elheiga等[7]报道,ACC2基因主要在人的心脏和骨骼肌有较高的表达量。而相关研究也表明,大鼠ACC2主要在骨骼肌和心脏等氧化组织中表达[4]。上述研究结果与本试验类似,说明草鱼与其他哺乳动物一样,其ACC2基因主要是在肌肉中表达。由于ACC2的产物MA是CPT-Ⅰ的有效抑制剂,ACC2基因缺陷的哺乳动物其心脏和骨骼肌中MA的水平明显下降,其他的研究也证实ACC2的活性决定心脏中MA的水平,进一步说明ACC2基因可以调控骨骼肌和心脏中脂肪酸的氧化[16-17]。
3.3 不同脂肪源饲料对草鱼生长指标的影响本试验中,各组草鱼脏体比差异不显著,可能是由于饲料脂肪水平较低引起的;鱼油组及豆油组的增重率显著高于猪油组,饲料系数则是猪油组显著高于鱼油组和豆油组,说明鱼油和豆油的促生长效果较好且两者无显著差异,它们是草鱼良好的脂肪源,猪油不适合单独作为草鱼的脂肪源。Zhang等[18]研究发现,以鱼油、豆油和棕榈油为脂肪源分别饲养牛蛙,它们的增重率均较高且三者间无显著差异,但均显著高于以家禽脂肪和猪油为脂肪源的牛蛙。陈涛等[19]在饲料中分别添加4%猪油、4%豆油、4%豆油-鱼油混合物和4%鱼油为脂肪源饲喂红罗非鱼稚鱼60 d,结果发现,豆油组和豆油+鱼油混合物组红罗非鱼稚鱼的末均体重和增重率显著高于猪油组,猪油组饲料系数显著高于其他3组,豆油组鱼体的生长性能最好,肝体比在各组间无显著性差异,由此表明豆油是红罗非鱼稚鱼良好的脂肪源。成永旭等[20]分别采用豆油和猪油作为草鱼脂肪源,结果发现猪油组草鱼肝胰脏和肌肉中脂肪含量最高,肠系膜脂肪积累也最多,且肝胰脏的形态已有脂肪肝病变倾向,而豆油组草鱼肝胰脏形态正常,说明以豆油为草鱼脂肪源优于猪油。刘玮等[21]分别以鱼肝油、豆油、菜油、猪油和混合油为脂肪源配制饲料饲喂草鱼,结果发现混合油组及鱼肝油组草鱼生长效果最好,相对增重率和饲料系数最低,豆油组及猪油组的生长状况次之,上述研究与本试验结果基本一致。
3.4 不同脂肪源饲料对草鱼肝胰脏和肌肉中及饥饿再投喂对草鱼肝胰脏中ACC2基因表达的影响本试验以鱼油、豆油和猪油为脂肪源,饲养草鱼12周,探究ACC2基因在草鱼肝胰脏和肌肉中的表达变化,结果发现,各组草鱼肝胰脏及肌肉ACC2 mRNA的相对表达量均无显著差异(图 2),说明ACC2在草鱼体内表达是相对稳定的,不受饲料脂肪种类的影响。到目前为止,分析鱼类在投喂不同脂肪源饲料后体内ACC2 mRNA相对表达量变化的研究相对较少。据Olson等[22]研究发现,敲除小鼠骨骼肌中ACC2基因后对其体重、食物摄入量以及体组成没有产生显著影响,同时发现小鼠骨骼肌中丙二酰辅酶A总量和脂肪酸氧化率也没有发生显著变化,这说明小鼠体内发生了代谢补偿,ACC2基因缺失对能量平衡的影响较小。对雄性大鼠的研究发现其骨骼肌脂肪酸氧化与ACC2基因的磷酸化没有直接的相关性[23]。Torstensen等[24]以毛鳞鱼油、棕榈油、葵花籽油和混合油(毛鳞鱼油:葵花籽油=1:1)为脂肪源饲喂大西洋鲑,发现不同脂肪源饲料对鲑鱼肌肉和肝脏中脂肪酸β氧化及鲑鱼的生长没有产生显著影响,从侧面验证了本试验中不同脂肪源饲料对草鱼肌肉中ACC2基因表达无显著影响的结果。本实验室前期研究发现,饲喂不同脂肪源饲料对草鱼肝胰脏中ACC1基因表达也无显著影响[10]。
本研究中,在饥饿再投喂12 h后草鱼肝胰脏中ACC2 mRNA的相对表达量显著增加,喂食24 h后ACC2 mRNA的的相对表达量又显著下降(图 3)。Oh等[25]研究发现大鼠肝脏中ACC2 mRNA的相对表达量在喂食后12 h时出现显著增加,再投喂24 h后也显著增加,这些结果说明大鼠ACC2基因的表达在转录水平受调控。上述研究与本试验结果存在部分差异,可能是由于物种不同造成的。据Ryu等[26]报道,饥饿再投喂高脂饲粮后小鼠肝脏中ACC1 mRNA的相对表达量显著升高。相关研究也发现鸡在饥饿再投喂2 h后,其肝脏中ACC1和脂肪酸合成酶基因mRNA的相对表达量显著上升,而与脂肪酸氧化相关基因CPT-Ⅰ mRNA的相对表达量显著下降[27],而本试验中草鱼肝胰脏中ACC2 mRNA的相对表达量在饥饿再投喂0~6 h内无显著差异,说明在此期间草鱼肝胰脏中脂肪酸的合成占主导地位。
4 结论① 本试验从草鱼肝胰脏和心脏混合组织中克隆出了ACC2基因全长cDNA,主要的功能位点ATP结合位点、生物素结合位点与其他脊椎动物相比基本保守。
② 草鱼ACC2基因主要在肌肉等脂肪分解活跃的组织中表达。
③ 投喂不同脂肪源饲料对草鱼肝胰脏中ACC2 mRNA的相对表达量无显著影响。
④ 饥饿再投喂后,肝胰脏中ACC2 mRNA的相对表达量在投喂12 h后最高。
| [1] |
ABU-ELHEIGA L, BRINKEY W R, ZHONG L, et al. The subcellular localization of acetyl-CoA carboxylase 2[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(4): 1444-1449. DOI:10.1073/pnas.97.4.1444 |
| [2] |
HARDIE D G, PAN D A. Regulation of fatty acid synthesis and oxidation by the AMP-activated protein kinase[J]. Biochemical Society Transactions, 2002, 30(6): 1064-1070. |
| [3] |
BARBER M C, PRICE N T, TRAVERS M T. Structure and regulation of acetyl-CoA carboxylase genes of metazoa[J]. Biochimica et Biophysica Acta:Molecular & Cell Biology of Lipids, 2005, 1733(1): 1-28. |
| [4] |
KREUZ S, SCHOELCH C, THOMAS L, et al. Acetyl-CoA carboxylases 1 and 2 show distinct expression patterns in rats and humans and alterations in obesity and diabetes[J]. Diabetes/Metabolism Research and Reviews, 2009, 25(6): 577-586. |
| [5] |
LOPEZ-CASILLAS F, BAI D H, LUO X C, et al. Structure of the coding sequence and primary amino acid sequence of acetyl-coenzyme A carboxylase[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(16): 5784-5788. |
| [6] |
MAO J Q, MARCOS S, DAVIS S K, et al. Genomic distribution of three promoters of the bovine gene encoding acetyl-CoA carboxylase alpha and evidence that the nutritionally regulated promoter Ⅰ contains a repressive element different from that in rat[J]. The Biochemical Journal, 2001, 358(1): 127-135. |
| [7] |
ABU-ELHEIGA L, ALMARZAl-ORTEGA D B, BALDINI A, et al. Human acetyl-CoA carboxylase 2.Molecular cloning, characterization, chromosomal mapping, and evidence for two isoforms[J]. Journal of Biological Chemistry, 1997, 272(16): 10669-10677. DOI:10.1074/jbc.272.16.10669 |
| [8] |
ABU-ELHEIGA L, MATZUK M M, ABO-HASHEMA K A, et al. Continuous fatty acid oxidation and reduced fat storage in mice lacking acetyl-CoA carboxylase 2[J]. Science, 2001, 291(5513): 2613-2616. DOI:10.1126/science.1056843 |
| [9] |
KIM K W, YAMANE H, ZONDLO J, et al. Expression, purification, and characterization of human acetyl-CoA carboxylase 2[J]. Protein Expression and Purification, 2007, 53(1): 16-23. |
| [10] |
CHENG H L, JI N J, PENG Y X, et al. Molecular characterization and tissue-specific expression of the acetyl-CoA carboxylase alpha gene from Grass carp, Ctenopharyngodon idella[J]. Gene, 2011, 487(1): 46-51. |
| [11] |
KONDO H, SHIRATSUCHI K, YOSHIMOTO T, et al. Acetyl-CoA carboxylase from Escherichia coli:gene organization and nucleotide sequence of the biotin carboxylase subunit[J]. Proceedings of the National Academy of Sciences of the United States of America, 1991, 88(21): 9730-9733. DOI:10.1073/pnas.88.21.9730 |
| [12] |
张夏青, 许建和, 潘茜, 等. 半滑舌鳎乙酰辅酶A羧化酶α基因全长cDNA分子克隆及饲料脂肪水平对其在肝脏中表达的影响[J]. 动物营养学报, 2016, 28(2): 485-497. |
| [13] |
ABU-ELHEIGA L, JAYAKUMAR A, BALDINI A, et al. Human acetyl-CoA carboxylase:characterization, molecular cloning, and evidence for two isoforms[J]. Proceedings of the National Academy of Sciences of the United States of America, 1995, 92(9): 4011-4015. |
| [14] |
TAKAI T, WADA K, TANABE T. Primary structure of the biotin-binding site of chicken liver acetyl-CoA carboxylase[J]. FEBS Letters, 1987, 212(1): 98-102. |
| [15] |
SONE H, KAMIYAMA S, HIGUCHI M, et al. Biotin augments acetyl CoA carboxylase 2 gene expression in the hypothalamus, leading to the suppression of food intake in mice[J]. Biochemical and Biophysical Research Communications, 2016, 476(3): 134-139. DOI:10.1016/j.bbrc.2016.04.152 |
| [16] |
RESZKO A E, KASUMOV T, DAVID F, et al. Regulation of malonyl-CoA concentration and turnover in the normal heart[J]. Journal of Biological Chemistry, 2004, 279(33): 34298-34301. DOI:10.1074/jbc.M405488200 |
| [17] |
TONG L. Acetyl-coenzyme A carboxylase:crucial metabolic enzyme and attractive target for drug discovery[J]. Cellular and Molecular Life Sciences, 2005, 62(16): 1784-1803. DOI:10.1007/s00018-005-5121-4 |
| [18] |
ZHANG C X, HUANG K K, LU K L, et al. Effects of different lipid sources on growth performance, body composition and lipid metabolism of bullfrog Lithobates catesbeiana[J]. Aquaculture, 2016, 457: 104-108. |
| [19] |
陈涛, 杨艳, 卢航, 等. 不同脂肪源对红罗非鱼稚鱼生长及肌肉脂肪酸组成的影响[J]. 饲料工业, 2017, 38(4): 29-35. |
| [20] |
成永旭, 王联合, 陈居明. 饲料中不同脂肪源对草鱼生长及其肌肉和肝脏脂肪含量的影响[J]. 水产科技情报, 1995(4): 171-172. |
| [21] |
刘玮, 徐萍, 任本根, 等. 不同脂肪源饲料对草鱼稚鱼生长的影响[J]. 水产学报, 1995, 19(4): 362-365. |
| [22] |
OLSON D P, PULINILKUNNIL T, CLINE G W, et al. Gene knockout of ACC2 has little effect on body weight, fat mass, or food intake[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(16): 7598-7603. DOI:10.1073/pnas.0913492107 |
| [23] |
ALKHATEEB H, HOLLOWAY G P, BONEN A. Skeletal muscle fatty acid oxidation is not directly associated with AMPK or ACC2 phosphorylation[J]. Applied Physiology, Nutrition, and Metabolism, 2011, 36(3): 361-367. |
| [24] |
TORSTENSEN B E, LIE Ø, FRØYLAND L. Lipid metabolism and tissue composition in Atlantic salmon (Salmo salar L.)-effects of capelin oil, palm oil, and oleic acid-enriched sunflower oil as dietary lipid sources[J]. Lipids, 2000, 35(6): 653-664. |
| [25] |
OH S Y, PARK S K, KIM J W, et al. Acetyl-CoA carboxylase beta gene is regulated by sterol regulatory element-binding protein-1 in liver[J]. The Journal of Biological Chemistry, 2003, 278(31): 28410-28417. DOI:10.1074/jbc.M300553200 |
| [26] |
RYU M H, SOHN H S, HEO Y R, et al. Differential regulation of hepatic gene expression by starvation versus refeeding following a high-sucrose or high-fat diet[J]. Nutrition, 2005, 21(4): 543-552. DOI:10.1016/j.nut.2005.01.001 |
| [27] |
SANEYASU T, SHIRAGAKI M, KURACHI K, et al. Effects of short-term refeeding on the expression of genes involved in lipid metabolism in chicks (Gallus gallus)[J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 2013, 166(1): 1-6. DOI:10.1016/j.cbpb.2013.06.003 |




