2. 新希望六和股份有限公司, 北京 100102;
3. 甘肃省肉羊繁育生物技术工程实验室, 民勤 733300
2. New Hope Liuhe Group Co., Ltd., Beijing 100102, China;
3. Biotechnology Engineering Laboratory of Gansu Meat Sheep Breeding, Minqin 733300, China
反刍动物的瘤胃是一个复杂的生态系统,其内生存着大量的微生物。根据微生物的分布形态,可分为液相、固相和固液混合相。而微生物中起主要消化分解作用的是细菌,主要可分为纤维素降解菌、淀粉降解菌、半纤维素降解菌、脂肪降解菌、乳酸利用菌和乳酸产生菌等[1]。
瘤胃微生物区系可受到自身采食的饲粮影响[2-3]。不同的断奶和补饲时间可以使反刍动物营养物质的摄入发生改变,影响瘤胃微生物区系和瘤胃组织的发育,进而影响饲料利用率[4-5]。另外,羔羊随着日龄的增长,饲粮采食量会发生改变,大量的外界微生物也会植入到瘤胃中,使瘤胃内的微生物区系日渐丰富。能够降解利用纤维素是羔羊反刍功能形成的标志之一,这与瘤胃内纤维素降解菌的定殖有关,因此本试验选用白色瘤胃球菌、黄色瘤胃球菌、产琥珀酸丝状杆菌、溶纤维丁酸弧菌等几种主要的纤维素降解菌和1种半纤维素降解菌布氏密螺旋体作为检测瘤胃内微生物动态变化的研究对象,另外鉴于饲粮所含淀粉对羔羊生长发育的重要性,本试验所研究的细菌还包括1种重要的淀粉降解菌牛链球菌。变性梯度凝胶电泳(DGGE)技术具有可靠性强、重现性高、方便快捷等优点[6],被广泛应用于微生物群落多样性分析中。本研究通过实时定量PCR和DGGE技术,研究断奶日龄对羔羊瘤胃内微生物多样性的影响,同时检测初生到84日龄羔羊瘤胃内微生物的动态变化,为明确断奶日龄对羔羊瘤胃微生物区系的影响提供基础数据。
1 材料与方法 1.1 试验动物与设计选取体重[(3.51±0.57) kg]接近、健康状况良好的66只湖羊公羔,在1、14和28日龄各屠宰6只羊。剩余的48只按照体重相近原则随机分为28日龄断奶组和56日龄断奶组。所有试验动物从出生后7日龄饲喂开食料,59日龄换生长羔羊颗粒料,换料过渡期为10 d。28日龄断奶组和56日龄断奶组在42、56、70、84日龄分别屠宰6只。试验中所用湖羊羔羊均购于甘肃省金昌中天羊业有限公司。试验期为羔羊1~84日龄。
1.2 样品采集动物屠宰打开瘤胃后,取出瘤胃内容物用匀浆机混匀,所有内容物过4层纱布得到瘤胃液,然后将瘤胃液装入冻存管,放入装有液氮的液氮罐中保存,带回实验室后将样品置于-80 ℃保存。
1.3 PCR分析微生物总DNA的提取采用动物粪便基因组DNA提取试剂盒(Stool DNA Kit 50,OMEGA,美国),提取按照说明书方法进行。所用引物参考Firkins等[7]、Khafipour等[8]和Ley等[9],由上海生工生物工程股份有限公司合成,引物序列见表 1。
|
|
表 1 实时定量PCR引物参数 Table 1 Parameters of primers for real-time PCR |
实时定量RCR的操作仪器为Bio-Rad CFX 96型实时定量PCR检测系统(BioRad, 美国)。以SYBR Premix Ex TaqTM试剂(北京全式金生物技术有限责任公司)建立20 μL反应体系, 包括SYBR Green Ⅰ型荧光染料10 μL、上游和下游引物各0.4 μL、总DNA模板2 μL及双蒸去离子水7.2 μL。反应条件为:95 ℃预变性10 min;95 ℃变性15 s,退火温度下退火30 s,72 ℃延伸30 s并采集突光信号,共40个循环。按仪器操作说明选择熔解曲线进行后续分析。
瘤胃微生物16S rDNA V3区的PCR扩增采用以下体系(50 μL):2 μL总DNA模板(约100 ng);GC-338f和533r引物各0.6 μL;5 μL buffer,4 μL dNTP,0.8 μL Taq聚合酶(TIANGEN Taq Polymerase);37 μL双蒸去离子水。
GC-338f引物序列:5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGACTCCTACGGGAGGCAGCAG-3′(下划线部分为GC夹子); 533r引物序列:5′-TTACCGCGGCTGCTGGCAC-3′。
PCR扩增程序如下:94 ℃ 5 min;94 ℃ 30 s, 60 ℃ 20 s,每个循环降低0.5 ℃,72 ℃30 s,10个循环;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 30 s,25个循环;72 ℃ 30 s。
依据下列公式计算目标微生物相对丰度:
微生物相对丰度(%)=2-(Ct目标菌-Ct总细菌)。
式中:Ct目标菌为以目标菌引物扩增后所测循环阈值; Ct总细菌为以总细菌引物扩增后所测循环阈值。
1.4 DGGE分析DGGE分析参照张宁[10]的方法。
1.5 聚类分析UPGAMA聚类分析参照周奕毅[11]的方法。
1.6 数据分析DGGE图谱通过Quantity-One(Bio-Rad)软件处理比较每个泳道样品间的相似性,并进行UPGAMA聚类分析[11], 另外用该软件分析DGGE图谱得到Shannon-Wiener多样性指数。数据采用SPSS 23.0软件进行单因素方差分析(one-way ANOVA),显著水平为P < 0.05。
2 结果与分析利用2%的琼脂糖凝胶电泳检测总DNA提取效果,经过分析凝胶成像系统成像图,本试验所用微生物总DNA提取成功。用提取的微生物总DNA作为模板进行PCR扩增,成功扩增出16S rDNA V3区片段(图 1)。
|
图 1 16S rDNA V3区PCR扩增 Figure 1 PCR amplification of 16S rDNA V3 region |
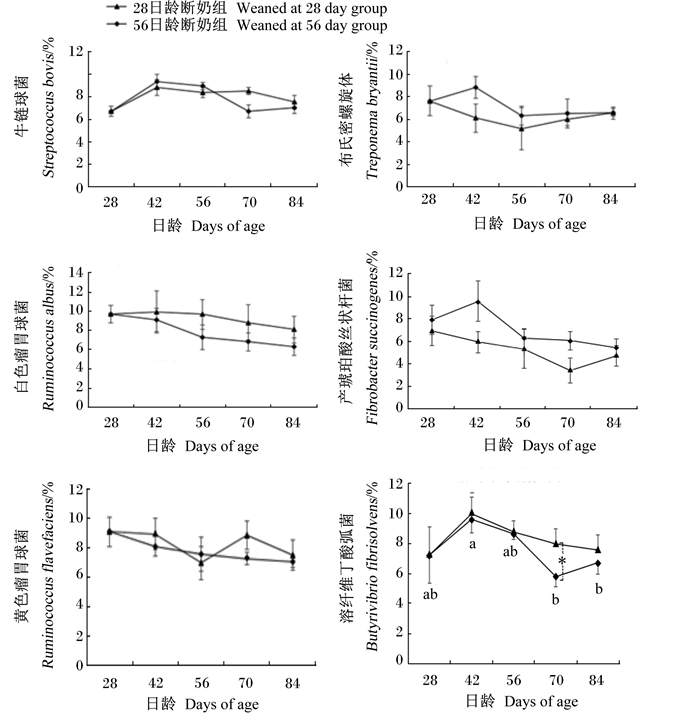
断奶日龄对瘤胃内白色瘤胃球菌、黄色瘤胃球菌、牛链球菌、产琥珀酸丝状杆菌和布氏密螺旋体相对丰度的影响见图 2。断奶日龄对白色瘤胃球菌、黄色瘤胃球菌、牛链球菌、产琥珀酸丝状杆菌和布氏密螺旋体的相对丰度无显著影响(P>0.05)。28日龄断奶组瘤胃内溶纤维丁酸弧菌相对丰度呈现28日龄较低,42日龄达到最高,之后逐渐下降的趋势。56日龄断奶组瘤胃内溶纤维丁酸弧菌相对丰度呈现28日龄较低,42日龄达到最高,之后下降,在70日龄达最低,84日龄略升高的趋势;42日龄显著高于70和84日龄(P < 0.05),其他日龄之间无显著差异(P>0.05)。70日龄时,28日龄断奶组瘤胃内溶纤维丁酸弧菌相对丰度显著高于56日龄断奶组(P < 0.05)。
|
同组数据点标注不同小写字母表示差异显著(P < 0.05);同一时间点数据点标注*表示差异显著(P < 0.05)。 Data points of the same group with different small letters mean significant difference (P < 0.05); data points of the same time point with * mean significant difference (P < 0.05). 图 2 28和56日龄断奶羔羊瘤胃纤维分解菌的相对丰度 Figure 2 Relative abundance of cellulolytic bacteria in rumen of lambs weaned at 28 and 56 days of age |
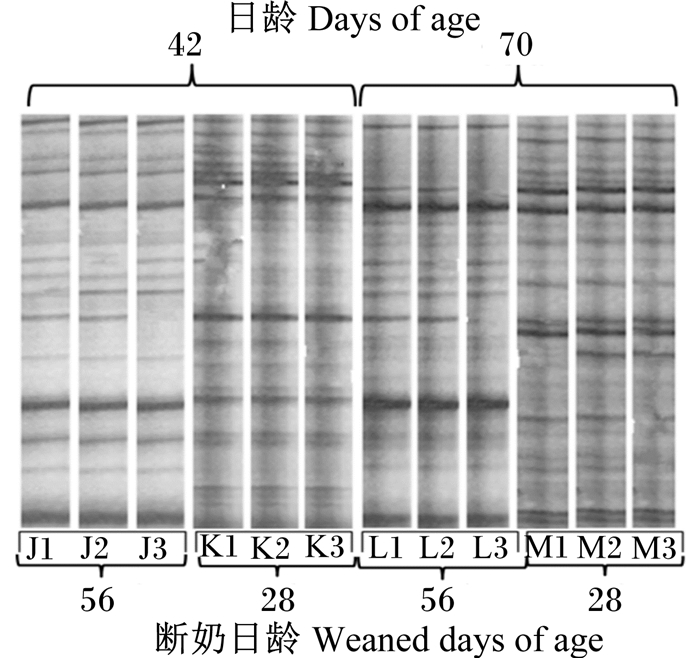
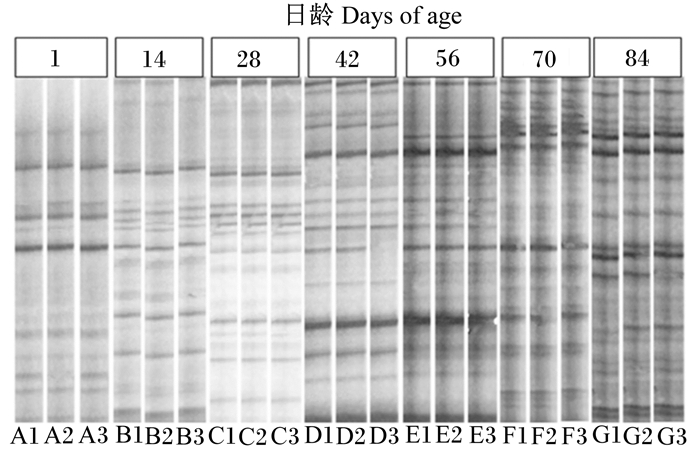
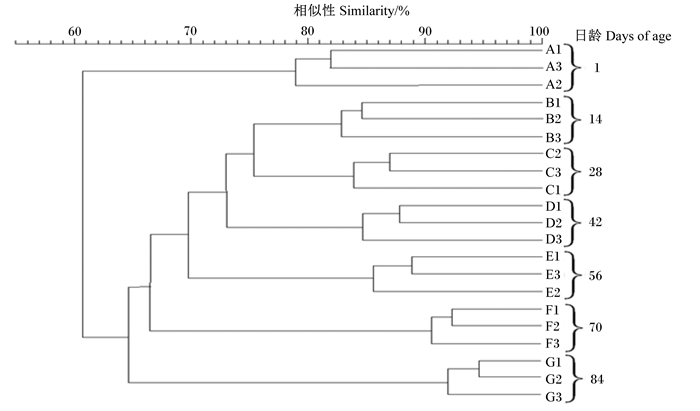
本文DGGE图谱(图 3、图 4)和聚类分析图谱(图 5)仅展示部分样品。28和56日龄断奶羔羊瘤胃微生物DGGE图谱见图 3。羔羊1~84日龄瘤胃内微生物DGGE图谱见图 4,对1~84日龄瘤胃内微生物DGGE图谱进行聚类分析,结果见图 5,可知随着日龄的增长,同一时间点的不同羔羊间的相似度逐渐升高,波动最大的为1~14日龄和56~70日龄这2个时间段。
|
图 3 28和56日龄断奶羔羊瘤胃微生物DGGE图谱 Figure 3 DGGE profiles of ruminal microbe of lambs weaned at 28 and 56 days of age |
|
图 4 羔羊1~84日龄瘤胃内微生物DGGE图谱 Figure 4 DGGE profiles of ruminal microbe of lambs at 1 to 84 days of age |
|
图 5 羔羊1~84日龄瘤胃内微生物聚类分析图谱 Figure 5 Cluster analysis map of ruminal microbe of lambs at 1 to 84 days of age |
根据DGGE图谱计算出多样性指数,结果见表 2。结果显示,在42日龄时28日龄断奶组多样性指数显著高于56日龄断奶组(P < 0.05),在70日龄时28日龄断奶组和56日龄断奶组无显著性差异(P>0.05)。从瘤胃内整个微生物区系的发育角度来看,70和84日龄的多样性指数显著高于所有其他日龄(P < 0.05),1日龄显著低于所有其他日龄(P < 0.05),其他日龄间均无显著性差异(P>0.05)。
|
|
表 2 羔羊瘤胃微生物多样性指数 Table 2 The diversity index of ruminal microbe of lambs |
本试验中,70日龄时28日龄断奶组羔羊瘤胃内溶纤维丁酸弧菌的相对丰度显著高于56日龄断奶组,56日龄断奶组日龄的变化引起瘤胃内溶纤维丁酸弧菌的相对丰度出现差异。Jami等[12]通过实时定量PCR的方法检测犊牛从出生到成年瘤胃内细菌数量的变化,发现犊牛断奶后瘤胃内溶纤维丁酸弧菌的数量下降。本课题组的另一项研究显示,56日龄断奶组在断奶后采食量升高(569.03 g/d vs. 333.83 g/d)[13],28日龄断奶组与56日龄断奶组瘤胃内溶纤维丁酸弧菌在70日龄所产生的差异可能是由于采食量的升高导致瘤胃内pH降低,不利于溶纤维丁酸弧菌生长。
柴建民[14]通过研究10、20、30日龄3个早期断奶处理的羔羊发现,跟60日龄断奶相比较,早期断奶羔羊瘤胃内微生物的Shannon指数和Simpson指数与对照组差异显著,说明断奶对瘤胃中微生物区系具有很大的影响,韩旭峰[15]采用精粗比分别为30 : 70、50 : 50、70 : 30的3种饲粮饲喂陕北白绒山羊发现,随着饲粮精粗比的升高,几种不同功能的瘤胃细菌出现了变化,纤维降解菌的含量呈显著减少的趋势。Jami等[12]检测了以色列荷斯坦牛从出生到24月龄的5个不同月龄阶段的瘤胃微生物,发现随着年龄的增长,牛瘤胃微生物的多样性和数量呈现出了显著变化。
42日龄时28日龄断奶组与56日龄断奶组条带数目和颜色深度出现差异且明显,这由于28日龄断奶组断奶后加大了开食料的采食量,因为不同饲粮颗粒上附着的微生物群落数量基本相同[16],所以开食料的采食加快了瘤胃内微生物的植入速度。70日龄时56日龄断奶组条带数目和颜色深度均高于28日龄断奶组,56日龄断奶组断奶后开食料采食量会增加,会导致瘤胃微生物植入的速度明显加快。Chen[17]研究发现,当犊牛饲粮转变为淀粉水平相对较高的高谷物饲粮时,犊牛瘤胃微生物菌群组成的多样性降低;另外,根据Khafipour等[8]、Tajima等[18]和张红涛[19]的研究,饲粮中性洗涤纤维(NDF)水平的升高使得瘤胃微生物区系的物种丰富度和多样性均增高,本文在将开食料改变为生长羔羊颗粒料时,淀粉水平由38.81%降低为28.93%,NDF水平由18%升高至22%,这种饲粮营养成分的转变也可能是56日龄断奶组微生物种类、数量较大的原因。
随着日龄的增长,羔羊固体料的采食量会随之增长,固体料采食的增长会引起瘤胃内微生物区系产生变化[20]。14日龄的DGGE图谱条带数目和颜色深度明显高于7日龄。这是由于在7日龄对羔羊进行了补饲,虽然日龄较小的羔羊对固体开食料采食量并不大,但是通过舔舐固体料可以让微生物植入瘤胃[21]。70日龄的DGGE图谱条带数目高于56日龄,鉴于56~70日龄羔羊瘤胃内总挥发性脂肪酸的含量显著升高(102.31 mmol/L vs. 96.72 mmol/L), 而瘤胃内pH也呈增高趋势[22],这说明在将开食料更换为生长羔羊料后,瘤胃微生物群落的活动使得饲粮在瘤胃中迅速发酵,而发酵底物的增多则有利于微生物增殖,同样的,相对较高的pH也有利于纤维降解菌的增殖,因此这种现象可能是由断奶后开食料采食量增加以及固体饲料的成分的转变共同引起的。
Bomba等[23]通过对比7周龄断奶与9周龄断奶犊牛瘤胃内微生物发育状况,发现通过早期补饲开食料7周龄犊牛瘤胃内微生物发育与9周龄犊牛差异不大。在本研究中也是在28日龄断奶的情况下采取了早期补饲的措施,因此我们的研究结果与Bomba等[23]一致。Yáñez-Ruiz等[24]研究表明,刚出生的反刍动物瘤胃内含有极少数的微生物甚至不含有微生物,随着与外界接触微生物逐渐植入到瘤胃中。本试验中随着日龄的增长,组内相似度逐渐升高,这与Rey等[25]的研究结果一致。随着日龄的增长,反刍动物瘤胃内的微生物区系逐渐趋于稳定状态,达到一种动态的平衡,说明随饲粮植入的微生物对瘤胃内微生物区系的影响已逐渐变小。从1~84日龄组内相似度逐渐升高的趋势下,波动最大的为1~14日龄和56~70日龄这2个时间段。本研究在7日龄对羔羊补饲开食料以及在56日龄进行了断奶,这2个处理改变了羔羊饲粮营养物质摄入组成,因此引起了瘤胃内微生物区系的变化。
Bomba等[23]研究发现,犊牛断奶后瘤胃内微生物多样性指数呈显著性升高。本试验中,28日龄断奶组在42日龄时已经断奶,而56日龄断奶组在42日龄时仍未断奶,造成了在42日龄时28日龄断奶组多样性指数显著高于56日龄断奶组。从结果中可以得出,7日龄补饲和56日龄断奶对瘤胃内的微生物区系影响较大。Jami等[12]研究发现,改变反刍动物的饲粮营养物质组成可以直接影响瘤胃内微生物区系的组成。本研究补饲与断奶的处理间接改变了动物饲粮营养结构组成。
4 结论① 在7日龄开始饲喂开食料条件下,28日龄断奶的羔羊瘤胃微生物多样性在42日龄时较56日龄断奶的羔羊丰富。
② 羔羊瘤胃内微生物多样性随着日龄的增长逐渐丰富,14日龄(7日龄开始补饲开食料)较1日龄显著增加,70日龄较断奶(56日龄)前显著增加。
| [1] |
冯仰廉. 反刍动物营养学[M]. 北京: 科学出版社, 2006.
|
| [2] |
淡瑞芳, 张海涛, 龙瑞军, 等. 瘤胃微生物生态研究方法评述[J]. 草业科学, 2007, 24(7): 77-82. |
| [3] |
DE FILIPPO C, CAVALIERI D, DI PAOLA M, et al. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa[J]. Proceedings of The National Academy of Sciences of the United States of America, 2010, 107(33): 14691-14696. DOI:10.1073/pnas.1005963107 |
| [4] |
陈昊, 刘婷, 吴建平, 等. 牛至精油对新生犊牛生长发育和血液免疫的影响[J]. 草业科学, 2017, 34(10): 2141-2148. |
| [5] |
罗惦, 柴林荣, 常生华, 等. 我国青藏高原地区牦牛草地放牧系统管理及优化[J]. 草业科学, 2017, 34(4): 881-891. |
| [6] |
宫曼丽, 任南琪, 邢德峰. DGGE/TGGE技术及其在微生物分子生态学中的应用[J]. 微生物学报, 2004, 44(6): 845-848. |
| [7] |
FIRKINS J L, YU Z. Ruminant nutrition symposium:how to use data on the rumen microbiome to improve our understanding of ruminant nutrition[J]. Journal of Animal Science, 2015, 93(6): 1450-1470. |
| [8] |
KHAFIPOUR E, LI S C, PLAIZIER J C, et al. Rumen microbiome composition determined using two nutritional models of subacute ruminal acidosis[J]. Applied and Environmental Microbiology, 2009, 75(22): 7115-7124. DOI:10.1128/AEM.00739-09 |
| [9] |
LEY R E, HAMADY M, LOZUPONE C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320(5883): 1647-1649. DOI:10.1126/science.1155725 |
| [10] |
张宁. 断奶和补饲时间对湖羊羔羊肌肉脂肪酸和瘤胃微生物多样性的影响[D]. 硕士学位论文. 兰州: 兰州大学, 2016.
|
| [11] |
周奕毅. 茶皂素抑制湖羊甲烷生成的微生物学机制研究[D]. 硕士学位论文. 杭州: 浙江大学, 2009.
|
| [12] |
JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [13] |
马志远. 早期断奶对湖羊羔羊生产性能及胃肠道发育的影响[D]. 硕士学位论文. 兰州: 兰州大学, 2015.
|
| [14] |
柴建民. 断母乳日龄对羔羊生长性能与胃肠道发育的影响[D]. 硕士学位论文. 北京: 中国农业科学院, 2015.
|
| [15] |
韩旭峰. 日龄、日粮精粗比对陕北白绒山羊瘤胃微生物区系影响的研究[D]. 博士学位论文. 杨凌: 西北农林科技大学, 2015.
|
| [16] |
MARTIN C, MICHALET-DOREAU B. Variations in mass and enzyme activity of rumen microorganisms:effect of barley and buffer supplements[J]. Journal of The Science of Food and Agriculture, 1995, 67(3): 407-413. DOI:10.1002/(ISSN)1097-0010 |
| [17] |
CHEN Y H. Changes in ruminal bacterial communities of beef cattle during high concentrated diet transition and experimental induced subacute acidosis[D]. Master's Thesis. Edmonton: University of Alberta, 2011.
|
| [18] |
TAJIMA K, AMINOV R I, NAGAMINE T, et al. Diet-dependent shifts in the bacterial population of the rumen revealed with real-time PCR[J]. Applied and Environmental Microbiology, 2001, 67(6): 2766-2774. DOI:10.1128/AEM.67.6.2766-2774.2001 |
| [19] |
张红涛. 不同玉米青贮水平对荷斯坦后备牛瘤胃液微生物组及其代谢组的影响[D]. 博士学位论文. 北京: 中国农业大学, 2017.
|
| [20] |
KHAN M A, BACH A, WEARY D M, et al. Invited review:transitioning from milk to solid feed in dairy heifers[J]. Journal of Dairy Science, 2016, 99(2): 885-902. DOI:10.3168/jds.2015-9975 |
| [21] |
EL-SHAZLY K, HUNGATE R E. Fermentation capacity as a measure of net growth of rumen microorganisms[J]. Applied Microbiology, 1965, 13: 62-93. |
| [22] |
LIU T, LI F D, WANG W M, et al. Effects of lamb early starter feeding on the expression of genes involved in volatile fatty acid transport and pH regulation in rumen tissue[J]. Animal Feed Science and Technology, 2016, 217: 27-35. DOI:10.1016/j.anifeedsci.2016.04.006 |
| [23] |
BOMBA A, KMET V, KONIAROVÁ I, et al. Production of volatile fatty acids in the rumen of calves during dietary-microbial stimulation and early weaning[J]. Veterinární Medicina, 1989, 34(3): 141-148. |
| [24] |
YÁÑEZ-RUIZ D R, ABECIA L, NEWBOLD C J. Manipulating rumen microbiome and fermentation through interventions during early life:a review[J]. Frontiers in Microbiology, 2015, 34: 1133. |
| [25] |
REY M, ENJALBERT F, COMBES S, et al. Establishment of ruminal bacterial community in dairy calves from birth to weaning is sequential[J]. Journal of Applied Microbiology, 2014, 116(2): 245-257. DOI:10.1111/jam.2014.116.issue-2 |




