2. 扬州大学农业科技发展研究院, 扬州 225009
2. Institutes of Agricultural Science and Technology Development, Yangzhou University, Yangzhou 225009, China
鹅具有消化利用含高水平粗纤维饲粮的生理特点,然而,家禽本身并不能分泌纤维素分解酶或半纤维素分解酶。鹅的空肠后段、回肠、盲肠内含有的大量微生物可分泌消化纤维素的酶,对纤维组分的降低起到了关键作用。Yang等[1]研究表明,鹅结扎盲肠后,酸性洗涤纤维、中性洗涤纤维的利用率有所下降。Garcia[2]研究表明,当给鹅饲喂低能量、高纤维的饲粮时,鹅的盲肠对养分的消化率起到了关键作用。通过添加不同比例的粗纤维饲料,如稻壳[3]或羊草[4],可改变鹅的盲肠微生物组成。目前,已有的报道主要集中于粗纤维饲料原料含量改变引起的粗纤维水平变化对鹅生长性能、肠道结构和生长发育等的影响[3-6]。本团队的前期研究发现,通过微晶纤维素提高饲粮粗纤维水平可以显著改善仔鹅28日龄体重和平均日增重(ADG)[7]。虽然盲肠微生物可能在其中发挥了调节生长性能的重要作用,但上述研究并未阐述饲粮粗纤维水平对盲肠微生物的影响。推测通过添加适量的微晶纤维素改变饲粮粗纤维水平可以改善仔鹅生长早期的盲肠微生物区系。因此,本试验以微晶纤维素为纤维来源,研究饲粮粗纤维水平对1~28日龄仔鹅盲肠微生物区系的影响,为研究鹅对饲粮粗纤维的消化机制提供依据。
1 材料与方法 1.1 试验设计与饲养管理选择健康、体重接近的1日龄扬州鹅公鹅(购自扬州市高邮扬州鹅种鹅场)468只,随机分为3个组,每组6个重复,每个重复26只。对照组(Ⅰ组)饲喂基础饲粮,试验组在基础饲粮基础上分别添加1.86%(Ⅱ组)和3.72%(Ⅲ组)纯纤维素(微晶纤维素,河南旗诺食品配料有限公司,纯度99.8%),使Ⅰ、Ⅱ、Ⅲ组饲粮粗纤维水平分别达2.5%、4.3%和6.1%。基础饲粮的配制参照本实验室多年来的相关科研成果,饲粮组成及营养水平参见Li等[8]。全程舍内网上平养,自由采食和饮水,并保持圈内卫生、通风良好。严格执行种鹅场提供的常规免疫程序。试验期28 d。
1.2 生长性能测定饲养过程中观察和记录鹅群死淘状况,记录死亡鹅脚号、日期、耗料量和剩余总料及体重。每周末以重复为单位对试验鹅进行称重、记录采食量,以7 d为间隔计算平均日采食量(ADFI)、平均日增重和料重比(F/G)。
1.3 样品采集在28日龄时,从每个重复中随机选取接近该重复平均体重的2只试验鹅解剖,无菌操作分离盲肠,用灭菌剪刀轻轻剖开肠段,取出内容物分2份置于无菌离心管中,-70 ℃冰箱中保存,一份用于PCR扩增后对16S rRNA的V3~V4区进行Illumina MiSeq测序;另一份在PCR扩增后进行PCR扩增片段变性梯度凝胶电泳(PCR-DGGE)和实时荧光定量PCR检测。
1.4 总DNA提取、定量、纯度检测、PCR扩增及细菌16S rRNA测序分析取28日龄仔鹅盲肠内容物1份进行微生物总DNA的提取(粪便基因组DNA提取试剂盒,天根生化科技有限公司)、定量和纯度检测(Qubit 2.0 DNA检测试剂盒,NANODROP 1000 Spectrophotometer仪,美国Thermo公司)、PCR扩增(PTC-200PCR基因扩增仪,美国MJ公司)、PCR产物回收(PCR产物凝胶回收试剂盒,天根生化科技有限公司)及细菌16S rRNA测序分析(上海生工生物工程有限公司,Illumina MiSeq 2000平台)。详细操作参照试剂盒说明书,详细步骤参见Li等[8]的研究方法。同一组的12个样品PCR产物各取10 ng按1 : 1混合,上机测序浓度为20 pmol/L。
1.5 基因组总DNA 16S rRNA的V3区PCR-DGGE由于16S rRNA测序中样品重复数有限,本研究通过PCR-DGGE检测方法从另一个角度对盲肠中主要微生物的丰度进行补充研究。按上述1.4的方法,取第2份仔鹅盲肠内容物重新进行微生物总DNA的提取、定量、纯度检测、PCR扩增、PCR产物定量和混合。参照Muyzer等[9]的方法,制作8%的聚丙烯酰胺凝胶,对16S rRNA的V3区的PCR扩增产物进行电泳分离。变性剂浓度为45%~60%,电泳缓冲液为1×TAE,60 ℃恒温80 V电泳15 h,电泳结束后使用硝酸银(AgNO3)染色,显色定影后用Vilber凝胶成像扫描系统照像,详细参数和步骤参见刘蓓一[3]。
1.6 实时荧光定量PCR由于16S rRNA测序中样品重复数有限,本研究通过实时荧光定量PCR法从另一个角度对盲肠中的主要微生物进行补充研究。引物序列和荧光定量PCR试剂盒(SYBR Green Ⅰ)购自美国Invitrogen公司。按照试剂盒说明书进行操作,利用Roche荧光定量PCR仪(罗氏公司)实时检测。详细引物序列参见刘蓓一[3],检测步骤参见谢燕娟[10]。每个样品以自身总菌的DNA为内参,实时荧光定量PCR的CT值经过2-ΔΔCT法[11]计算获得最终结果,反映每种细菌相对于自身肠道总菌数量的相对丰度。为了便于比较,对照组相对值表示为1.00,试验组相对值为对照组的倍数。
1.7 数据统计与分析生长性能和实时荧光定量PCR检测数据采用Excel 2010统计处理,SPSS 17.0软件进行单因素方差分析(one-way ANOVA),LSD法进行差异显著性比较。本试验对97%相似水平的操作分类单元(OTU)序列进行分类学分析,并在门、属2个水平上统计每个样品的群落组成,并用Excel 2010和Power Point 2010绘制柱状图。分别计算ACE指数、Chao1指数、Shannon指数、Simpson指数,比较各组之间微生物区系的丰富度及多样性。
2 结果 2.1 饲粮粗纤维水平对1~28日龄仔鹅生长性能的影响由表 1可知,1~7日龄和8~14日龄,各组的平均日采食量、平均日增重、料重比均无显著差异(P>0.05);15~21日龄,Ⅱ和Ⅲ组的平均日增重高于Ⅰ组,料重比低于Ⅰ组,但差异不显著(P>0.05);22~28日龄,Ⅱ和Ⅲ组的平均日增重显著高于Ⅰ组(P < 0.05),料重比显著低于Ⅰ组(P < 0.05)。
|
|
表 1 饲粮粗纤维水平对1~28日龄仔鹅生长性能的影响 Table 1 Effects of dietary crude fiber level on growth performance of geese during 1 to 28 days of age |
由表 2可知,通过Illumina MiSeq高通量测序从3组盲肠内容物样品中共得到71 178条序列总数以及4 640个OTU总数,其中,Ⅰ组检测出21 055条序列总数以及1 318个OTU总数。Ⅱ和Ⅲ组的序列总数分别比Ⅰ组高6%和32%,OTU总数分别比Ⅰ组高17%和35%。Ⅱ和Ⅲ组的ACE指数分别比Ⅰ组高97%和98%,Chao1指数分别比Ⅰ组高20%和73%,Shannon指数分别比Ⅰ组高31%和72%,Simpson指数分别比Ⅰ组低42%和16%。以上alpha多样性指数(ACE指数、Chao1指数、Shannon指数、Simpson指数)共同显示,与Ⅱ、Ⅲ组相比,Ⅰ组盲肠微生物物种丰度和多样性都更低。
|
|
表 2 饲粮粗纤维水平对28日龄仔鹅盲肠微生物alpha多样性指数的影响 Table 2 Effects of dietary crude fiber level on alpha diversity index in cecum microflora of geese at 28 days of age |
由图 1可知,3组盲肠内容物样品在门水平上的微生物种群大部分均为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria),并且饲粮粗纤维水平导致的盲肠微生物差异主要存在于这3个门中,其中对变形菌门的影响最大。与Ⅱ和Ⅲ组相比,Ⅰ组的变形菌门相对丰度增加,而拟杆菌门和厚壁菌门相对丰度降低。其中,与Ⅱ组相比,Ⅰ组变形菌门相对丰度增加了86.1%,拟杆菌门、厚壁菌门相对丰度分别降低了9.9%和3.5%;与Ⅲ组相比,Ⅰ组变形菌门相对丰度增加了116.6%,拟杆菌门和厚壁菌门相对丰度分别降低了5.8%和10.1%。
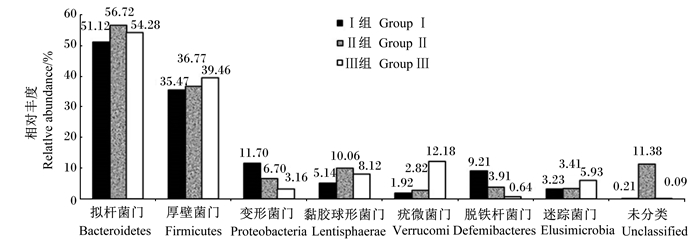
|
图 1 饲粮粗纤维水平对28日龄仔鹅盲肠微生物种在门水平的相对丰度的影响 Fig. 1 Effects of dietary crude fiber level on relative abundances of cecal microflora at phylum level of geese at 28 days of age |
由图 2可知,与Ⅱ和Ⅲ组相比,Ⅰ组拟杆菌属(Bacteroides)、巨单胞菌属(Megamonas)、普雷沃菌属(Paraprevotella)的相对丰度降低。其中,Ⅰ组与Ⅱ组相比,以上3种菌属的相对丰度分别降低了17.7%、62.0%、48.9%;Ⅰ组与Ⅲ组相比,分别降低了11.4%、81.9%、36.7%。
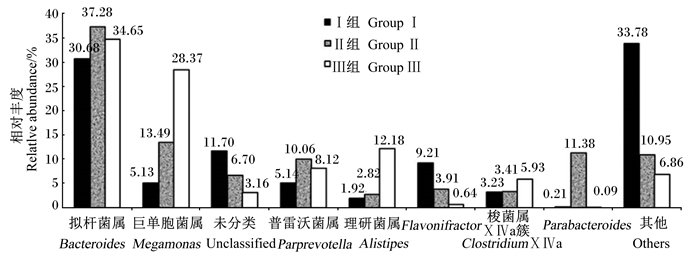
|
图 2 饲粮粗纤维水平对28日龄仔鹅盲肠微生物在属水平的相对丰度的影响 Fig. 2 Effects of dietary crude fiber level on relative abundances of cecal microflora at genus level of geese at 28 days of age |
由图 3可知,Ⅰ组与Ⅲ组距离最远,盲肠微生物相似性最低。Ⅱ组与Ⅲ距离最小,盲肠微生物的相似性最高。Ⅰ组与Ⅱ、Ⅲ组之间盲肠微生物的相似性均低于Ⅱ组与Ⅲ组之间盲肠微生物的相似性。
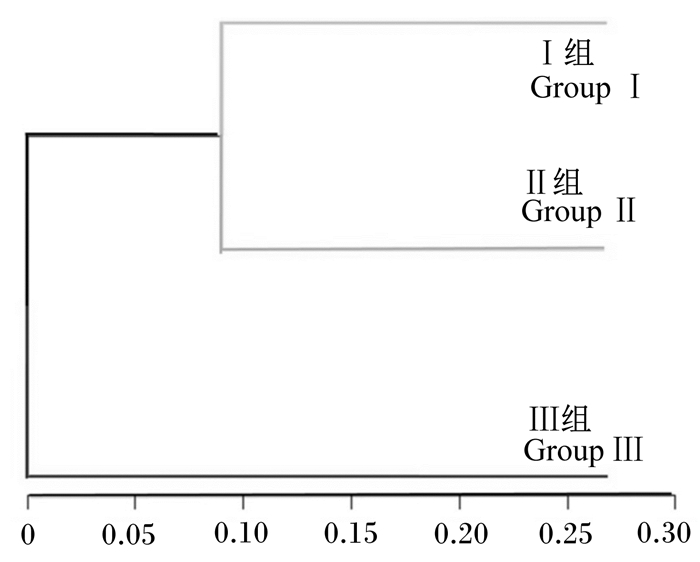
|
图 3 饲粮粗纤维水平对28日龄仔鹅盲肠微生物相似性聚类分析的影响 Fig. 3 Effects of dietary crude fiber level on similarity cluster analysis of cecal microflora of geese at 28 days of age |
3组试验饲粮的仔鹅肠道菌群16S rRNA的V3区PCR-DGGE指纹图谱见图 4-A,条带数分析结果见图 4-B。指纹图谱显示饲粮粗纤维水平影响了鹅盲肠微生物结构与组成。28日龄时,Ⅱ和Ⅲ组的仔鹅盲肠中的微生物条带数分别比Ⅰ组多18.2%和31.8%。
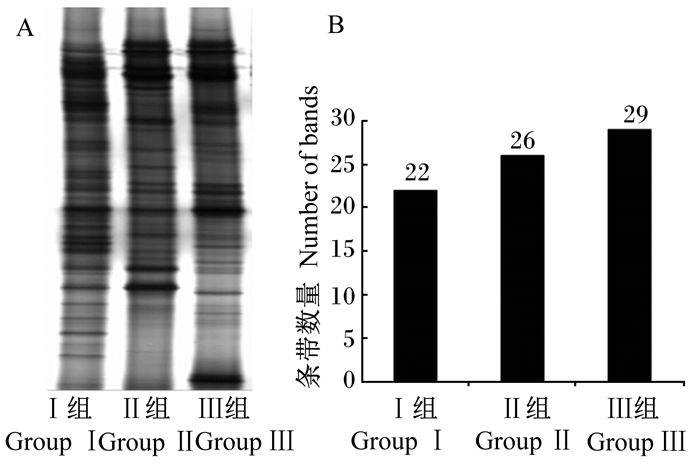
|
图 4 饲粮粗纤维水平对28日龄仔鹅盲肠微生物PCR-DGGE指纹图谱的影响 Fig. 4 Effects of dietary crude fiber level on PCR-DGGE fingerprinting of cecal microflora of geese at 28 days of age |
由表 3可知,Ⅰ组变形菌门的代表菌大肠杆菌的相对丰度比Ⅱ和Ⅲ组显著增加(P < 0.05);而Ⅰ组中厚壁菌门的代表菌梭菌Ⅳ、梭菌ⅩⅣ和乳酸菌的相对丰度比Ⅱ和Ⅲ组显著降低(P < 0.05),此外,韦荣球菌的相对丰度也显著低于Ⅱ组(P < 0.05)。Ⅰ组拟杆菌门的代表菌拟杆菌和放线菌门的代表菌双歧杆菌的相对丰度比Ⅱ和Ⅲ组显著降低(P < 0.05)。Ⅲ组除拟杆菌的相对丰度显著高于Ⅱ组(P < 0.05)外,其他细菌的相对丰度均与Ⅱ组差异不显著(P>0.05)。
|
|
表 3 饲粮粗纤维水平对28日龄仔鹅盲肠微生物相对丰度的影响 Table 3 Effects of dietary crude fiber level on relative abundance of cecal microflora of geese at 28 days of age |
本试验显示,饲粮粗纤维水平在2.5%~6.1%时,雏鹅在1~7日龄、8~14日龄、15~21日龄3个阶段的生长性能不受饲粮粗纤维水平的显著影响。但是到了第22~28日龄,仔鹅对粗纤维的需求量增加,过低的饲粮粗纤维水平(2.5%)将阻碍饲料转化效率并降低生长速度。此外,在1~21日龄,扬州鹅仔鹅采食粗纤维水平在4.3%~6.1%变化饲粮对生长性能无有害影响。一般情况下,随着饲粮粗纤维水平的上升,饲粮中的有效能等主要营养物质的含量会下降,而植酸等抗营养因子水平增加,可能导致家禽,尤其是雏禽的生长性能下降。章双杰等[12]研究了1~4周太湖鹅雏鹅对饲粮粗纤维水平(3.5%、5.0%和6.5%)的反应,认为低饲粮粗纤维水平(3.5%)导致饲料转化效率高于高饲粮粗纤维水平(6.5%)。杨桂芹等[13]研究了饲粮粗纤维水平对1~35日龄鹅生长性能的影响,也认为低粗纤维水平饲粮组的平均日增重、料重比明显优于中、高粗纤维水平饲粮组,且以3.5%的饲粮粗纤维水平为宜。张玲等[14]研究了3种饲粮粗纤维水平(3.5%、5.5%和7.5%)对1~4周龄肉鹅生长性能的影响,结果显示,3.5%粗纤维水平饲粮组在育雏结束时的平均日增重极显著高于5.5%和7.5%粗纤维水平饲粮组。以上2个研究认为3.5%的饲粮粗纤维水平适合生长前期的仔鹅,该结论与本研究结果接近。类似地,张慧[15]认为1~4周龄雏鹅饲粮中粗纤维水平以3.24%~4.02%为宜。这些饲粮中粗纤维适宜水平(或耐受量)的上限低于本研究的原因可能有3个:第一,可能与不同鹅的品种有关;第二,本试验采用的低粗纤维水平组是极低的粗纤维水平(2.5%),说明过低的粗纤维水平影响鹅的正常生长,而4.3%~6.1%的粗纤维水平适合扬州仔鹅在1~28日龄的生长;第三,本研究中导致粗纤维变化的因素是单一的微晶纤维素而不是常规的饲料原料,不会像普通粗纤维饲料、能量或蛋白质副产物原料那样随着粗纤维水平的增加而植酸或霉菌毒素等抗营养因子的含量也增加,故而本试验中仔鹅可以适应更高水平的粗纤维。
Gong等[16]研究结果表明,5个以上的个体混合样品才能代表整体的微生物组成。本研究用了6个重复共12只个体的混合样品,采用了基因组16S rRNA的Illumina MiSeq高通量测序分析方法探讨了饲粮粗纤维水平对生长前期仔鹅盲肠微生物区系的影响。Gong等[17]指出,通过基因组16S rRNA的分子分析方法获得的微生物种群多样性大于传统的培养方法。本研究中,PCR-DGGE法得到的条带只有30多条,而Illumina MiSeq高通量分析得到的序列总数多达27 891条,OTU总数达1 778个,与以上结论相似。虽然PCR-DGGE所表现的微生物DNA条带很有限,但是验证了高通量测序显示的微生物多样性趋势,即4.3%和6.1%粗纤维水平组高于2.5%粗纤维水平组。
通过对菌种、菌属等数码信息进行归类操作,将序列按照彼此的相似性分归为许多小组,即OTU,该数据可以反映肠道菌群的物种数目信息。其中用Chao1算法估计群落中含OTU数目的指数,即Chao1指数,在生态学中常用来估计物种总数,反映肠道菌群的物种丰度[18]。ACE指数同样是用来估计群落中OTU数目的指数,也是生态学中估计物种总数的常用指数之一。Shannon指数和Simpson指数都是反映样品中微生物多样性的指数,Shannon指数越大,说明样品多样性越高,微生物分配越均匀[19];而Simpson指数越大,说明群落多样性越低[20]。本试验结果中,饲粮粗纤维水平最低的对照组的ACE指数、Chao1指数、Shannon指数低于其他2组,而Simpson指数高于其他2组。这些结果与PCR-DGGE结果一致,表明2.5%的低粗纤维水平饲粮降低了仔鹅盲肠微生物丰度以及多样性。与生长性能相一致,饲粮粗纤维水平最低的对照组22~28日龄的平均日增重低于其他2组,并且料重比高于其他2组,说明低粗纤维水平饲粮可能通过降低仔鹅盲肠微生物丰度和多样性导致22~28日龄仔鹅生长性能下降。相反,适当地添加纤维素可能通过增加盲肠微生物丰度和多样性促进了22~28日龄仔鹅生长性能的提高。
通过盲肠微生物门水平的组成柱状图分析发现,拟杆菌门、厚壁菌门和变形菌门是盲肠微生物的主要组成部分,这与鹅[9]和鸡[21]盲肠中的微生物组成的报道相一致。低粗纤维水平饲粮导致变形菌门丰度增加,而拟杆菌门和厚壁菌门丰度降低,与仔鹅饲养后期(29~70日龄)受饲粮粗纤维水平影响的盲肠微生物变化结果[9]一致。Daly等[22]认为,厚壁菌门中包含有较多的纤维分解菌。Matsui等[23]指出,当盲肠中含有较多的拟杆菌门时,其纤维分解菌含量也较多,发酵作用也较强。本研究中,提高饲粮粗纤维水平增加了28日龄仔鹅盲肠微生物中拟杆菌门、厚壁菌门的相对丰度,说明提高饲粮粗纤维水平促进了纤维分解菌的生长。与该推测一致,本团队的前期研究还显示,高纤维素组的粗纤维、中性洗涤纤维和酸性洗涤纤维的消化率与低纤维组无显著差异,但是由于饲粮中的粗纤维含量更高,粗纤维被消化的总量大约是低纤维素组的2倍[7]。
变形菌门包括很多病原菌,如沙门氏菌、大肠杆菌、幽门螺杆菌、霍乱弧菌等。本研究中,最低的粗纤维水平(2.5%)组与较高的粗纤维水平(4.5%、6.3%)组相比,变形菌门的相对丰度增加幅度高达86.1%~116.6%,从而潜在地增加了有害微生物对仔鹅肠道健康的危害。本研究的实时荧光定量PCR也反映出Ⅰ组大肠杆菌的相对丰度比Ⅱ和Ⅲ组显著提高,与16S rRNA高通量测序结果一致。适当地提高饲粮粗纤维水平提高了仔鹅22~28日龄的平均日增重,并改善了饲料转化效率,与微生物结果一致。本团队的前期研究显示,与2.5%粗纤维水平相比,4.5%和6.3%粗纤维水平导致仔鹅对能量和蛋白质的消化率以及粗纤维的消化总量更高[7]。因此,适宜的饲粮粗纤维水平可能通过减少了变形菌门中的有害微生物,并增加了拟杆菌们和厚壁菌门中的有益微生物提高了仔鹅早期(22~28日龄)的肠道健康和营养物质消化率,从而改善了生长性能。
盲肠微生物种群在属水平上的主要菌群变化也和门水平上的变化一致。其中,过低的粗纤维水平导致拟杆菌门中的代表菌拟杆菌属、普雷沃菌属和理研菌属的相对丰度降低,并且厚壁菌门中的Flavonifractor和梭菌ⅩⅣa的相对丰度也降低。拟杆菌门的代表菌属拟杆菌属在帮助分解多糖、提高多糖利用率[24]、加快黏膜[25]及免疫系统的发育、提高机体的免疫功能[26]、维持肠道微生态平衡[27]方面起到了重要的作用。拟杆菌门的另一个代表菌属普雷沃菌属(Paraprevotella)能够将淀粉、多肽和粗纤维成分如果胶、木聚糖等发酵转化为短链脂肪酸[28]。过低的饲粮粗纤维水平导致巨单胞菌属的相对丰度下降,而该菌可以发酵各种碳水化合物,产生乙酸、丙酸和乳酸。短链脂肪酸对肠道健康有重要影响,可作为肠道上皮细胞的首选能源,并能保护肠道上皮,抑制肠炎和肠道癌症的发生[29]。本研究中,与4.5%、6.3%粗纤维水平饲粮相比,低粗纤维水平(2.5%)饲粮降低了28日龄仔鹅盲肠中拟杆菌属、普雷沃菌属的相对丰度,还导致未分类菌相对丰度的增加,该结果与Li等[8]关于纤维素影响鹅70日龄盲肠微生物丰度的研究结果一致。过低的饲粮粗纤维水平(2.5%)导致22~28日龄(本试验)和29~70日龄(Li等[7])鹅的生长性能下降。以上研究说明,过低的粗纤维水平可能由于提供的粗纤维底物有限而导致拟杆菌属和普雷沃菌属的相对丰度下降,从而部分导致仔鹅的饲料转化效率和生长速度降低,这与Li等[8]的研究一致。过低的饲粮粗纤维水平还导致未分类菌相对丰度的增多,但具体是什么菌发挥主要影响还有待于进一步研究。
实时荧光定量PCR结果显示,饲粮粗纤维水平过低导致厚壁菌门的代表菌梭菌Ⅳ、梭菌ⅩⅣ,拟杆菌门的代表菌拟杆菌的相对丰度下降,与刘蓓一[3]的研究结果一致;梭菌Ⅳ群中的柔嫩梭菌以及梭菌ⅩⅣa群中的真杆菌/罗氏菌是产丁酸的主要细菌,而丁酸可促进调节性T淋巴细胞(Treg)的发育,抑制肠炎和过敏性腹泻[30]。在刘蓓一[3]的研究中,饲粮粗纤维水平的变化来源于不同比例的稻壳替代基础饲粮,其结果显示随着饲粮粗纤维水平的增加(40%的稻壳),仔鹅盲肠拟杆菌属、梭菌Ⅳ和梭菌ⅩⅣ等相对丰度有所增加。本研究中,梭菌ⅩⅣ的相对丰度变化趋势在16S rRNA测序和实时荧光定量PCR 2种方法的检测下具有一致的结果,且梭菌ⅩⅣ相对丰度高的试验组其鹅的生长性能获得提高。本研究结果表明,过低的饲粮粗纤维水平导致厚壁菌门的代表菌梭菌和乳酸菌、放线菌门的代表菌双歧杆菌的相对丰度降低,与张名爱等[4]的研究结果一致,其在五龙鹅饲粮中以不同比例的羊草构成了饲粮粗纤维水平的变化。高粗纤维水平饲粮中的纤维素水解最终分解产物为挥发性脂肪酸,呈酸性,并且高粗纤维水平饲粮可导致乳酸菌和双歧杆菌相对丰度增加,将肠道中的可发酵性糖转变成醋酸和乳酸,从而可降低盲肠pH,抑制杂菌的滋生[4]。大肠杆菌属中存在很多致病性血清型,可通过黏附素、外毒素和内毒素破坏肠道健康。本试验中,增加饲粮粗纤维水平导致大肠杆菌相对丰度减少,与刘蓓一[3]报道的适当提高粗纤维水平(40%)降低大肠杆菌相对丰度的研究结果一致,然而他们的研究还表明当粗纤维水平过高时(60%稻壳替代基础饲粮)反而会导致大肠杆菌相对丰度的增加。低粗纤维水平饲粮降低了22~28日龄仔鹅的生长速度和饲料转化效率,其原因可能与低粗纤维水平饲粮减少了纤维素分解菌及一些产生短链脂肪酸的有益微生物的相对丰度、并增加了大肠杆菌等潜在致病菌的相对丰度有关。
另外,在微生物聚类分析图中,把不同组之间的相似性转变为距离,以便于直观地分析[31]。2组之间距离越远,相似性越低,反之,相似性越高。本试验中,2.5%粗纤维水平饲粮组与4.3%、6.5%粗纤维水平饲粮组之间的盲肠微生物相似性较低,4.3%与6.5%粗纤维水平饲粮组之间的盲肠微生物相似性较高。与本研究的生长性能结果一致,即4.3%和6.5%粗纤维水平饲粮组仔鹅22~28日龄平均日增重显著高于2.5%粗纤维水平饲粮组,而4.3%和6.5%粗纤维水平饲粮组之间无显著差异。这进一步表明饲粮粗纤维水平不仅可以影响仔鹅盲肠微生物区系,也可能通过调节盲肠微生物区系反向影响仔鹅生长性能。
4 结论① 过低的饲粮粗纤维水平(2.5%)降低了盲肠微生物的多样性,降低了拟杆菌门及本门中拟杆菌属、普雷沃菌属、理研菌属的相对丰度,降低了厚壁菌门及本门中Flavonifractor、梭菌Ⅳ、梭菌ⅩⅣ、韦荣球菌、乳酸菌的相对丰度,降低了巨单胞菌属的相对丰度和放线菌门中的双歧杆菌的相对丰度,增加了变形菌门及本门中的大肠杆菌的相对丰度。
② 通过添加纤维素提高饲粮粗纤维水平到4.3%和6.1%,可增加28日龄仔鹅盲肠微生物多样性和潜在有益菌的相对丰度,降低潜在有害菌的相对丰度,改善盲肠微生物平衡。
| [1] |
YANG H M, WANG Z Y, WANG J, et al. Effects of caecectomy on digestibility of crude protein, calcium, phosphorus, neutral detergent fibre and acid detergent fibre in geese[J]. Archiv fur Geflügelkunde, 2009, 73(3): 189-192. |
| [2] |
GARCIA D M.The role of the giant Canada goose (Branta Canadensis maxima) cecum in nutrition[D]. Ph.D.Thesis.Columbia: University of Missouri, 2006: 1-132.
|
| [3] |
刘蓓一.扬州鹅肠道微生物多样性及其受饲粮纤维水平的调控研究[D].博士学位论文.扬州: 扬州大学, 2012: 71-113. http://cdmd.cnki.com.cn/Article/CDMD-11117-1013181522.htm
|
| [4] |
张名爱, 王宝维, 龙芳羽, 等. 日粮纤维水平对鹅肠道正常菌群的影响[J]. 福建农林大学学报(自然科学版), 2007, 36(2): 159-162. |
| [5] |
ZHANG H Q, YE H, WANG W C, et al. Effects of dietary fiber on growth performance, enzyme activities, microflora and digesta physicochemical characteristics of Lionhead geese[J]. Journal of Food Agriculture and Environment, 2013, 11(3): 1653-1658. |
| [6] |
HE L W, MENG Q X, LI D Y, et al. Effect of different fibre sources on performance, carcass characteristics and gastrointestinal tract development of growing Greylag geese[J]. British Poultry Science, 2015, 56(1): 88-93. DOI:10.1080/00071668.2014.981503 |
| [7] |
LI Y P, WANG Z Y, YANG H M, et al. Effects of dietary fiber on growth performance, slaughter performance, serum biochemical parameters, and nutrient utilization in geese[J]. Poultry Science, 2017, 96(5): 1250-1256. |
| [8] |
LI Y P, YANG H M, XU L, et al. Effects of dietary fiber levels on cecal microbiota composition in geese[J]. Asian-Australasian Journal of Animal Sciences, 2018, 31(8): 1285-1290. DOI:10.5713/ajas.17.0915 |
| [9] |
MUYZER G, DE WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700. |
| [10] |
谢燕娟.低粗纤维饲粮对仔鹅生长发育及盲肠微生物的影响[D].硕士学位论文.扬州: 扬州大学, 2016: 43-56. http://cdmd.cnki.com.cn/Article/CDMD-11117-1016284399.htm
|
| [11] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [12] |
章双杰, 宋迟, 汤青萍, 等. 不同代谢能、粗蛋白和粗纤维水平对太湖鹅生长性能和屠宰性能的影响[J]. 中国家禽, 2015, 37(13): 27-32. |
| [13] |
杨桂芹, 高妍. 0~35日龄肉用仔鹅日粮适宜粗纤维和粗蛋白水平的研究[J]. 中国家禽, 2006, 28(2): 11-13, 16. DOI:10.3969/j.issn.1004-6364.2006.02.004 |
| [14] |
张玲, 周根来, 段修军, 等. 日粮代谢能和粗纤维水平对0~4周龄肉鹅生产性能及血清生化指标的影响[J]. 中国兽医学报, 2015, 35(7): 1187-1192, 1198. |
| [15] |
张慧. 不同粗纤维含量饲料对豁鹅雏鹅增重的影响[J]. 辽宁畜牧兽医, 1995(2): 4-5. |
| [16] |
GONG J H, SI W D, FORSTER R J, et al. 16S rRNA gene-based analysis of mucosa-associated bacterial community and phylogeny in the chicken gastrointestinal tracts:from crops to ceca[J]. FEMS Microbiology Ecology, 2007, 59(1): 147-157. DOI:10.1111/j.1574-6941.2006.00193.x |
| [17] |
GONG J H, FORSTER R J, YU H, et al. Molecular analysis of bacterial populations in the ileum of broiler chickens and comparison with bacteria in the cecum[J]. FEMS Microbiology Ecology, 2002, 41(3): 171-179. DOI:10.1111/j.1574-6941.2002.tb00978.x |
| [18] |
CHAO A. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics, 1984, 11(4): 265-270. |
| [19] |
SHANNON C E. The mathematical theory of communication[J]. M.D. Computing:Computers in Medical Practice, 1997, 14(4): 306-317. |
| [20] |
WANG Y, SHENG H F, HE Y, et al. Comparison of the levels of bacterial diversity in freshwater, intertidal wetland, and marine sediments by using millions of illumina tags[J]. Applied and Environmental Microbiology, 2012, 78(23): 8264-8271. DOI:10.1128/AEM.01821-12 |
| [21] |
张李荣, 杨海明, 龚道清, 等. 应用Illumina MiSeq高通量测序技术分析饲粮添加乳双歧杆菌对蛋雏鸡盲肠微生物的影响[J]. 浙江农业学报, 2018, 30(10): 1630-1639. DOI:10.3969/j.issn.1004-1524.2018.10.03 |
| [22] |
DALY K, STEWART C S, FLINT H J, et al. Bacterial diversity within the equine large intestine as revealed by molecular analysis of cloned 16S rRNA genes[J]. FEMS Microbiology Ecology, 2001, 38(2/3): 141-151. |
| [23] |
MATSUI H, KATO Y, CHIKARAISHI T, et al. Microbial diversity in ostrich ceca as revealed by 16S ribosomal RNA gene clone library and detection of novel Fibrobacter species[J]. Anaerobe, 2010, 16(2): 83-93. DOI:10.1016/j.anaerobe.2009.07.005 |
| [24] |
BÄCKHED F, DING H, WANG T, et al. The gut microbiota as an environmental factor that regulates fat storage[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(44): 15718-15723. DOI:10.1073/pnas.0407076101 |
| [25] |
STAPPENBECK T S, HOOPER L V, GORDON J I. Developmental regulation of intestinal angiogenesis by indigenous microbes via Paneth cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(24): 15451-15455. DOI:10.1073/pnas.202604299 |
| [26] |
HOOPER L V. Bacterial contributions to mammalian gut development[J]. Trends in Microbiology, 2004, 12(3): 129-134. DOI:10.1016/j.tim.2004.01.001 |
| [27] |
SEARS C L. A dynamic partnership:celebrating our gut flora[J]. Anaerobe, 2005, 11(5): 247-251. DOI:10.1016/j.anaerobe.2005.05.001 |
| [28] |
SCHEPPACH W, LUEHRS H, MENZEL T. Beneficial health effects of low-digestible carbohydrate consumption[J]. British Journal of Nutrition, 2001, 85(Suppl.1): S23-S30. |
| [29] |
VON ROSENVINGE E C, SONG Y, WHITE J R, et al. Immune status, antibiotic medication and pH are associated with changes in the stomach fluid microbiota[J]. The ISME Journal, 2013, 7(7): 1354-1366. DOI:10.1038/ismej.2013.33 |
| [30] |
KAMADA N, NÚÑEZ G. Regulation of the immune system by the resident intestinal bacteria[J]. Gastroenterology, 2014, 146(6): 1477-1488. DOI:10.1053/j.gastro.2014.01.060 |
| [31] |
GOWER J C, ROSS G J S. Minimum spanning trees and single linkage cluster analysis[J]. Journal of the Royal Statistical Society.Series C:Applied Statistics, 1969, 18(1): 54-64. |




