奶水牛作为南方地区重要的地域畜牧资源,因其乳中含有丰富的营养和功能性成分越来越引起人们的关注和重视[1]。近年来,有一些关于奶水牛瘤胃微生物的研究,但大多数集中在单一年龄阶段。张慧敏等[2]对2.5岁的海子水牛瘤胃微生物进行高通量测序结果显示,优势菌门是拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和纤维杆菌门(Fibrobacteres),具有木质纤维素降解功能。Pandya等[3]对3岁左右的成年印度奶水牛瘤胃微生物进行16S rDNA测序分析,发现厚壁菌门和噬细胞菌-曲绕杆菌-拟杆菌菌门是优势菌群。有关不同年龄瘤胃微生物的研究,多集中在荷斯坦奶牛、牦牛和羊上,如逄宾宾等[4]在关于健康中国荷斯坦奶牛不同发育阶段瘤胃的菌群多样性研究中发现,中国荷斯坦奶牛瘤胃菌群结构组成存在显著差异,其中3、6月龄奶牛瘤胃菌群相对丰度低、结构简单,18、30月龄奶牛瘤胃菌群相对丰度高、结构复杂;Jami等[5]研究从出生到成年奶牛瘤胃菌群的组成变化,发现随年龄增长菌群多样性也增加,犊牛与成年奶牛瘤胃菌群组成差异显著;Guo等[6]研究不同年龄山羊瘤胃菌群组成,刘凯珍等[7]研究不同年龄牦牛瘤胃菌群差异,结果都显示不同年龄的瘤胃菌群组成存在一定差异并且有物种特异性。迄今为止,有关不同年龄奶水牛瘤胃菌群的变化鲜有报道。
本试验的目的是通过基于16S rRNA测序进行多样性分析,研究不同年龄奶水牛瘤胃菌群结构的变化和差异,探究奶水牛瘤胃菌群随宿主年龄增长变化的规律,为今后奶水牛瘤胃菌群调控提供理论指导依据。
1 材料与方法 1.1 试验设计及饲粮在广西水牛所水牛研究基地选择处于相近生活环境下的25头不同年龄的奶水牛,饲粮精粗比为40 : 60,试验饲粮组成及营养水平见表 1。用胃管式瘤胃液采样管采集所有奶水牛的瘤胃液,根据年龄分为4个组,分别为Y组(12月龄,n=9,胎次=0)、M组(3~5岁,n=7,胎次=1)、E组(6~8岁,n=4,胎次=2)和O组(9岁及以上,n=5,胎次≥3),采取的瘤胃液样品置于冰盒中迅速带回实验室,-20 ℃保存备用。除了Y组有3头杂交牛和1头尼里水牛外,其余所有奶水牛品种均为摩拉。
|
|
表 1 试验饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the experimental diet (DM basis) |
采用Sharma等[8]的十六烷基三甲基溴化铵(CTAB)法提取瘤胃液微生物总DNA,按照德国QIAGEN公司的DNA试剂盒进行DNA纯化;将质检合格的DNA样品作为模板,采用细菌16S rRNA基因V3~V4区的通用引物338F-806R来对其扩增DNA,长度约为468 bp。引物序列338F:5′-ACTCCTACGGGAGGCAGCAG-3′;引物序列806R:5′-GGACTACHVGGGTWTCTAAT-3′。将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris-HCl洗脱。
1.3 IIumina Miseq测序参照电泳初步定量结果,将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合,然后进行Miseq文库的构建,并使用IIumina Miseq 250平台(IIumina,美国)进行16S rRNA基因测序。
1.4 数据统计分析利用Usearch(Version 7.0)软件平台进行聚类操作统计各样本的菌群组成,并用R语言工具统计和作图(物种分类数据库为silva128/16s_bacteria);利用R语言工具制作Rank-Abundance曲线图来解释物种丰度和物种均匀度;运用Mothur(Version v.1.30.1)软件进行指数分析,反映菌群的相对丰度和多样性;Qiime软件和R语言工具进行beta多样性分析和作图,并运用单因素方差分析(one-way ANOVA)和LEfSe差异分析等统计方法对不同组间物种进行显著性分析,P < 0.05为差异显著,0.05 < P < 0.10为有显著差异的趋势;利用Network网络分析软件进行物种之间相关性分析。
2 结果与分析 2.1 样本DNA和测序数据评估琼脂糖凝胶电泳检测DNA质量结果显示,提取的DNA有部分低分子质量片段聚集,但是均可在高分子质量处观察到一条清晰可见的光带,提取的DNA浓度均大于30 ng/μL,OD260/OD280值都在2.00±0.15范围内;提取的DNA纯度良好,PCR产物目的条带大小正确,浓度合适,可进行后续试验。通过16S rRNA基因测序质控筛选后进行测序深度检测,Shannon稀释曲线显示曲线趋向平坦(图 1),说明本次测序数据量足够大,可以反映样本中绝大多数的微生物多样性信息。
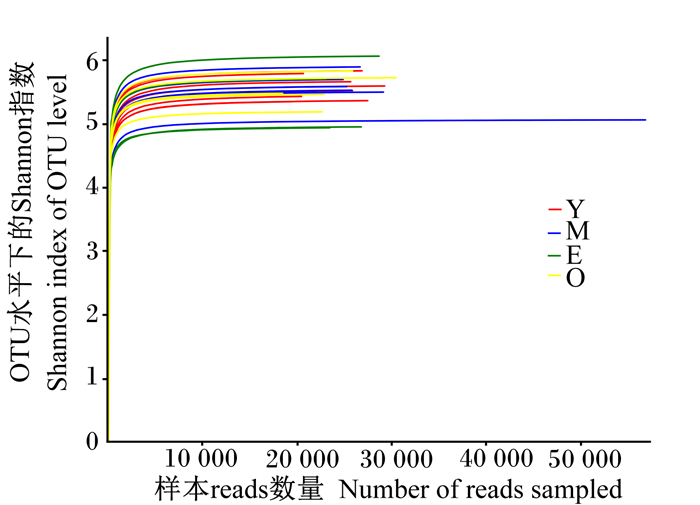
|
Y:Y组Y group(12月龄12 months of age,n=9);M:M组M group(3~5岁3 to 5 years of age,n=7);E:E组E group(6~8岁6 to 8 years of age,n=4);O:O组O group(9岁及以上more than 9 years of age,n=5)。下表和下图同the same as below tables and figures。 图 1 Shannon稀释曲线图(相似阈值为97%) Fig. 1 Shannon rarefaction curves (similarity threshold of 97%) |
不同年龄组奶水牛瘤胃菌群进行alpha多样性分析结果见表 2。Sobs指数随着年龄增长逐渐减少,其中Y组的操作分类单元(OTU)数最高,O组最低且与其他3组呈现差异显著的趋势(P=0.07)。多样性分析中Shannon指数E组略低于其他3个组,但是4个年龄组间统计差异不显著(P>0.05);Simpson指数E组比其他3组都略高,但是4个年龄组间统计差异不显著(P>0.05)。Ace和Chao指数随年龄增长逐渐下降(P < 0.05),Y组显著高于其他3组,而其他3组间均没有显著差异(P>0.05)。
|
|
表 2 奶水牛瘤胃菌群的alpha多样性分析 Table 2 Alpha diversity analysis of ruminal microflora of dairy buffalo |
对不同年龄组瘤胃菌群样本进行层级聚类(hierarchical clustering)分析,检测菌群组成的相似性和差异性。如图 2-A所示,可以看出4个年龄组瘤胃菌群组成有着不同差异程度的类群,尤其是Y组和M组菌群组成样本的组间差异与组内较为相似,而E组和O组呈现一定相似性而组内个体差异显著,表明不同年龄组水牛瘤胃菌群组成有一定差异性, 并且随着年龄增长,个体间瘤胃菌群组成的差异性增加(图 2-B)。
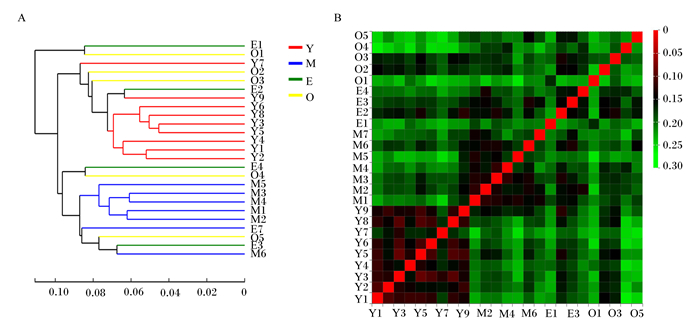
|
E1~4:E组样本samples in group E;O1~O5:O组样本samples in group O;Y1~Y9:Y组样本samples in group Y; M1~M6:M组样本samples in group M。 图 2 层级聚类分析(A)和群落Heatmap分析(B) Fig. 2 Hierarchical clustering analysis (A) and microflora Heatmap analysis (B) |
本试验共获得18个细菌门分类物种,其相对丰度分布见图 3。无论哪个年龄组,奶水牛瘤胃在门水平的优势菌群主要由拟杆菌门、厚壁菌门和变形菌门(Proteobacteria)组成,三者占整个菌群的89%以上;次级优势菌门是疣微菌门(Verrucomicrobia)、螺旋体菌门(Spirochaetae)、生糖菌门(Saccharibacteria)、柔膜菌门(Tenericutes)、蓝藻菌门(Cyanobacteria)、放线菌门(Actinobacteria)、黏胶球形菌门(Lentisphaerae)、迷踪菌门(Elusimicrobia)、纤维杆菌门、未分类菌门(unclassified_k_norank);其余相对丰度低于0.10%的门类被聚集到一起。
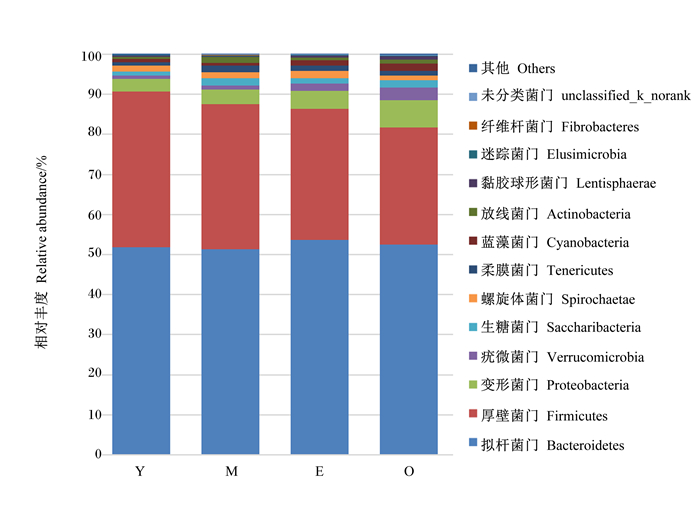
|
图 3 奶水牛瘤胃菌群门水平相对丰度 Fig. 3 Relative abundance of rumen microflora of dairy buffalo at phylum level |
在属水平上共鉴定出334个细菌属,其相对丰度分布见图 4。其中拟杆菌门下普雷沃氏菌属_1(Prevotella_1)是4个年龄组中相对丰度最高的优势菌属,次要优势菌属均低于10%,包括:理研菌科_RC9菌属(Rikenellaceae_RC9_gut_group)、未排位拟杆菌_BS11菌属(norank_f_Bacteroidales_BS11_gut_group)、克里斯滕森菌科_R-7菌属(Christensenellaceae_R-7_group)、未分类梭菌目菌属(unclassified_o_Clostridiale)、瘤胃球菌科_NK4A214菌属(Ruminococcaceae_NK4A214_group)、解琥珀酸弧菌属(Succiniclasticum)、瘤胃球菌属_2 (Ruminococcus_2)、乳酸菌属(Lactobacillus)等;其余相对丰度低于1%的菌属被聚集到一起。
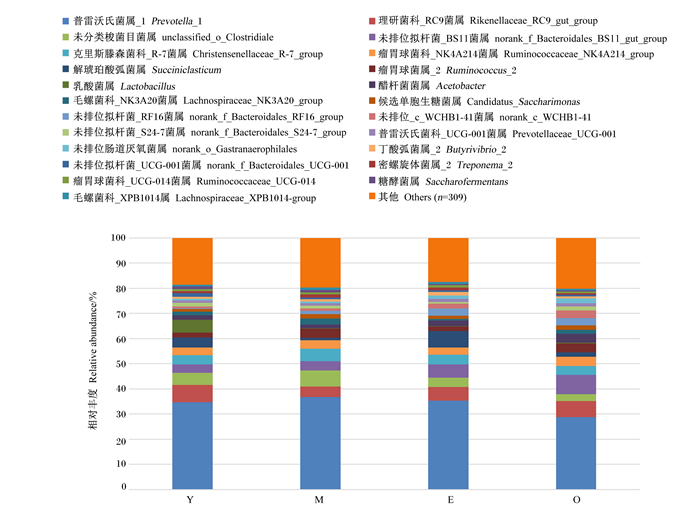
|
图 4 奶牛瘤胃菌群属水平相对丰度 Fig. 4 Relative abundance of rumen microflora of dairy buffalo at genus level |
进一步分析属水平下相对丰度在1%以上的菌属,发现拟杆菌门各菌属相对丰度不均匀极端分布,普雷沃氏菌属_1相对丰度最高,而其余菌属相对丰度都较低;年龄最高的O组高相对丰度和低相对丰度菌属分布较Y组和M组集中(图 5-A);而厚壁菌门下各菌属的相对丰度分布平均差距较小,即相对丰度比较聚集(图 5-B);其余次级优势菌属的分布,整体随着年龄增长相对丰度呈逐渐上升的趋势,且各菌属相对丰度分布越来越分散(图 5-C)。
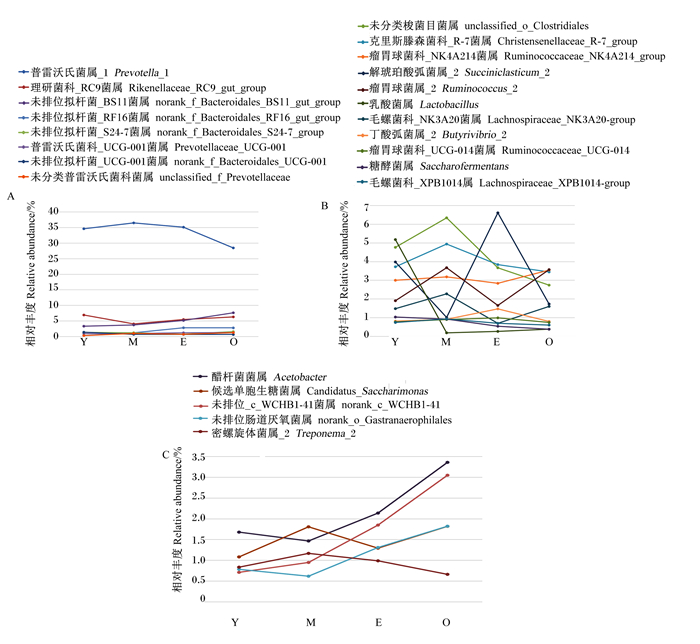
|
图 5 奶水牛瘤胃菌群拟杆菌门(A)、厚壁菌门(B)中菌属和其他优势菌属(C)相对丰度变化趋势 Fig. 5 Change of relative abundance of bacterial genus in Bacteroidetes (A), Firmicutes (B) and other dominant genera (C) in rumen microflora of dairy buffalo |
比较不同年龄奶水牛瘤胃细菌差异性发现,拟杆菌门、厚壁菌门、变形菌门、疣微菌门等门水平上相对丰度排前12位且占总数99%以上的菌群,在不同年龄组间没有显著差异(P>0.05);在属水平上,相对丰度排前25位的菌属主要是由拟杆菌门和厚壁菌门组成,其中20个菌属包括核心优势菌属普雷沃氏菌属_1、理研菌科_RC9菌属、未排位拟杆菌_BS11菌属等在4个年龄组间差异不显著(P>0.05)。通过LEfSe差异分析(LDA阈值为2)发现,在属水平下呈显著差异性的物种,Y组43个,M组29个,E组和O组分别有11和23个,差异物种随着年龄的增长逐渐减小,E组介于M组和O组之间,年龄比较接近两者,因此差异物种最少;此外,这些差异物种大多以次级优势菌为主(图 6)。
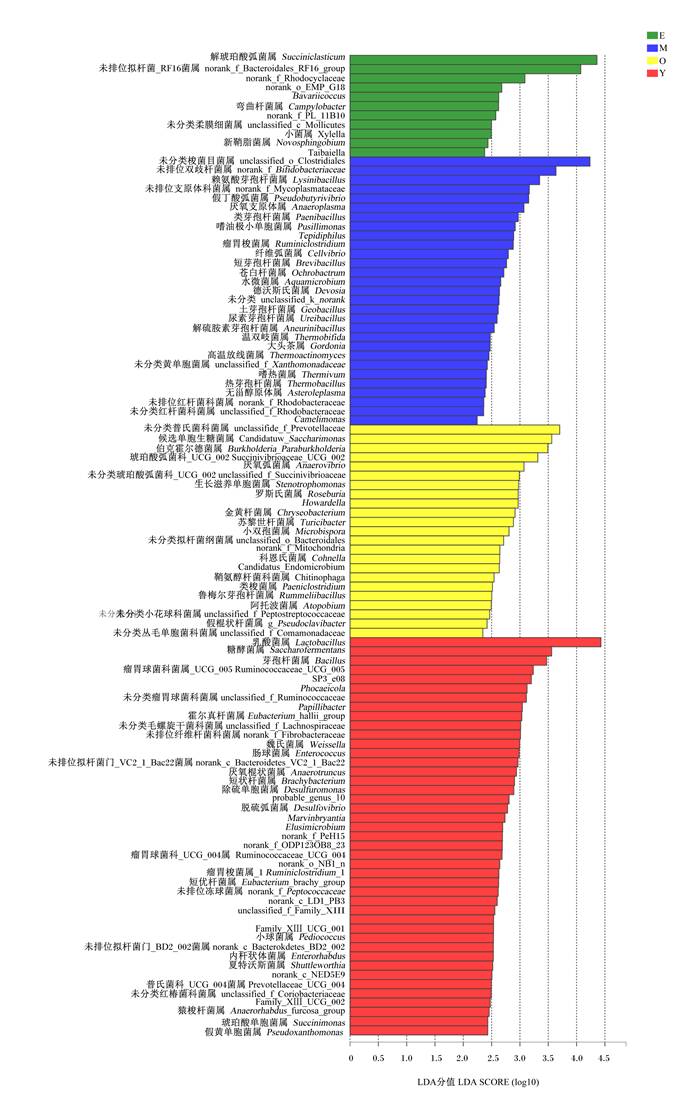
|
图 6 奶水牛瘤胃菌群LEfSe属水平上物种差异判别分析(LDA>2) Fig. 6 LEfSe analysis of rumen microflora of dairy buffalo at genus level (LDA>2) |
从图 6可知,在差异显著的前10位菌属中,主要是由优势菌门中的拟杆菌科和厚壁菌门梭菌科的非优势菌属组成,且厚壁菌门下差异菌属数量多;其中Y组相对丰度差异最大的特征菌属是乳酸菌属和糖酵菌属(Saccharofermentans)(P < 0.05),M组的差异效果最大的特征菌属是未分类梭菌目菌属和未排位双歧杆菌科菌属(g_norank_f_Bifidobacteriaceae)(P < 0.05),E组的特征菌属是解琥珀酸弧菌属和未排位拟杆菌_RF16菌属(norank_f_Bacteroidales_RF16_group)(P < 0.05),O组的特征菌属是未分类普雷沃氏菌科菌属(unclassified_f_Prevotellaceae)和候选单胞生糖菌属(Candidatus_Saccharimonas)(P < 0.05)。从年龄增长的分布变化中,厚壁菌门整体差异菌属随着年龄增长,相对丰度逐渐减少聚拢,而拟杆菌门和放线菌门的整体差异菌属则以Y组相对丰度低于其他年龄阶段,呈整体增加的趋势(图 7)。
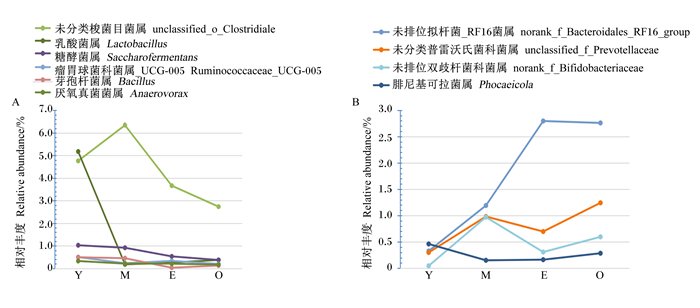
|
图 7 厚壁菌门(A)和拟杆菌门、放线菌门(B)中的菌属相对丰度变化趋势 Fig. 7 Change of relative abundance of bacterial genus in Firmicutes (A), Bacteroidetes and Actinobacteria (B) |
通过计算属水平菌属之间的相关,分析4个年龄组奶水牛瘤胃菌群属水平(相对丰度排前25位的菌属)上的相互关系发现,奶水牛瘤胃菌群不同属之间和同菌门菌属的相互作用随着年龄增长,其紧密性逐渐减少(图 8)。Y组在属水平上,有74个呈正相关,7个呈负相关关系;M组有47个呈正相关和7个呈负相关关系;E组正相关关系39个,负相关24个;O组则是20个正相关,22个负相关。
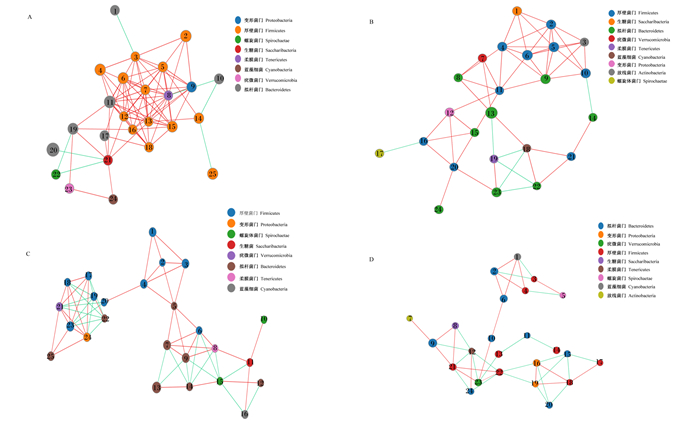
|
图A中菌属分别为bacterial genus in Fig. A were 1:普雷沃氏菌科_UCG-001菌属Prevotellaceae_UCG-001;2:乳酸菌属Lactobacillus;3:瘤胃球菌科_UCG-014菌属Ruminococcaceae_UCG-014;4:未分类梭菌目菌属unclassified_o_Clostridiales;5:毛螺旋菌科_NK3A20菌属Lachnospiraceae_NK3A20_group;6:克里斯滕森菌科_R-7菌属Christensenellaceae_R-7_group;7:糖酵菌属Saccharofermentans;8:未排位柔膜细菌_RF_9 norank_o_Mollicutes_RF9;9:醋杆菌属Acetobacter;10:未排位拟杆菌_UCG-001菌属norank_f_Bacteroidales_UCG-001:11:理研菌科_RC9菌属Rikenellaceae_RC9_gut_group;12:毛螺旋菌科_XPB1014菌属Lachnospiraceae_XPB1014_group;13:芽孢杆菌属Bacillus;14:瘤胃球菌属_2 Ruminococcus_2;15:瘤胃球菌科_NK4A214菌属Rumincoccaceae_NK4A214_group;16:瘤胃球菌科_UCG-010菌属Ruminococcaceae_UCG-010;17:未排位拟杆菌_S24-7菌属norank_f_Bacteroidales_S24-7_group;18:丁酸弧菌属_2 Butyrivibrio_2;19:未排位拟杆菌_BS11菌属norank_f_Bacteroidales_BS11_gut_group;20:普雷沃氏菌属_1 Prevotella_1;21:候选单胞生糖菌属Candidatus_Saccharimonas;22:密螺旋体菌属_2 Treponema_2;23:norank_c_WCHB1-41;24:norank_o_Gastranaerophilales;25:解琥珀酸弧菌属Succiniclasticum。图B中菌属分别为bacterial genus in Fig. B were 1:候选单胞生糖菌属Candidatus_Saccharimonas;2:瘤胃球菌科_NK4A214菌属Ruminococcaceae_NK4A214_group;3:未排位双歧杆菌科菌属norank_f_Bifidobacteriaceae;4:毛螺旋菌科_NK3A20菌属Lachnospiraceae_NK3A20_group;5:克里斯滕森菌科_R-7菌属Christensenellaceae_R-7_group;6:未分类梭菌目菌属unclassified_o_Clostridiales;7:norank_c_WCHB1-41;8:未分类普雷沃氏菌科菌属unclassified_f_Prevotellaceae;9:理研菌科_RC9菌属Rikenellaceae_RC9_gut_group;10:瘤胃球菌属_2 Ruminococcus_2;11:丁酸弧菌属_2 Butyrivibrio_2;12:醋杆菌属Acetobacter;13:普雷沃氏菌属_1 Prevotella_1;14:普雷沃氏菌科_UCG-001菌属Prevotellaceae_UCG-001;15:未排位拟杆菌_RF-16菌属norank_f_Bacteroidales_RF16_group;16:毛螺旋菌科_XPB1014菌属Lachnospiraceae_XPB1014_group;17:密螺旋体菌属_2 Treponema_2;18:norank_o_Gastr anaerophilales; 19:未排位柔膜细菌_RF_9 norank_o_Mollicutes_RF9:20:瘤胃球菌科_UCG-014菌属Ruminococcaceae_UCG-014;21:解琥珀酸弧菌属Succiniclasticum;22:未排位拟杆菌_BS11菌属norank_f_Bacteroidales_BS11_gut_group;23:未排位拟杆菌_UCG-001菌属norank_f_Bacteroidales_UCG-001;24:未排位拟杆菌_S24-7菌属norank_f_Bacteroidales_S24-7_group。图C中菌属分别为bacterial genus in Fig. C were 1:未分类梭菌目菌属unclassified_o_Clostridiales;2:毛螺旋菌科_XPB1014菌属Lachnospiraceae_XPB1014_group;3:克里斯滕森菌科_R-7菌属Christensenellaceae_R-7_group;4:瘤胃球菌科_NK4A214菌属Ruminococcaceae_NK4A214_group;5:未排位拟杆菌_S24-7菌属norank_f_Bacteroidales_S24-7_group;6:瘤胃球菌科_UCG-014菌属Ruminococcaceae_UCG-014;7:理研菌科_RC9菌属Rikenellaceae_RC9_gut_group:8:未排位柔膜细菌_RF_9 norank_o_Mollicutes_RF9;9:未排位拟杆菌_BS11菌属norank_f_Bacteroidales_BS11_gut_group;10:Sphaerochaeta;11:候选单胞生糖菌属Candidatus_Saccharimonas;12:未排位拟杆菌_UCG-001菌属g_norank_f_Bacteroidales_UCG-001;13:普雷沃氏菌属_1 Prevotella_1;14:普雷沃氏菌科UCG-001菌属Prevotellaceae_UCG-001;15:密螺旋体菌属_2 Treponema_2;16:norank_o_Gastranaerophilales;17:丁酸弧菌属_2 Butyrivibrio_2;18:韦永氏秋菌科_UCG_001 Veillonellaceae_UCG-001;19:瘤胃球菌属_2 Ruminococcus_2;20:毛螺旋菌科_NK3A20菌属Lachnospiraceae_NK3A20_group;21:norank_c_WCHB1-41B;22:未分类普雷沃氏菌科菌属unclassified_f_Prevotellaceae;23:解琥珀酸弧菌属Succiniclasticum;24:醋杆菌属Acetobacter;25:未排位拟杆菌_RF-16菌属norank_f_Bacteroidales_RF16_group。图D中菌属分别为bacterial genus in Fig. D were 1:norank_o_Gastranaerophilales;2:未排位拟杆菌_BS11菌属norank_f_Bacteroidales_BS11_gut_group;3:瘤胃球菌科_UCG-014菌属Ruminococcaceae_UCG-014;4:解琥珀酸弧菌属Succiniclasticum;5:密螺旋体菌属_2 Treponema_2;6:理研菌科_RC9菌属Rikenellaceae_RC9_gut_group;7;未排位双歧杆菌科菌属norank_f_Bifidobacteriaceae;8:候选单胞生糖菌属Candidatus_Saccharimonas;9:普雷沃氏菌属_1 Prevotella_1;10:未排位拟杆菌_RF-16菌属norank_f_Bacteroidales_RF16_group;11:未排位拟杆菌_UCG-001菌属norank_f_Bacteroidales_UCG-001;12:未排位柔膜细菌_RF_9 norank_o_Mollicutes_RF9;13:瘤胃球菌科_NK4A214菌属Ruminococcaceae_NK4A214_group;14:瘤胃球菌属_2 Ruminococcus_2;15:未排位拟杆菌_S24-7菌属norank_f_Bacteroidales_S24-7_group;16:醋杆菌属Acetobacter;17:g_Veillonellaceae_UCG-001;18:丁酸弧菌属_2 Butyrivibrio_2;19:丛毛单胞菌属Comamonas;20:普雷沃氏菌科_UCG-001菌属Prevotellaceae_UCG-001;21:未分类梭菌目菌属unclassfied_o_Clostridiales;22:克里斯滕森菌科_R-7菌属Christensenellaceae_R-7_group;23:g_norank_c_WCHB1-41;24:未分类普雷沃氏菌科菌属unclassified_f_Prevotellaceae。图A、B、C、D分别为Y组、M组、E组以及O组;红线表示正相关,蓝线表示负相关A, B, C and D represent groups Y, M, E and O, respectively. Red and blue lines mean positive and negative correlation, respectively. 图 8 奶水牛瘤胃菌群属水平物种相关性网络图(相关系数阈值为0.5) Fig. 8 Co-occurrence analysis of rumen microflora of dairy buffalo at genus level (correlation coefficient threshold of 0.5) |
alpha多样性分析表明,检测到的实际总菌数量(Sobs指数)在9岁及以上年龄的奶水牛组显著低于其他3个年龄组,呈依次递减的趋势;瘤胃细菌丰度的Ace指数和Chao指数表明,12月龄奶水牛瘤胃菌群相对丰度显著高于9岁或以上的瘤胃菌群,并呈现依次递减趋势。4个年龄组在Shannon指数上差异不显著,而Simpson指数结果显示9岁及以上组的多样性显著低于其他3个年龄组,两者结果不尽一致,可能是Shannon指数与Simpson指数侧重点不一样,前者对于稀疏物种敏感,后者对于优势物种比较敏感,即年长宿主瘤胃菌群优势物种的多样性较低。本试验中,12月龄和3~5岁奶水牛的Chao和Shannon指数与逄宾宾等[4]报道的健康中国荷斯坦奶牛18和30月龄奶牛瘤胃菌群以及和郭伟[9]报道的不同年龄山羊瘤胃菌群多样性研究的结果一致;本试验随年龄增长多样性增加的结果与其他物种如小鼠、恒河猴和大熊猫等[9-11]肠道微生物的结果也一致,可能是由于瘤胃等消化道微生物在发育阶段竞争机制导致一些菌群丰度减少甚至消失,进而导致多样性减少;年长宿主菌群多样性的增加有可能是宿主自身机能和免疫功能等下降从而增加了外来细菌定植[12-14]。
beta多样性分析结果表明,12月龄、3~5岁、9岁及以上年龄奶水牛瘤胃菌群组成结构具有一定的年龄特征性,并且随着年龄增长个体之间瘤胃菌群的结构差异性也在增大。12月龄与3~5岁年龄组奶水牛瘤胃菌群结构的相似性要大于年龄稍长的2个年龄组,可能因为后2组间的年龄跨度较大所造成的,也有可能是随着宿主年龄的增长个体间的差异增大,从而导致瘤胃菌群结构差异性增大,与许多反刍动物和人的研究结果相似,说明不同物种消化道菌群结构随着年龄增长的变化规律具有一定相似性[4-11, 15]。
本研究发现,4个年龄组的奶水牛瘤胃菌群相对丰度位于前3位的优势菌门依次是拟杆菌门、厚壁菌门和变形菌门,这与前人报道结果[4-5, 8]基本一致。本试验还发现,不同年龄组瘤胃细菌相对丰度存在一定差异和波动,其中优势菌属中厚壁菌门随着年龄增加其菌属数量逐渐降低,变形菌门相对丰度则随年龄增加而增多,这与逄宾宾等[4]在中国荷斯坦奶牛上发现的结果相似,但是与Jami等[5]和Li等[16]有关牛从出生到断奶期间瘤胃菌群的变化趋势相反。大量文献已经证实,变形菌门下含有一些致病菌,能够反映微生态失调或不稳定的肠道微生物群落组成结构[16-18]。Co-occurrence物种相关性分析发现,随着奶水牛年龄增长其瘤胃菌群之间的稳定性逐渐减弱,可能跟变形菌门相对丰度随年龄增加而增加有一定关联性。
在属水平上,普雷沃氏菌属_1是4个年龄组奶水牛瘤胃中相对丰度最高的优势菌属,12月龄奶水牛瘤胃的优势菌属从高到低依次是理研菌科_RC9菌属、克里斯滕森菌科_R-7菌属和乳酸菌菌属等;3~5岁奶水牛瘤胃优势菌属有未分类梭菌目菌属、克里斯滕森菌科_R-7菌属和理研菌科_RC9菌属等;6~8岁奶水牛瘤胃优势菌属为解琥珀酸弧菌属、理研菌科_RC9菌属和未排位拟杆菌_BS11菌属;9岁及以上奶水牛瘤胃优势菌属还有未排位拟杆菌_BS11菌属、克里斯滕森菌科_R-7菌属和理研菌科_RC9菌属等,而逄宾宾等[4]发现,3月龄荷斯坦奶牛瘤胃优势菌属是普雷沃氏菌属_1、毛螺旋科_NK3A20菌属(Lachnospiraceae_NK3A20)、罗氏菌属(Rothia); 而30月龄牛瘤胃中这3个菌属相对丰度下降,优势菌属是普雷沃氏菌属_1、未分类琥珀酸弧菌科_UCG-002菌属(unclassified_f_Succinivibrionaceae_UCG-002)、丹毒斯菌菌属(Erysipelothrix rosenbach)等;郭伟[9]发现不同年龄山羊的瘤胃优势菌属是普雷沃氏菌属、地杆菌属(Geobacter)、埃希氏杆菌属(Escherichia/Shigella)、帕拉普氏杆菌属(Paraprevotella)等,说明不同反刍动物的瘤胃优势菌群存在一定差异。
本试验中厚壁菌门下的乳酸菌属相对丰度从12月龄到3~5岁龄急剧减少,之后一直维持较低水平。乳酸菌属被大量研究报道作为人体益生菌和衰老的标记微生物[19-21],其在12月龄阶段相对丰度显著高于其他阶段,与衰老肠道微生态变化研究报道[22]一致。本试验发现,3~5岁奶水牛瘤胃特征有益菌是未排位双歧杆菌属等菌属,与Takagi等[22]报道结果一致,这可能是由于双歧杆菌对不同年龄组人肠黏液的黏附力差异所致[23]。9岁及以上奶水牛瘤胃菌群的差异性特征物种有未分类普雷沃氏菌科菌属、候选单胞生糖菌属和未分类琥珀酸弧菌科_UCG-002等,其中未分类琥珀酸弧菌科_UCG-002在逄宾宾等[4]研究中是30月龄牛瘤胃中的优势菌种;还有Wongwilaiwalin等[24]报道,降解木质纤维素的微生物由拟杆菌门和厚壁菌门下的菌属组成,本试验差异显著的物种也集中在由优势菌门中的拟杆菌科和厚壁菌门梭菌科的非优势菌属组成,这说明这种年龄变化的差异有可能也体现了奶水牛瘤胃微生物降解木质纤维素能力的变化。
物种间相关性分析发现,随着奶水牛年龄增长其瘤胃菌群之间相互作用的数量和频率逐渐减少,负相关的物种数量逐渐增加,表明物种间的紧密性逐渐下降。从瘤胃菌群生态系统的稳定性来说,物种之间的相关性越紧密越复杂,菌群结构越稳定,其功能也越健全,生产能力越高,同时整个菌群系统的自身调节能力也越高,反之亦然。本研究中发现随年龄增加,瘤胃优势菌相对丰度和有益菌相对丰度下降、无益菌数量和丰度增加,提示随年龄增加瘤胃菌群结构稳定性减弱。
4 结论随着奶水牛年龄增长,其瘤胃菌群数量有显著下降的趋势,相对丰度显著减少,优势菌群和亚优势菌群呈现一定的年龄特征变化;奶水牛瘤胃存在着一些年龄特征菌属,其菌群结构紧密性随年龄增加而减弱,表明宿主年龄增长可影响瘤胃菌群结构和功能的稳定。
| [1] |
梁停停, 刘文丽, 许浩, 等. 水牛瘤胃微生物研究进展[J]. 中国奶牛, 2017(2): 1-5. |
| [2] |
张慧敏, 夏海磊, 黄强, 等. 海子水牛瘤胃微生物的宏基因组学分析[J]. 动物营养学报, 2017, 29(11): 4151-4161. DOI:10.3969/j.issn.1006-267x.2017.11.039 |
| [3] |
PANDYA P R, SINGH K M, PARNERKAR S, et al. Bacterial diversity in the rumen of Indian Surti buffalo (Bubalus bubalis), assessed by 16S rDNA analysis[J]. Journal of Applied Genetics, 2010, 51(3): 395-402. DOI:10.1007/BF03208869 |
| [4] |
逄宾宾, 王雨, 杨妍, 等. 健康中国荷斯坦奶牛不同发育阶段瘤胃的菌群多样性研究[J]. 黑龙江畜牧兽医, 2018(5): 14-18, 261. |
| [5] |
JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [6] |
GUO W, LI Y, WANG L Z, et al. Evaluation of composition and individual variability of rumen microbiota in yaks by 16S rRNA high-throughput sequencing technology[J]. Anaerobe, 2015, 34: 74-79. DOI:10.1016/j.anaerobe.2015.04.010 |
| [7] |
刘凯珍, 王立志.成年和老年牦牛瘤胃古菌结构与组成的差异性研究[C]//中国畜牧兽医学会动物营养学分会第十二次动物营养学术研讨会论文集.武汉: 中国畜牧兽医学会动物营养学分会, 2016.
|
| [8] |
SHARMA B G, BASU S, SHARMA M M. Characterization of adsorbed ionic sufactants on a mica substrate[J]. Langmuir, 1996, 12: 6506-6512. DOI:10.1021/la960537j |
| [9] |
郭伟.不同年龄阶段山羊瘤胃古菌, 细菌菌群结构组成研究[D].硕士学位论文.雅安: 四川农业大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10626-1016049261.htm
|
| [10] |
杨展.衰老肠道微生态的变化及干预措施研究[D].博士学位论文.北京: 中国人民解放军军事医学科学院, 2017. http://cdmd.cnki.com.cn/Article/CDMD-90106-1017230456.htm
|
| [11] |
朱华, 肖冲, 尚海泉, 等. 基于高通量测序的不同年龄恒河猴肠道菌群结构差异分析[J]. 中国实验动物学报, 2019, 27(1): 72-78. DOI:10.3969/j.issn.1005-4847.2019.01.012 |
| [12] |
KUMAR M, BABAEI P, JI B Y, et al. Human gut microbiota and healthy aging:recent developments and future prospective[J]. Nutrition and Healthy Aging, 2016, 4(1): 3-16. DOI:10.3233/NHA-150002 |
| [13] |
CLAESSON M J, CUSACK S, O'SULLIVAN O, et al. Composition, variability, and temporal stability of the intestinal microbiota of the elderly[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl.1): 4586-4591. |
| [14] |
MARTINEZ-MEDINA M, DENIZOT J, DREUX N, et al. Western diet induces dysbiosis with increased E.coli in CEABAC10 mice, alters host barrier function favouring AIEC colonisation[J]. Gut, 2014, 63(1): 116-124. DOI:10.1136/gutjnl-2012-304119 |
| [15] |
王晓艳.成年与老年大熊猫肠道菌群16S rDNA-RFLP技术分析[D].硕士学位论文.雅安: 四川农业大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10626-1014102951.htm
|
| [16] |
LI R W, CONNOR E E, LI C J, et al. Characterization of the rumen microbiota of pre-ruminant calves using metagenomic tools[J]. Environmental Microbiology, 2011, 14(1): 129-139. |
| [17] |
DEVKOTA S, WANG Y W, MUSCH M W, et al. Dietary-fat-induced taurocholic acid promotes pathobiont expansion and colitis in IL10-/-mice[J]. Nature, 2012, 487(7405): 104-108. DOI:10.1038/nature11225 |
| [18] |
BYNDLOSS M X, BÄUMLER A J. The germ-organ theory of non-communicable diseases[J]. Nature Reviews Microbiology, 2018, 16(2): 103-110. DOI:10.1038/nrmicro.2017.158 |
| [19] |
BÄCKHED F, DING H, WANG T, et al. The gut microbiota as an environmental factor that regulates fat storage[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(44): 15718-15723. DOI:10.1073/pnas.0407076101 |
| [20] |
ZHANG C H, LI S F, YANG L, et al. Structural modulation of gut microbiota in life-long calorie-restricted mice[J]. Nature Communications, 2013, 4: 2163. DOI:10.1038/ncomms3163 |
| [21] |
YATSUNENKO T, REY F E, MANARY M J, et al. Human gut microbiome viewed across age and geography[J]. Nature, 2012, 486(7402): 222-227. DOI:10.1038/nature11053 |
| [22] |
TAKAGI T, NAITO Y, INOUE R, et al. Differences in gut microbiota associated with age, sex, and stool consistency in healthy Japanese subjects[J]. Journal of Gastroenterology, 2019, 54(1): 53-63. DOI:10.1007/s00535-018-1488-5 |
| [23] |
TⅡHONEN K, OUWEHAND A C, RAUTONEN N. Human intestinal microbiota and healthy ageing[J]. Ageing Research Reviews, 2010, 9(2): 107-116. DOI:10.1016/j.arr.2009.10.004 |
| [24] |
WONGWILAIWALIN S, LAOTHANACHAREON T, MHUANTONG W, et al. Comparative metagenomic analysis of microcosm structures and lignocellulolytic enzyme systems of symbiotic biomass-degrading consortia[J]. Applied Microbiology and Biotechnology, 2013, 97(20): 8941-8954. DOI:10.1007/s00253-013-4699-y |




