瘤胃作为一个相对独立、复杂的生态系统,不仅仅关系到山羊的消化道健康,更关系到山羊整体的各个方面。瘤胃内微生物具有高度的特异性和广泛性,它们不仅起到帮助机体消化、吸收营养物质,还能够增强机体抵抗力,对外来入侵的病原体进行抵抗[1]。酸中毒状态下,有关反刍动物的血液代谢、饲料效率等方面的研究一直都是反刍动物营养代谢研究的热点[2-4]。近年来,具有典型症状的急性瘤胃酸中毒在实际生产中仍较为常见,亚急性瘤胃酸中毒也时有发生,且不同的反刍动物个体之间存在较大的变异性[5]。随着高能、高淀粉饲粮成为奶牛泌乳阶段或肉用反刍动物育肥阶段的典型营养特性[6-7],有关瘤胃微生态的研究更加具有实际意义。现阶段,关于动物和人类的肠道微生态研究不断深入,肠道菌群的功能不断扩展。与此同时,肠道微生态制剂的研制更是层出不穷。本试验通过构建山羊急性瘤胃酸中毒模型,研究在瘤胃亚健康状态下菌群结构与组成的变化,丰富瘤胃细菌在疾病下的生存、繁殖规律,为反刍动物肠道微生态制剂的开发提供依据。
16S rRNA是细菌一段编码特殊rRNA的DNA序列,其序列包含9个可变区和10个保守区,几乎存在于所有细菌的基因组中,在不同细菌种类间具有高度的保守性和特异性,其可变区因细菌而异,且变异程度与细菌的系统发育程度密切相关,因此被人类广泛用于细菌的鉴别。伴随着PCR技术的普及,16S rRNA测序技术在分析微生物和生态系统多样性中以其准确性较好、测序较为全面且价格低廉等优势被广泛应用。本试验采用16S rRNA测序技术,对瘤胃液中的细菌进行测序,分析山羊瘤胃由健康向酸中毒状态转变时瘤胃内菌群组成与结构的变化,以期为探究疾病状态下瘤胃细菌的繁殖规律、完善瘤胃微生态理论提供依据。
1 材料与方法 1.1 试验动物与模型建立选取10~14月龄健康波尔山羊4只,所有试验羊每天按早、中、晚分3次饲喂,粗料以花生秸秆为主,辅以玉米秸秆和苹果渣等,同时每天每只羊补饲0.4 kg精料,所有试验羊自由饮水,自由采食,定期防疫驱虫。造模前颈静脉采血分离血清进行造模前血气分析。通过灌服玉米面构建山羊急性瘤胃酸中毒模型,以40 g/kg BW的比例确定玉米面用量,灌服时用温水将玉米面混合成糊状。瘤胃插管后,先采集瘤胃液再进行玉米面灌服。灌服结束后,密切观察山羊的临床表现。在灌服后6~12 h,山羊逐渐出现酸中毒症状,立即进行第2次颈静脉采血,分离血清做血气分析。
1.2 试验设计与样品采集试验采用自身前后对照试验设计,变化因素为是否发生急性瘤胃酸中毒。瘤胃液的采集共有2个时间点:第1个时间点为造模前山羊正常状态下采集瘤胃液;第2个时间点为造模后,综合山羊病史、临床表现和粪尿、血液的检测结果,确诊山羊发生急性酸中毒后,立即进行的瘤胃液采集。每个时间点每只山羊使用灭菌离心管收集瘤胃液40 mL,保存于-20 ℃,共8个样本。将造模前采集的4份瘤胃液设为健康组,造模后采集的4份瘤胃液设为瘤胃酸中毒组。
1.3 16S rRNA测序及数据分析首先对样品瘤胃液中菌群的总DNA进行提取,质检合格后,针对细菌16S rRNA基因的V4区进行PCR扩增。之后对PCR产物进行纯化,完成建库。对文库的片段范围及浓度进行检测,检测合格的文库在HiSeq平台进行测序。下机数据经过数据过滤,滤除低质量的reads,剩余高质量的Clean data用于后期分析[8];通过reads之间的Overlap关系将reads拼接成Tags,序列拼接使用FLASH软件[9];在给定的相似度下,利用软件USEARCH(v7.0.1090)[10]将Tags聚成操作分类单元(OTU),将OTU与数据库比对并进行物种注释;基于OTU和物种注释结果进行样品物种复杂度分析以及组间物种差异分析。
1.4 数据统计数据的显著性关系使用GraphPad prism 7.0软件进行分析,以平均值±标准差(mean±SD)表示。采用t检验建立统计学差异,P < 0.05表示健康组与瘤胃酸中毒组之间差异显著。
2 结果与分析 2.1 造模成功的判定在灌服玉米面后的6~12 h,4只山羊先后出现精神沉郁,卧地不起且头部向后回转贴于腹部;呼吸和脉搏加快,鼻镜和皮肤干燥,眼窝出现不同程度地凹陷,食欲减退甚至废绝,反刍减少甚至停止,有磨牙虚嚼;瘤胃蠕动次数减少,有的蠕动停止,瘤胃内容物胀满松软,拍动有水声回响;粪便呈现如下的变化:正常颗粒状粪便到粪便稀软,再到不成形的糊状便,最后发展为稀水状粪便。表 1检测结果显示,瘤胃液与粪便、尿液pH下降至5.0~6.5,瘤胃液的pH下降幅度较大;显微镜观察发现瘤胃内纤毛虫活力明显下降,数量明显减少,甚至无纤毛虫。对有以上症状的山羊进行血气分析,结果见表 2。pH < 7.32,提示酸血症;碳酸氢根(HCO3-)浓度下降至低于正常范围下限,提示代谢性酸中毒;剩余碱(BE)低于正常范围下限,同样提示代谢性酸中毒;二氧化碳总量(TCO2)下降则提示机体碱储损失。
|
|
表 1 山羊瘤胃液与粪便、尿液pH测定结果 Table 1 Determination results of pH of rumen fluid, feces and urine of goats |
|
|
表 2 山羊血气分析结果 Table 2 Analysis results of blood-gas of goats |
根据4只山羊的病史、临床表现及瘤胃液、粪便、尿液、血液的检测结果,确诊4只山羊均发生了急性瘤胃酸中毒,玉米面成功诱导了山羊的急性瘤胃酸中毒。
2.2 测序结果及合理性通过测序共获得377 614个有效序列,利用UPARSE在97%相似度下进行聚类,获得每个样本的OTU数。8个样本共产生1 685个OTU。如图 1所示,健康组有1 606个OTU,瘤胃酸中毒组有1 427个OTU,2组共享1 348个OTU。
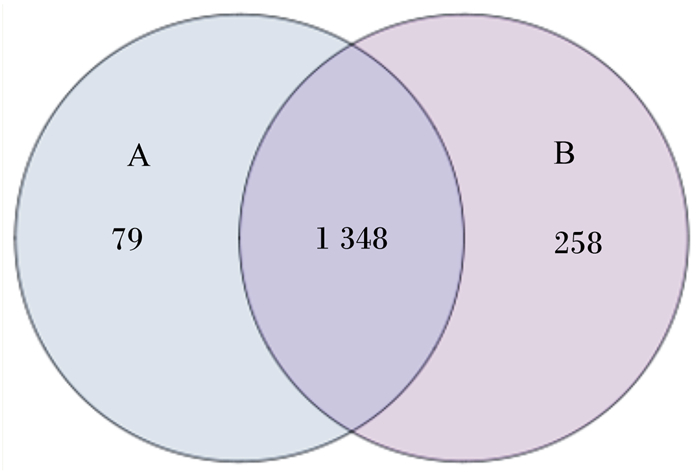
|
图 1 基于样本OTU的Venn图 Fig. 1 Venn diagram based on sample OTU |
样本稀释曲线如图 2-A所示,随着试验测序深度逐渐增加,各条曲线趋于平缓,说明该测序量合理,可以满足覆盖样本中的大多数微生物,继续增加测序量产生的新的OTU较少。结合所有样本的覆盖度曲线(图 2-B),充分表明本试验中对于样本的测序量及测序深度合理,更深入的测序对瘤胃液中检出新的OTU贡献很小。
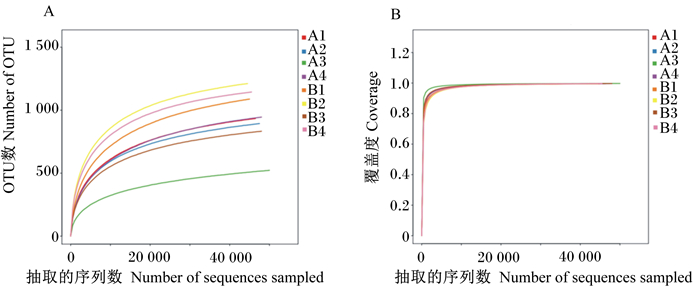
|
A:样本测序的稀释曲线(基于OTU,97%相似性);B:样本测序的覆盖度曲线。 A: dilution curve of sequencing samples (based on OTU, 97% similarity); B: coverage curve of sequencing samples. 图 2 样本测序合理性 Fig. 2 Rationality of sample sequencing |
Alpha多样性是对样本中物种丰富度和多样性进行分析,主要包括Observed species指数、Chao1指数、Ace指数、Shannon指数和Simpson指数。前3个指数可以反映样品中群落的丰富度,数值越大说明物种数量越多,即丰富度越高;后2个指数表示群落的多样性,Shannon指数越大、Simpson指数越小表明多样性越高[11]。如表 3所示,健康组和瘤胃酸中毒组之间Observed species指数、Chao1指数、Ace指数、Simpson指数差异不显著(P>0.05),但是健康组的Observed species指数、Ace指数、Chao1指数在数值上大于瘤胃酸中毒组,Simpson指数在数值上小于瘤胃酸中毒组。健康组的Shannon指数显著大于瘤胃酸中毒组(P < 0.05)。
|
|
表 3 Alpha多样性指数 Table 3 Alpha diversity indexes (n=4) |
根据各样本丰度差异的统计结果,基于遗传距离矩阵,采用类平均法(unweighted pair group method with arithmetic mean, UPGMA)对样本进行聚类分析,如图 3所示。图中相同颜色的标号表示属于同一个分组。样本越靠近,枝长越短,说明2个样本的物种组成越相似。瘤胃酸中毒组的4个样本依次聚类在一起,形成一大簇。健康组中A1、A2、A4这3个样本聚类为1个大簇,A3样品单独聚类为1个大簇。瘤胃酸中毒组和健康组的样本各自聚类在一起,彼此之间无交叉,表明瘤胃酸中毒组和健康组样本的物种组成存在差异,组内各样本之间相似性较高。
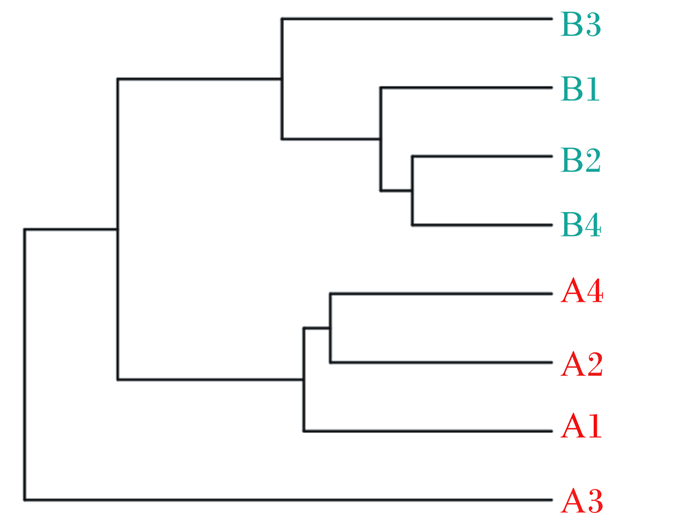
|
图 3 样品间物种组成聚类分析 Fig. 3 Cluster analysis of species composition among samples |
PCoA是基于样本间的距离矩阵来寻找主坐标的一种统计分析方法,通过PCoA可以观察到个体或者群体之间的差异。本试验采用加权的方式进行计算,即保留物种丰度特征。由图 4可知,健康组的样本依据其所在组别而相互聚类在一起,说明健康组的组内相似性比组间相似性高;而瘤胃酸中毒组的样本却没有依组别而发生组内聚集。健康组的组内个体聚集度相对高于瘤胃酸中毒组,表明健康组的菌群组成相似性高于瘤胃酸中毒组。
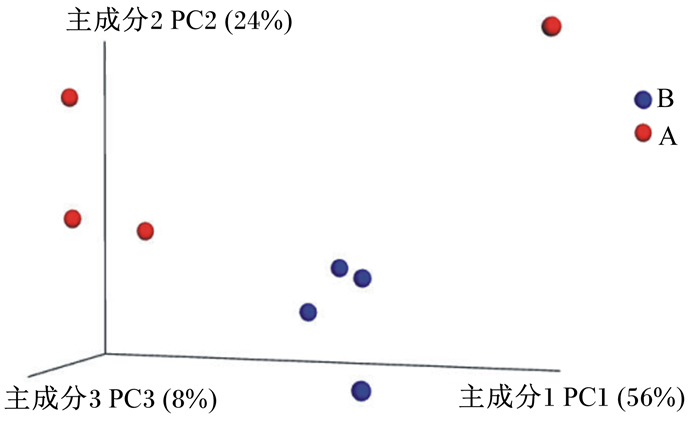
|
图 4 主坐标分析 Fig. 4 PCoA |
将试验得到的有效序列在不同分类水平上进行物种注释及组间秩和检验统计分析。如图 5所示,在门水平上共注释得到23个菌门,相对丰度都在0.1%以上。拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为2组中的优势菌门。其中,拟杆菌门是2组中共享的优势菌门。瘤胃酸中毒组中A1样本产生了相对丰度较高的放线菌门(Actinobacteria)。对比可得,拟杆菌门、纤维杆菌门(Fibrobacteres)相对丰度在发生急性瘤胃酸中毒后出现下降,厚壁菌门、螺旋体门(Spirochaetes)相对丰度则有所上升。经统计学分析,门水平上产生的22个菌门(不包含未分组)的相对丰度在2个组之间的差异均不显著(P>0.05)。
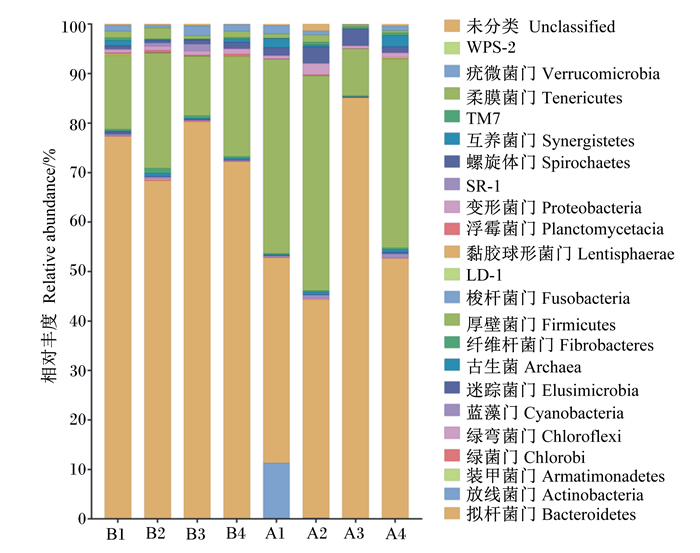
|
图 5 门水平上物种注释 Fig. 5 Species annotation at phylum level |
从纲水平开始,将相对丰度低于0.5%的物种全部合并成一类,不单独进行注释。健康组和瘤胃酸中毒组在纲、目、科、属水平上相对丰度存在显著差异的物种见表 4。
|
|
表 4 纲、目、科、属水平上物种相对丰度统计 Table 4 Relative abundance statistics of species at class, order, family and genus level (n=4) |
在纲水平上共注释得到45个菌纲,其中相对丰度大于0.5%的有16个。统计发现,瘤胃酸中毒组的芽孢杆菌纲(Bacilli)、蓝藻菌纲(Chloroplast)、γ-变形菌纲(Gammaproteobacteria)相对丰度显著高于健康组(P < 0.05)。
在目水平上共注释得到77个菌目,相对丰度大于0.5%的有15个。统计发现,瘤胃酸中毒组中隶属于厚壁菌门的乳酸杆菌目(Lactobacillales)和变形菌门下的气单胞菌目(Aeromonadales)相对丰度显著高于健康组(P < 0.05);瘤胃酸中毒组中疣微菌门下的LD1-PB3和变形菌门下的GMD14H09相对丰度显著低于健康组(P < 0.05)。
在科水平上共注释得到111个菌科,相对丰度大于0.5%的有22个。统计发现,厚壁菌门下的链球菌科(Streptococcaceae)和变形菌门下的Mitochondria、琥珀酸弧菌科(Succinivibrionaceae)在瘤胃酸中毒组中的相对丰度显著高于健康组(P < 0.05);隶属于疣微菌门(Verrucomicrobia)的WCHB1-25在健康组中的相对丰度显著高于瘤胃酸中毒组(P < 0.05)。
在属水平上共注释得到171个菌属,但由于属种类众多且部分属相对丰度太低,仅对相对丰度大于0.5%的21个属进行分析。统计发现,瘤胃酸中毒组中厚壁菌门下的链球菌属(Streptococcus)、Sharpea和螺旋体门下的密螺旋体属(Treponema)相对丰度显著大于健康组(P < 0.05)。
2.5.2 物种分类热图为了更好地了解样本内细菌的相对丰度变化情况,在属的水平上,对21种相对丰度大于0.5%的菌属进行双向聚类分析,结果显示在图 6。
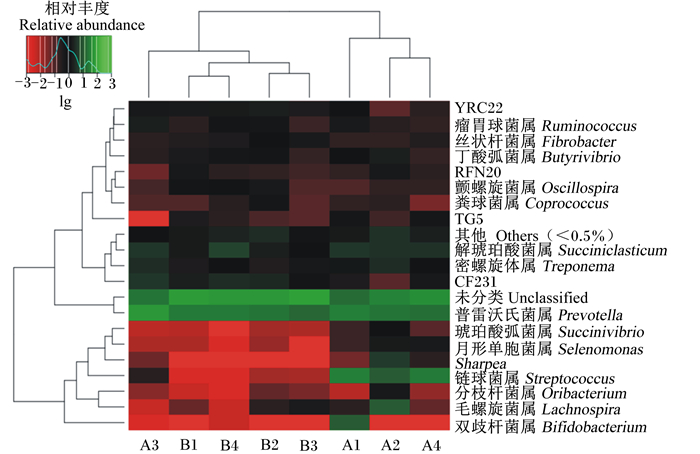
|
图 6 属水平上物种分类热图 Fig. 6 Thermal map of species classification at genus level |
由图 6可知,在纵向上,8个样本可分为2个大簇,左数第1大簇包含5个样本(A3、B1、B4、B2、B3),左数第2大簇包含3个样本(A1、A2、A4)。除A3以外,其余各个样本依据其所属组别相互聚类在一起,表明组内物种相似性高于组间物种相似性。由系统发育树可知,瘤胃酸中毒组中A2和A4聚类为一簇,健康组中B1和B4、B2和B3聚为一簇,它们之间的物种相似度高于同组内的其他样本。整体来看,在瘤胃发生酸中毒之后,瘤胃内容物菌群相对丰度在各个样本之间差异较大。
在横向上,21个菌属以及相对丰度低于0.5%的其他菌属,共计22个分类单元,可被分为3个大簇。未分类菌属和拟杆菌门下的普雷沃氏菌属(Prevotella)聚为中间的1个大簇,在其上方和下方的菌属分别构成1个大类。其中,在其下方的大类中,主要是由隶属于厚壁菌门的各个菌属所组成,包括月形单胞菌属(Selenomonas)、Sharpea、链球菌属(Streptococcus)、分枝杆菌属(Oribacterium)、毛螺旋菌属(Lachnospira);此外,还有变形菌门下的琥珀酸弧菌属(Succinivibrio)和放线菌门下的双歧杆菌属(Bifidobacterium)。
2.6 LEfSe分析LEfSe分析,即LDA Effect Size(linear discriminant analysis effect size)分析,是一种用于发现高维生物标识的分析工具,能够识别不同丰度的特征以及组与组之间具有统计学差异的生物标识。如图 7所示,在2组之间无显著差异的物种统一着色为黄色,红色节点表示在瘤胃酸中毒组中起到重要作用的物种类群,绿色节点表示在健康组中起到重要作用的物种类群,由内至外辐射的圆圈表示了由门至属的分类级别。在门水平上,纤维杆菌门(Fibrobacteres)是造成急性瘤胃酸中毒前后菌群群落显著性差异的重要因子,其下的纤维杆菌纲(Fibrobacteria)、纤维杆菌目(Fibrobacterales)、纤维杆菌科(Fibrobacteraceae)在健康组的各个分类水平下作用显著。而在瘤胃酸中毒组,芽孢杆菌纲(Bacilli)、乳酸杆菌目(Lactobacillales)、链球菌科(Streptococcaceae)分别在各自的分类水平下起到重要作用。另外,在科水平上,瘤胃酸中毒组的Paraprevotellaceae和疣微菌科(Ruminococcaceae)同样发挥了重要的作用。
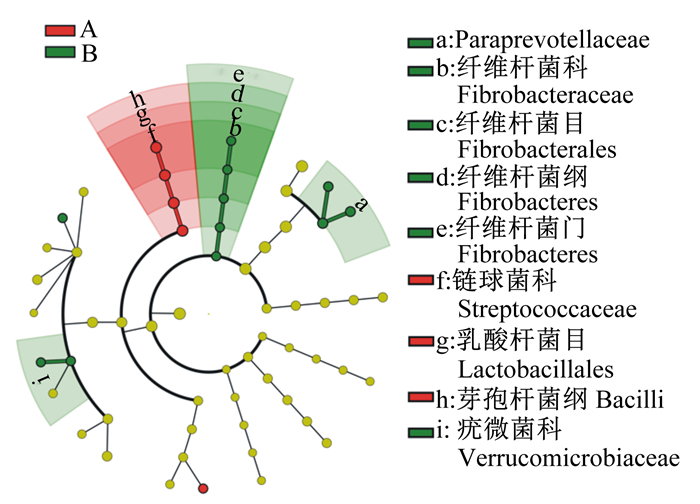
|
图 7 LEfSe分析结果 Fig. 7 LEfSe analysis results |
PICRUSt是由Langille等[12]于2013年开发,是最早被开发的基于16S rRNA基因序列来预测微生物群落功能的工具。如图 8所示,山羊发生急性瘤胃酸中毒前后菌群的缬氨酸、亮氨酸和异亮氨酸生物合成,糖酵解/糖异生,磷酸转移酶系统(PTS),戊糖和葡萄糖醛酸酯相互转化,氧化磷酸化,其他聚糖降解,氮代谢和蛋白质折叠及其相关加工8种功能存在显著差异。其中,前4种功能在发生急性瘤胃酸中毒后明显增强,后4种功能在发生急性瘤胃酸中毒后明显减弱。
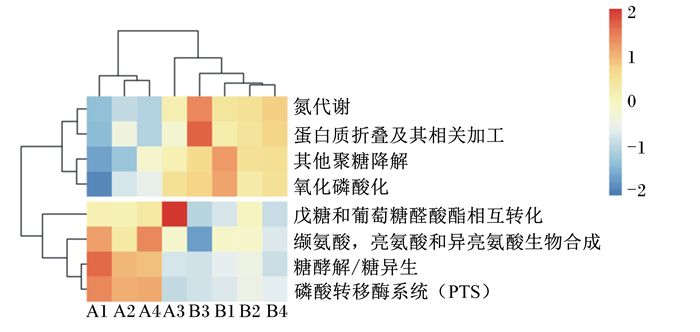
|
图 8 PICRUSt分析结果 Fig. 8 PICRUSt analysis results |
以往瘤胃酸中毒多发生于牛、羊等反刍动物过食谷物、碳水化合物的摄入在短时间内大量增加等情况。而随着我国养殖业的兴盛,在集约化、工厂化的生产模式下,过食谷物而导致的瘤胃酸中毒已较为少见。实际生产中,高精料饲喂是目前导致瘤胃酸中毒发生的主要原因。除高精料饲喂外,金磊等[13]报道,即使饲喂同一正常饲粮并且遗传背景相同的山羊,因为其能量代谢率可能存在显著差异,也会导致瘤胃菌群结构与组成发生显著差异,这是瘤胃酸中毒发生的一个潜在风险。因此,关于瘤胃酸中毒的相关研究符合生产需求,具有较高实际意义。
前人研究表明,高精料饲粮诱发奶牛亚急性瘤胃酸中毒后,瘤胃菌群的丰度指数(Chao1指数和Ace指数)和多样性指数(Shannon指数)显著降低[14]。本试验结果表明,在成功诱导急性瘤胃酸中毒之后,山羊瘤胃菌群多样性指数中的Shannon指数显著低于造模前,Simpson指数高于造模前,菌群丰度指数(Observed species指数、Ace指数、Chao1指数)低于造模前,瘤胃内菌群的数量有所下降但差异不显著,菌群多样性显著降低。以往我们一直认为在酸中毒状态下,瘤胃菌群的数量和多样性都会下降,但本试验结果表明,急性酸中毒状态下,细菌总数并无显著变化,而菌群多样性有显著下降。这表明急性酸中毒会使瘤胃菌群多样性明显下降,从而影响瘤胃菌群的组成与结构;也说明在急性瘤胃酸中毒时,菌群组成和结构的变化与细菌总数的变化可能并不同步。
通过UPGMA聚类方法对造模前后共8个样本进行聚类分析发现,造模前后的样本之间存在较大差异且组间差异性较高。然后,对8个样本距离矩阵进行PCoA,进一步证明了造模前的4个样本间菌群相似性较高,而造模后的4个样本间菌群相似性明显下降。由此可以判断出,急性瘤胃酸中毒前后的菌群组成存在较大差异,酸中毒后的菌群相似性下降,充分说明了酸中毒对瘤胃菌群组成的影响。
由以上分析可知,发生急性瘤胃酸中毒后瘤胃菌群的多样性有较大幅度下降,菌群组成的差异明显,且组内样本间相似性也有所下降。通过对不同分类水平上的物种注释与统计分析发现了导致这种差异的具体原因。已有研究报道,厚壁菌门、拟杆菌门和变形菌门是瘤胃细菌的三大优势菌群[15]。其中,拟杆菌门是瘤胃微生物中非纤维性碳水化合物的主要降解者,含有与降解非纤维类多糖有关的基因[16]。本试验结果同样证实厚壁菌门、拟杆菌门和变形菌门为瘤胃细菌的优势菌门。有研究表明,厚壁菌门下的链球菌属和解琥珀酸菌属是厚壁菌门的主要优势菌属[17]。厚壁菌门含有大量与纤维分解、碳水化合物代谢有关的菌属,如瘤胃球菌属、丁酸弧菌属、假丁酸弧菌属等[18]。本试验结果表明,厚壁菌门的相对丰度在发生急性瘤胃酸中毒后呈上升趋势,表明与瘤胃消化代谢淀粉类碳水化合物有关的细菌的数量有较大幅度的提高,这可能是瘤胃细菌维持在酸性环境中生存的必需。
在发生急性瘤胃酸中毒后,厚壁菌门下的乳酸杆菌目、Sharpea、链球菌属的相对丰度明显高于酸中毒前。已有研究报道,瘤胃中链球菌的发酵终产物为乳酸,其大量增殖可能是发生急性瘤胃酸中毒的主要原因[19]。Tajima等[20]发现,牛链球菌在动物适应高精料饲粮的前期可能发挥着重要作用。由此推断,瘤胃中的链球菌和乳酸杆菌通过利用葡萄糖(淀粉被淀粉酶、麦芽糖酶或麦芽糖磷酸化酶分解所产生)产生乳酸,导致血液pH升高。同时,这些能够以葡萄糖为底物的细菌大量增殖,成为优势菌群,改变瘤胃菌群的微生态平衡。它们相对丰度的变化可作为发生急性瘤胃酸中毒的一个潜在判读指标。
Sadet等[21]发现高精料的饲喂模式会影响密螺旋体菌属和毛螺旋体菌属对瘤胃上皮的黏附与定植。本试验结果同样证明了这一结论,在发生酸中毒后瘤胃密螺旋体属的相对丰度有显著升高,为之后进一步研究高精料饲粮对瘤胃上皮黏附菌群提供了依据。同时,结果发现变形菌门下的琥珀酸弧菌科、Mitochondria的相对丰度在发生急性瘤胃酸中毒后显著升高。有研究报道,变形菌门的大部分菌属可以和其他菌属竞争生存[22]。在本研究的试验设计中,由于造模导致瘤胃内环境由正常向酸性转变,蛋白质等易降解的营养物质基本被消耗殆尽,变形菌门相对丰度的增加可能与在营养浓度低的环境下不同菌群争夺资源、适度生长有关。
据报道,拟杆菌门细菌是瘤胃内厌氧菌群的主要代表之一,高精料饲喂可能会导致拟杆菌门细菌数量减少[19, 23]。隶属于拟杆菌门的普雷沃氏菌属是瘤胃菌属中占绝对地位的优势菌属[24]。普雷沃氏菌属不仅是活性高的蛋白质降解菌,而且还能利用淀粉以及果胶[25]。本试验结果表明,在急性瘤胃酸中毒前后瘤胃内普雷沃氏菌属的相对丰度并无显著变化,这与前人的研究结果并不一致。结合我们的试验设计推断,普雷沃氏菌属可能利用丰富的淀粉底物在短时间内进行了大量繁殖,从而出现保持相对丰度不变的结果。Li等[26]认为,普雷沃氏菌属的生长繁殖可能会受到其他活性高的纤维分解菌的抑制。由LEfSe分析可知,纤维杆菌在菌群变化中起到重要作用,该作用可能是指在急性瘤胃酸中毒发生后纤维杆菌的自溶或分解,纤维杆菌菌群的衰败可能有利于普雷沃氏菌属的生存。虽有报道称低pH可能会导致革兰氏阴性菌的死亡或自溶[19],但通过我们的试验发现,低pH对普雷沃氏菌属的这种作用可能受持续时间的影响,该持续时间有待进一步的研究。
通过PICRUSt软件进行测序后的基因功能预测,发现在发生急性瘤胃酸中毒后,瘤胃菌群内具有缬氨酸、亮氨酸和异亮氨酸生物合成,磷酸转移酶系统,戊糖和葡萄糖醛酸酯相互转化,糖酵解/糖异生4种功能的基因的相对丰度较高,这与瘤胃菌群生存所需的代谢功能,如碳水化合物运转与代谢、氨基酸合成与代谢等相一致[27-28]。这一结果说明瘤胃菌群在碳水化合物大量增加的情况下,会通过增强碳水化合物的转运与代谢等功能来适应生存环境的改变,从而有利于自身生存和繁殖。瘤胃菌群内具有蛋白质折叠及其相关加工、氧化磷酸化、其他聚糖降解和氮代谢4种功能的基因的相对丰度出现下降,这与瘤胃内酸性环境引发蛋白质降解、微生物蛋白质合成等功能受到抑制相一致[29-30]。功能预测结果显示,瘤胃菌群在pH较低的环境中氮源不足,蛋白质合成、加工与降解等功能明显下降。综合上述结果可知,瘤胃菌群受氮源不足的威胁,通过增强碳水化合物和氨基酸的利用,维持生存和繁殖。
4 结论山羊瘤胃菌群的结构和组成在发生急性瘤胃酸中毒前后存在较大差异,具体表现为:酸中毒后瘤胃菌群多样性下降,伴随组间相似性的下降;厚壁菌门下的链球菌属和乳酸杆菌属成为优势菌群,变形菌门菌群适度生长,拟杆菌门下的普雷沃氏菌属的相对丰度无显著变化;在蛋白质合成、加工与降解等功能受抑制的情况下,瘤胃菌群通过增加碳水化合物和氨基酸的利用,维持正常生存与繁殖。
| [1] |
张梦玲.荷斯坦奶牛胃肠道细菌菌群组成与功能的研究[D].硕士学位论文.南京: 南京农业大学, 2016.
|
| [2] |
NASROLLAHI S M, ZALI A, GHORBANI G R, et al. Short communication:blood metabolites, body reserves, and feed efficiency of high-producing dairy cows that varied in ruminal pH when fed a high-concentrate diet[J]. Journal of Dairy Science, 2019, 102(1): 672-677. DOI:10.3168/jds.2018-15022 |
| [3] |
GRILLI D J, FLIEGEROVÁK, KOPEČNYÝJ, et al. Analysis of the rumen bacterial diversity of goats during shift from forage to concentrate diet[J]. Anaerobe, 2016, 42: 17-26. DOI:10.1016/j.anaerobe.2016.07.002 |
| [4] |
ISHAQ S L, ALZAHAL O, WALKER N, et al. An investigation into rumen fungal and protozoal diversity in three rumen fractions, during high-fiber or grain-induced sub-acute ruminal acidosis conditions, with or without active dry yeast supplementation[J]. Frontiers in Microbiology, 2017, 8: 1943. DOI:10.3389/fmicb.2017.01943 |
| [5] |
闫佰鹏, 兰贵生, 李发弟, 等. 反刍动物亚急性瘤胃酸中毒变异性的机制解析[J]. 动物营养学报, 2018, 30(5): 1655-1661. DOI:10.3969/j.issn.1006-267x.2018.05.007 |
| [6] |
BOERMAN J P, POTTS S B, VANDEHAAR M J, et al. Milk production responses to a change in dietary starch concentration vary by production level in dairy cattle[J]. Journal of Dairy Science, 2015, 98(7): 4698-4706. DOI:10.3168/jds.2014-8999 |
| [7] |
HATEW B, PODESTA S C, VAN LAAR H, et al. Effects of dietary starch content and rate of fermentation on methane production in lactating dairy cows[J]. Journal of Dairy Science, 2015, 98(1): 486-499. DOI:10.3168/jds.2014-8427 |
| [8] |
FADROSH D W, MA B, GAJER P, et al. An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumina MiSeq platform[J]. Microbiome, 2014, 2(1): 6. DOI:10.1186/2049-2618-2-6 |
| [9] |
MAGOČ T, SALZBERG S L. FLASH:fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [10] |
EDGAR R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [11] |
HUO W J, ZHU W Y, MAO S Y. Impact of subacute ruminal acidosis on the diversity of liquid and solid-associated bacteria in the rumen of goats[J]. World Journal of Microbiology and Biotechnology, 2014, 30(2): 669-680. DOI:10.1007/s11274-013-1489-8 |
| [12] |
LANGILLE M G I, ZANEVELD J, CAPORASO J G, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [13] |
金磊, 周美丽, 王禄禄, 等. 不同能量代谢率的山羊瘤胃微生物结构与组成的差异性[J]. 微生物学通报, 2018, 45(1): 91-101. |
| [14] |
张瑞阳.组学技术研究亚急性瘤胃酸中毒对奶牛瘤胃微生物、代谢和上皮功能的影响[D].博士学位论文.南京: 南京农业大学, 2015.
|
| [15] |
BHATT V D, DANDE S S, PATIL N V, et al. Molecular analysis of the bacterial microbiome in the forestomach fluid from the dromedary camel (Camelus dromedarius)[J]. Molecular Biology Reports, 2013, 40(4): 3363-3371. DOI:10.1007/s11033-012-2411-4 |
| [16] |
RUSSELL J B, DIEZ-GONZALEZ F. The effects of fermentation acids on bacterial growth[J]. Advances in Microbial Physiology, 1998, 39: 205-234. |
| [17] |
KIM M, MORRISON M, YU Z T. STATUS of the phylogenetic diversity census of ruminal microbiomes[J]. FEMS Microbiology Ecology, 2011, 76(1): 49-63. |
| [18] |
EVANS N J, BROWN J M, MURRAY R D, et al. Characterization of novel bovine gastrointestinal tract Treponema isolates and comparison with bovine digital dermatitis treponemes[J]. Applied and Environmental Microbiology, 2011, 77(1): 138-147. |
| [19] |
NAGARAJA T G, TITGEMEYER E C. Ruminal acidosis in beef cattle:the current microbiological and nutritional outlook[J]. Journal of Dairy Science, 2007, 90. |
| [20] |
TAJIMA K, AMINOV R I, NAGAMINE T, et al. Diet-dependent shifts in the bacterial population of the rumen revealed with real-time PCR[J]. Applied and Environmental Microbiology, 2001, 67(6): 2766-2774. DOI:10.1128/AEM.67.6.2766-2774.2001 |
| [21] |
SADET S, MARTIN C, MEUNIER B, et al. PCR-DGGE analysis reveals a distinct diversity in the bacterial population attached to the rumen epithelium[J]. Animal, 2007, 1(7): 939-944. DOI:10.1017/S1751731107000304 |
| [22] |
BULYGINA E S, GALCHENKO V F, GOVORUKHINA N I, et al. Taxonomic studies on methylotrophic bacteria by 5S ribosomal RNA sequencing[J]. Journal of General Microbiology, 1990, 136(3): 441-446. DOI:10.1099/00221287-136-3-441 |
| [23] |
CHIQUETTE J, ALLISON M J, RASMUSSEN M A. Prevotella bryantii 25A used as a probiotic in early-lactation dairy cows:effect on ruminal fermentation characteristics, milk production, and milk composition[J]. Journal of Dairy Science, 2008, 91(9): 3536-3543. DOI:10.3168/jds.2007-0849 |
| [24] |
BEKELE A Z, KOIKE S, KOBAYASHI Y. Genetic diversity and diet specificity of ruminal Prevotella revealed by 16S rRNA gene-based analysis[J]. FEMS Microbiology Letters, 2010, 305(1): 49-57. DOI:10.1111/j.1574-6968.2010.01911.x |
| [25] |
李烨青, 奚雨萌, 曾涵芳, 等. 基于16S rRNA测序分析奶牛体外发酵中瘤胃细菌多样性的变化[J]. 动物营养学报, 2018, 30(10): 4059-4070. DOI:10.3969/j.issn.1006-267x.2018.10.031 |
| [26] |
LI Z P, LIU H L, LI G Y, et al. Molecular diversity of rumen bacterial communities from tannin-rich and fiber-rich forage fed domestic Sika deer (Cervus nippon) in China[J]. BMC Microbiology, 2013, 13: 151. DOI:10.1186/1471-2180-13-151 |
| [27] |
ERICKSON A R, CANTAREL B L, LAMENDELLA R, et al. Integrated metagenomics/metaproteomics reveals human host-microbiota signatures of Crohn's disease[J]. PLoS One, 2012, 7(11): e49138. DOI:10.1371/journal.pone.0049138 |
| [28] |
LAMENDELLA R, DOMINGO J W S, GHOSH S, et al. Comparative fecal metagenomics unveils unique functional capacity of the swine gut[J]. BMC Microbiology, 2011, 11: 103. DOI:10.1186/1471-2180-11-103 |
| [29] |
LENG R A, NOLAN J V. Nitrogen metabolism in the rumen[J]. Journal of Dairy Science, 1984, 67(5): 1072-1089. DOI:10.3168/jds.S0022-0302(84)81409-5 |
| [30] |
BACH A, CALSAMIGLIA S, STERN M D. Nitrogen metabolism in the rumen[J]. Journal of Dairy Science, 2005, 88: E9-E21. DOI:10.3168/jds.S0022-0302(05)73133-7 |




