木质纤维素作为最丰富的可再生生物质资源,年产量可达2 000亿t。农作物废弃原料如秸秆是巨大的生物质资源,但大多数秸秆被浪费或者露天焚烧,这样不仅浪费资源,还造成了环境的污染[1-2]。以木质纤维素为原料的清洁能源的开发有着可观的经济效益,且合理、有效地利用农作物废弃原料对环境污染、社会可持续发展具有重要意义。截至目前,生物方法是木质纤维素资源利用的主要方式,其前提是需获取高效降解木质纤维素的微生物并明确微生物之间的相互作用,但因木质纤维素在组成和结构上有着异常的复杂性,其降解需要多种微生物的协同互作,微生物的分类和代谢的多样性等尚不明确,微生物间的协同降解机制尚不清晰,从而限制了木质纤维素资源的开发和利用[3-4]。
反刍动物的瘤胃生态系统作为一种天然发酵体系,可对结构复杂的木质纤维素进行降解,从而使动物能够消化其饲料,而对木质纤维素的分解和代谢是由瘤胃中的微生物完成的。反刍动物的瘤胃内栖息着真菌、细菌、原虫和古细菌等多样而复杂的各种微生物,细菌和真菌是参与木质纤维素降解的主要瘤胃微生物[5-6]。瘤胃真菌不仅能够分泌比较全面的天然降解木质纤维素的酶系,而且能够降解晶体状和复杂结构的纤维素,在木质纤维素降解的研究中具有积极的意义[7]。但是由于厌氧真菌很难从瘤胃液中筛选和培养,因此对它们的生理生化特性及降解木质纤维素的作用机理了解不多。目前的研究主要集中在通过对筛选出的能降解木质纤维素的细菌菌株进行培养,研究其生理生化特性并对木质纤维素降解过程中可能存在的机理进行研究与推测。随着生物学技术的发展,特别是宏基因组学、宏转录组学和蛋白质组学等技术的发展,对降解木质纤维素的研究逐渐深入到基因水平,并在研究过程中取得了一定的成效[8]。本文主要围绕瘤胃内的木质纤维素降解菌、瘤胃微生物对木质纤维素的降解及微生物降解木质纤维素过程中的相互作用等方面进行综述。
1 瘤胃内木质纤维素降解菌的研究概况反刍动物瘤胃内存在着大量微生物,通过多种微生物的共同参与,在调节宿主生理功能、代谢反应和抵御病原体等方面发挥着重要作用[9]。瘤胃厌氧真菌在木质纤维素的降解中起着不可或缺的作用,它们不仅能分泌降解含有微晶纤维素的天然木质纤维素的酶系,而且能组装成具有高效催化活性的类似纤维小体复合物(cellulosome)[7]。但是瘤胃真菌由于很难分离纯化和传代培养,因此对它们的生理生化特性及降解木质纤维素的作用机制了解不多。而瘤胃细菌作为自然界中木质纤维素降解的主要作用者之一,因其生长快、易于大规模工业生产等特性,在降解木质纤维素中成为潜在应用前景的新候选者。
1.1 瘤胃细菌截至目前,已分离出并可培养的瘤胃细菌约有29个属69个种[10],其中白色瘤胃球菌(Ruminococcus albus)、巨大芽孢杆菌(Bacillus megaterium)、黄色瘤胃球菌(Ruminococcus flavefaciens)、苏云金芽孢杆菌(Bacillus thuringiensis)、溶纤维丁酸弧菌(Butyrivibrio fibrisolven)、小生纤维梭菌(Clostridium cellobioparus)、蜡状芽孢杆菌(Bacillus cereus)、产琥珀酸丝状杆菌(Fibrobacter succinogenes)和小瘤胃杆菌(Ruminobacer parvum)等细菌主要参与木质纤维素的降解,其中产琥珀酸丝状杆菌、白色瘤胃球菌和黄色瘤胃球菌为降解木质纤维素的优势菌。
白色瘤胃球菌是瘤胃降解木质纤维素的优势菌种之一,广泛存在于反刍动物瘤胃和胃肠道中,大部分分离菌株都能利用纤维素、木聚糖和纤维二糖为碳源,并能够产生β-葡萄糖苷酶、β-木糖苷酶和纤维素酶等多种木质纤维素降解酶[11]。大量研究表明白色瘤胃球菌酶含有家族37的碳水化合物结合模块(carbonhydrate binding module 37,CBM37,图 1-A),该模块能够识别多种多糖,包括纤维素、木聚糖、几丁质和地衣多糖[12-13]。研究表明,CBM37可能在白色瘤胃球菌分泌产生纤维素酶的黏附过程中作为桥梁将细菌和多糖底物衔接,并帮助酶固定在细菌膜表面[14]。但是有关CBM37在白色瘤胃球菌纤维素酶中的确切作用仍然是未知的,尽管它能识别并能与纤维素和许多其他多糖紧密结合,但其广泛的特异性和对该物种明显的独特性是令人质疑的,这也可能暗示着其具有更新颖的功能。因此,CBM37在白色瘤胃球菌纤维素酶中的确切作用还需进一步研究。
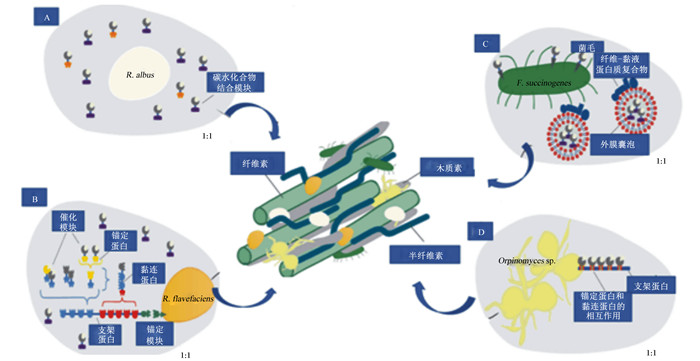
|
A:白色瘤胃球菌与含有CBM37的酶一起产生纤维小体复合物R. albus produces cellulosome complex along with CBM37-containing enzymes;B:黄色瘤胃球菌产生高度结构化的纤维小体复合物R. flavefaciens produces a highly structured cellulosome complex;C:产琥珀酸丝状杆菌产生外膜囊泡F. succinogenes produces outer membrane vesicules;D:厌氧根囊鞭菌属产生真菌纤维小体复合物Orpinomyces sp. produces fungal cellulosome complex。 图 1 瘤胃微生物降解木质纤维素示意图 Fig. 1 Schematic representations of cellulose-degrading machineries of rumen microorganisms |
大量研究表明黄色瘤胃球菌中存在类似纤维小体复合物(图 1-B),通过复杂的酶机制来降解纤维素[15]。研究表明黄色瘤胃球菌菌株都拥有一个支架蛋白编码的sca基因簇,进而合成纤维素酶复合体——类似纤维小体复合物[16]。黄色瘤胃球菌的类似纤维小体复合物因菌株而异[17],而黏连蛋白-锚定蛋白的相互作用主要在于菌株的特异性[18]。此外,黄色瘤胃球菌降解纤维素的能力也因菌株的不同而不同。研究发现黄色瘤胃球菌菌株FD-1中含有多种与类似纤维小体复合物相关的蛋白和酶,这说明该菌株可组成多种类似纤维小体复合物[18]。黄色瘤胃球菌菌株FD-1中同样含有类似的支架蛋白编码的sca基因簇,但它们的DNA序列差异很大,表明编码类似纤维小体复合物的支架蛋白编码的sca基因簇具有菌株特异性,这种非同寻常的特异性说明了黄色瘤胃球菌的高效纤维降解能力[19]。除了降解纤维素的糖苷水解酶(GH),黄色瘤胃球菌还编码类似数量的缺乏锚定蛋白的GH,这表明细菌结合了自由酶和纤维素酶来实现高水平的纤维降解。黄色瘤胃球菌的类似纤维小体复合物还需进一步体内分析和研究,以查明有关类似纤维小体复合物表达的更多酶亚基。
与能分解纤维素的瘤胃球菌不同的是,产琥珀酸丝状杆菌既不产生类似纤维小体复合物,也不分泌游离酶[20],它采用了另一种方法来降解纤维。在纤维素上生长时,它会产生外膜囊泡(图 1-C),用以降解纤维素和多糖类。在最近的研究中,这些外膜囊泡被分离出并通过蛋白质组学检测其含量发现大的外膜囊泡(平均49 nm)含有300多种不同的蛋白质,其中21%预测具有纤维降解功能。此外,外膜囊泡中的多蛋白质复合物似乎在纤维素结合和降解中发挥重要的作用[11]。这种细菌还会产生纤维黏液和菌毛蛋白,它们都在细菌的细胞壁上产生纤维素酶,并附着在纤维素上[21]。在大多数研究中,产琥珀酸丝状杆菌被认为是3种主要的纤维素降解菌中含量最丰富的[22-23]。早期对纤维降解的研究比较了纯分离的纤维素降解菌的酶学潜力,产琥珀酸丝状杆菌是瘤胃中最有效的木质纤维降解菌,它不仅可以高效降解结构异常复杂的结晶纤维素,而且在半纤维素的降解上也发挥着重要作用[24]。
1.2 瘤胃厌氧真菌由于厌氧真菌菌丝能够穿透植物细胞壁并溶解木质素成分,以及产生纤维素酶、半纤维素酶和酯酶等一系列高活性的降解植物细胞壁的纤维素降解酶,被认为在瘤胃中木质化组织的降解中发挥重要作用[25]。厌氧真菌还产生多种多糖降解酶,研究表明它们对发酵的贡献大于细菌[24]。瘤胃厌氧真菌对纤维素和半纤维素的降解均具有重要的作用,其中参与木质纤维素降解的主要包括6个属:新美鞭菌属(Neocallimastix)、盲肠鞭菌属(Caecomyces)、瘤胃壶菌属(Piromyces)、厌氧鞭菌属(Anaeromyces)、根囊鞭菌属(Orpinomyces)和枝梗鞭菌属(Cyllamyces)。其中,新美鞭菌属和瘤胃壶菌属降解木质纤维素效率较高[26]。对一些厌氧真菌物种的基因组分析表明,该基因库中富含半纤维素酶和纤维素酶,据此推测厌氧真菌通过细菌水平基因转移获得了GH,从而表现出高效纤维素降解活性[27]。
研究表明根囊鞭菌属、新美鞭菌属和瘤胃壶菌属等瘤胃厌氧真菌能够产生对高强度结晶状纤维素具有特异活性的类似纤维小体复合物(图 1-D)[27]。类似纤维小体复合物是由大量的糖基水解酶(β-葡萄糖苷酶、外切葡聚糖酶、内切葡聚糖酶和木聚糖酶)组装成的多酶复合体系,不同属菌株类似纤维小体复合物的组分和功能具有显著的差异[28]。与细菌类似纤维小体复合物相比,瘤胃厌氧真菌类似纤维小体复合物锚定蛋白域缺乏明确的序列同源性,支架蛋白和粘连蛋白也存在着显著的差异,这表明瘤胃真菌类似纤维小体复合物独立于细菌类似纤维小体复合物,但其结构和功能、合成与调控、分泌与组装以及作用机理有待进一步研究。
2 瘤胃微生物对木质纤维素的降解 2.1 纤维素的降解纤维素是植物细胞壁中最丰富的多糖,一般植物细胞壁中纤维素含量在20%~40%。它是由通过β-l, 4-糖苷键连接的D-吡喃葡萄糖单元形成的线性同聚多糖[29]。虽然纤维素是一种完全由葡萄糖单位组成的多糖,但它是一种很难被酶降解的底物,因为纤维素链与广泛的内部氢键和链间氢键紧密地结合在一起形成一种不溶的晶体化学结构。纤维素降解动力学较慢,降解程度不完全[18]。在纤维素降解期间,纤维素原纤维在链的末端受到外切葡聚糖酶的攻击,外切葡聚糖酶通常来自细菌中的GH48家族、有氧真菌中的GH6和GH7家族以及厌氧真菌中的GH6和GH48家族。这些特定的酶对于纤维素分解具有重要的作用。在降解纤维素的细菌中,GH48家族产生的酶在菌株纯培养生长时高度表达[15]。内切葡聚糖酶与外切葡聚糖酶具有协同作用,可在内部切割纤维素链。研究显示,主要的内切葡聚糖酶来自细菌的GH5、GH8和GH9家族以及真菌的GH6、GH7和GH45家族[30]。研究表明白色瘤胃球菌能够产生β-葡萄糖苷酶,可有效降解纤维二糖和纤维三糖,对纤维素的降解具有重要作用[31]。Lin等[32]研究显示溶纤维丁酸弧菌(Butyrivibrio fibrisolvens)H17c菌株能够产生GH3家族中的β-葡萄糖苷酶。对产琥珀酸丝状杆菌S85菌株的全基因组序列分析表明,该菌株具有104个GH、14个碳水化合物酯酶和4个多糖裂解酶[33],这种丰富的碳水化合物活性酶在高效降解纤维素方面发挥着至关重要的作用。由于厌氧真菌菌丝能够较好地穿透植物细胞壁并溶解木质素成分及产生一系列高活性的降解植物细胞壁的纤维素降解酶,因此,厌氧真菌在纤维素降解中发挥着重要的作用。研究发现,在瘤胃厌氧真菌中新美鞭菌属和瘤胃壶菌属降解木质纤维素的效率较高[27]。但是由于厌氧真菌很难分离和培养,因此在纤维素降解中研究的较少。
2.2 半纤维素的降解植物细胞壁的第2主要成分是由多种多糖组成的半纤维素,其中木聚糖含量最多。木聚糖是由木糖组成的链,这些链由不同的侧链糖组成。木聚糖酶通常来自GH10、GH11和GH30家族的内切木聚糖酶、GH43家族的外切木聚糖酶和侧链裂解酶[13]。尽管木聚糖的化学成分复杂,但微生物及其酶对木聚糖的降解能力较强,而且木聚糖降解的动力学也相对较快。半纤维素中也存在甘露聚糖、木葡聚糖和β-葡聚糖,甘露聚糖酶主要发现于GH5和GH26家族,木葡聚糖酶主要存在于CH5、GH12、GH44和GH74家族,而β-葡聚糖酶主要来自GH5、GH16和GH17家族[13]。
半纤维素是植物生物质中仅次于纤维素的最丰富的组成部分,在反刍动物营养和植物生物质工业转化为生物燃料和化学物质方面具有重要的底物作用[14]。由于半纤维素在组成和结构上的不均匀性,其降解受到的关注要少于纤维素。瘤胃中含有丰富的酶和细菌,这些酶和细菌在半纤维素的降解和利用方面具有多种功能,半纤维素的降解是由多种多样的细菌来完成的,这可能反映了细胞壁中半纤维素类型的多样性和复杂性。研究表明瘤胃中的普雷沃氏菌属(Prevotella)和丁酸弧菌属是高效的半纤维素降解菌[34]。Avguštin等[35]通过对普雷沃氏菌属和丁酸弧菌属进行分离和鉴定时发现它们具有非常广泛的酶库且具有不同的酶活性。普雷沃氏菌属和丁酸弧菌属除了具有很强的水解淀粉和半纤维素的能力外,还能水解蛋白质,并能同时发酵糖和氨基酸,它们的基因组包含多个来自不同家族的GH[36]。研究表明,产琥珀酸丝状杆菌和黄色瘤胃球菌同样会产生半纤维素酶,在半纤维素降解过程中有着不可或缺的作用[15]。虽然已知瘤胃中可溶性糖的一般降解途径,但对反刍动物的半纤维素降解尚不完全了解,半纤维素的利用和降解程度因瘤胃细菌属和种类而异,但这些变化的幅度以及菌株如何共同发挥作用仍不清楚,还有待进一步研究。
2.3 木质素的降解木质素是一种结构最复杂的碳水化合物,由各种生物稳定键组成。据估计,木质素占地球非化石有机碳的30%~35%。木质素降解在碳循环和生物燃料生产中起着重要作用[37]。木质素在传统营养学上被认为是不能被降解的物质,在瘤胃中有关降解木质纤维素的微生物和酶的研究很少。邢岩[38]以木质素为唯一碳源从牛瘤胃微生物中筛选出2株木质素降解菌,经鉴定分别为大肠埃希氏菌和胡萝卜软腐果胶杆菌,2株菌均产阿魏酸酯酶和乙酰酯酶。许浩[39]在水牛瘤胃中筛选出了木质素降解菌,经基因组测序发现与枯草芽孢杆菌具有较高的同源性,枯草芽孢杆菌带有漆酶(cotA)基因,如果该基因能够成功克隆并表达,将为瘤胃微生物降解木质素研究方面做出重要的贡献。
3 微生物在木质纤维素降解过程中的相互作用木质纤维素是由纤维素、半纤维素和木质素经多种化学键复合而成[40-41]。由于木质纤维素有异常复杂的结构,因此,单一的微生物或水解酶很难对其进行降解,需要瘤胃内的各种微生物及多种酶相互作用完成木质纤维素的降解,从而为宿主提供营养、能量。
3.1 原虫、细菌和真菌的相互作用反刍动物瘤胃中的细菌、真菌和原虫形成复杂的共生系统,共同参与木质纤维素的降解。由于细菌在数量上占主导地位,并具有多种代谢途径,因此瘤胃细菌对反刍动物摄入的木质纤维素的降解发挥着重要的作用[42]。由于瘤胃真菌可产生具有高活性的酶系列且优先附着于植物细胞壁上,对木质纤维素的降解比其他微生物更具优势[43],因此,在大颗粒和大片段植物纤维的降解过程中发挥重要作用。但是真菌在瘤胃中的数量较少且很难分离培养,因此分解纤维素的作用机制尚不清楚。
高巍[44]研究发现,将原虫、细菌和真菌混合培养时,其产气量和降解率显著高于原虫、细菌和真菌单独培养,尤其是细菌和原虫,这表明细菌和原虫在降解木质纤维素过程中存在协同效应。Lee等[45]研究同样发现,细菌和真菌共同培养可显著提高木质纤维素的降解率。因此,瘤胃微生物在降解木质纤维素的过程中存在非常复杂的相互作用。另有研究报道,只有产甲烷菌和寄生虫形成共生体,才能充分利用原生动物代谢的氢和甲酸来产生甲烷。在有产甲烷菌等利用氢的细菌存在的情况下,厌氧真菌能更有效地降解纤维素[46]。由于缺乏对瘤胃微生物之间相互作用的深入研究,所有这些理论都需要进一步验证。
3.2 微生物之间的竞争关系木质纤维素虽然是瘤胃生态系统中丰富的资源,但是也可能成为微生物竞争的共享资源,在一项关于厌氧真菌和瘤胃纤维素降解菌相互作用的研究中发现,细菌能够抑制真菌降解纤维素的能力,这种抑制作用是由细胞溶解细菌释放的细胞外蛋白引起的[47]。在纤维素底物上体外共同培养时发现,降解纤维素的产琥珀酸丝状杆菌、黄色瘤胃球菌和白色瘤胃球菌间的相互作用大多是抑制作用。虽然有研究显示体内这3种菌的附着率没有显著差异[48],但是Mosoni等[49]用放射标记法根据接种方式的不同(同时或顺序)观察纤维素降解菌对纤维素基质的黏附竞争时发现,这3种纤维素降解菌之间具有不同的相互抑制性作用。Emerson等[50]检测了这3种细菌共培养的具体生长情况,在纤维素底物过量时,白色瘤胃球菌与黄色瘤胃球菌、白色瘤胃球菌与产琥珀酸丝状杆菌或黄色瘤胃球菌与产琥珀酸丝状杆菌共同培养时,单个物种的细胞数大约相等。但是,当纤维素底物受到限制时,产琥珀酸丝状杆菌的竞争占有优势地位。这可能是该细菌产生了一种类似于细菌素的蛋白质,阻止了白色瘤胃球菌和黄色瘤胃球菌的生长,这种蛋白质的特异性作用取决于菌株。
4 木质纤维素降解菌组学的研究瘤胃微生物对木质纤维素的降解作用机理的揭示是一个全新的挑战,随着现代分子技术的发展,利用分子技术如宏基因组学、宏转录组学和蛋白质组学对瘤胃微生物群落结构、代谢途径及降解功能等进行解释,为瘤胃微生物在组学方面的应用提供了理论依据。
4.1 宏基因组学的研究宏基因组学是一种通过在环境样品中提全部微生物的DNA构建基因文库或直接测序,以研究微生物的多样性、进化关系和功能活性等目的的一种新颖的研究方法[9]。宏基因组学最初能够通过基因序列分析和功能的筛选选择出反刍动物瘤胃中降解木质纤维素的微生物种类及优势菌群。随着技术的发展,采用新一代测序技术研究降解木质纤维素菌群基因,能快速准确获得海量微生物基因数据以及更高的分类学信息,是研究微生物群落功能的重要途径[51-52]。
宏基因组学方法能够分析大量瘤胃中降解木质纤维素微生物的功能性基因和代谢通路机制。Hess等[53]通过对牛瘤胃微生物进行宏基因组学研究发现,瘤胃微生物群落能够产生一组非常丰富的酶,共有27 755个公认的碳水化合物活性基因,其中3 442个鉴定出为淀粉酶;随后,作者对奶牛瘤胃的宏基因组学研究表明,降解木质纤维素的GH家族的酶表达量更大,GH5、GH9、GH45、GH48家族中纤维素酶的表达量占总表达量的98%,其中GH48家族中纤维素酶的表达量最高。在半纤维素酶中,GH10、GH11和GH26家族最具代表性;作者还发现,大多纤维素酶和半纤维素酶来源于细菌,其中大部分纤维素酶序列由瘤胃球菌属和纤维杆菌属(Fibrobacter)表达,而半纤维素酶序列由瘤胃球菌属、普雷沃氏菌属和纤维杆菌属表达。Dai等[54]利用宏基因组测序与基于人工染色体功能筛选相结合的方法发现牦牛瘤胃中有150种降解木质纤维素的GH基因,其中大多数GH基因来自拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)。Ufarté等[55]对作用于多芳族化合物的细菌氧化还原酶通过基因组学鉴定发现这些酶来自于未培养的梭菌,属于复杂基因簇,涉及不同功能类型的蛋白质,包括半纤维素酶,可能通过协同作用对底物进行降解。宏基因组不仅可以直接获得微生物的功能性基因的丰富度和表达水平,还能通过基因数据与数据库的微生物物种进行比较,在未来工业化功能性酶领域的研究中具有重要意义。
4.2 宏转录组学的研究宏转录组学是在宏基因组学后兴起的一门学科,宏转录组学是以微生物全部RNA为研究对象分析特定时期群体生命体的全部基因组转录情况以及转录调控规律。宏转录组学通过研究微生物群落的转录情况及调控规律,分析确定其是否表达及表达量的多少,从而有效说明菌群的活力,是微生物群落代谢能力研究的重要方式。这种技术不但具有宏基因组学的全部优点,而且能够在转录表达水平上反映反刍动物瘤胃微生物的种类、多样性及潜在的基因功能。国内外已有采用宏转录组学技术研究木质纤维素降解的报道。Comtet-Marre等[56]通过宏转录组学高通量测序技术对泌乳奶牛瘤胃微生物群落中编码碳水化合物活性酶的表达基因功能进行分析鉴定,共鉴定出12 237个碳水化合物活性酶,主要由纤维二糖磷酸化酶(GH94家族)、淀粉酶(GH13家族)、半纤维素酶(GH43和GH10家族)、纤维素酶(GH9和GH48家族)、果胶酶(PL11家族)以及寡糖酶(GH2和GH3家族)组成,同时该研究还强调了真菌和纤毛原生动物对多糖降解的重要作用。田彦[57]采用宏转录组学技术对中国荷斯坦奶牛瘤胃微生物进行研究,确定了降解纤维素和半纤维素的微生物种类,并发现除了瘤胃球菌属、纤维杆菌属和普雷沃氏菌属外,瘤胃厌氧真菌中的新美鞭菌属、梨囊鞭菌属和根囊鞭菌属及纤毛原虫中的前毛虫属和多甲多泡毛虫属在纤维素和半纤维素降解过程中也发挥着重要作用。宏转录组学能将特定条件下的微生物群落及功能联系在一起,对群体能够进行各种相关功能的研究,但是微生物基因转录率的高低会让数据结果有偏差,需要与菌群DNA数据相结合,才能获得准确的微生物丰度变化和转录情况[58]。另外,RNA不仅含量极低,还容易降解和污染,在指定环境中提取总RNA时要格外小心[59-60]。
4.3 蛋白质组学的研究蛋白质组是一种基因组所表达的全套蛋白质。蛋白质组学则以蛋白质组为研究对象,研究内容包括蛋白质组成成分、蛋白质的表达水平、翻译后修饰和蛋白质之间的相互作用等[61]。与宏转录组学相比,蛋白质组学分析不仅能描述瘤胃微生物群落的基因表达蛋白的情况,而且在揭示降解木质纤维素酶的潜在功能和反刍动物对饲料的利用方面更占有优势。有关蛋白质组学技术在瘤胃微生物降解木质纤维素方面的研究国内外报道较少。Deusch等[62]对8 163个细菌蛋白质进行定量分析后发现了166个碳水化合物活性酶,并且GH的表达主要集中在GH57和GH2这2个家族。Snelling等[63]通过二维聚丙烯酰胺凝胶电泳(2D SDS-PAGE)技术测定饲喂高精料奶牛的瘤胃液中消化产物蛋白质,表明许多原核生物蛋白质组存在参与糖酵解的酶,如甘油醛-3-磷酸脱氢酶、磷酸烯醇丙酮酸羧激酶、磷酸甘油酸激酶和磷酸丙糖异构酶。在蛋白质水平的分析不但可以获取瘤胃微生物基因的表达情况,而且可深入了解微生物间的相互作用。对瘤胃微生物蛋白质组学的研究与分析,对揭示瘤胃微生物降解木质纤维素的策略具有重要意义[64]。
随着组学技术的发展,已有将宏基因组学、宏转录组学、蛋白质组学和宏代谢组学等技术联合应用于瘤胃微生物的研究。Deusch等[62]将宏基因组学、蛋白质组学和宏代谢组学相结合分析,揭示了普雷沃氏菌科是一个代谢功能广泛的群体,在浓缩和富含纤维的饲料消化中占据主导地位,并且为代谢组学提供了有关瘤胃微生物代谢活动的详细信息。Hassa等[65]利用多维组学对厌氧发酵中木质纤维素降解菌进行了研究,组装了数百个新菌的全基因组,揭示了微生物群落的遗传潜力。宏组学联合分析在瘤胃微生物中的应用还处于初步阶段,还有待进一步的研究。
5 小结数量巨大及种类繁多的瘤胃微生物形成了复杂的群落体系,它们通过相互之间的协同作用将反刍动物摄入的木质纤维素降解为宿主可利用的挥发性脂肪酸、糖类和蛋白质等物质。目前,尽管国内外有关瘤胃细菌和厌氧真菌降解木质纤维素及应用组学技术探索瘤胃微生物群落结构、代谢途径及降解功能等相关的研究已取得丰硕的成功,但是对有关瘤胃细菌和厌氧真菌产生的纤维素酶复合体——类似纤维小体复合物的结构功能、代谢与调控、产酶系统及作用机理等问题还需进一步的研究。相信随着技术的发展,尤其是宏组学联合分析技术的完善和发展,人们将揭示瘤胃微生物降解木质纤维素这一复杂的过程,为木质纤维素的开发和利用提供新思路。
| [1] |
ANWAR Z, GULFRAZ M, IRSHAD M. Agro-industrial lignocellulosic biomass a key to unlock the future bio-energy:a brief review[J]. Journal of Radiation Research and Applied Sciences, 2014, 7(2): 163-173. DOI:10.1016/j.jrras.2014.02.003 |
| [2] |
FERREIRA J A, MAHBOUBI A, LENNARTSSON P R, et al. Waste biorefineries using filamentous ascomycetes fungi:present status and future prospects[J]. Bioresource Technology, 2016, 215: 334-345. DOI:10.1016/j.biortech.2016.03.018 |
| [3] |
GROΒKOPF T, SOYER O S. Synthetic microbial communities[J]. Current Opinion in Microbiology, 2014, 18: 72-77. DOI:10.1016/j.mib.2014.02.002 |
| [4] |
许从峰, 艾士奇, 申贵男, 等. 木质纤维素的微生物降解[J]. 生物工程学报, 2019, 35(11): 2081-2091. |
| [5] |
丁立孝, 张青, 孙琴, 等. 瘤胃厌氧真菌对木质纤维素降解的研究进展[J]. 畜牧与饲料科学, 2010, 31(1): 16-19. DOI:10.3969/j.issn.1672-5190.2010.01.007 |
| [6] |
王超, 刘国道. 瘤胃微生物降解纤维素的研究进展[J]. 安徽农业科学, 2007, 35(13): 3771-3772, 3799. DOI:10.3969/j.issn.0517-6611.2007.13.002 |
| [7] |
陈富荣, 朱雅新, 东秀珠, 等. 瘤胃木质纤维素降解菌及降解酶基因的研究进展[J]. 微生物学报, 2010, 50(8): 981-987. |
| [8] |
WEIMER P J. Redundancy, resilience, and host specificity of the ruminal microbiota:implications for engineering improved ruminal fermentations[J]. Frontiers in Microbiology, 2015, 6: 296. |
| [9] |
盖叶顶, 王后福, 王淑玲, 等.瘤胃微生物宏组学分析及研究进展[J/OL].中国畜牧杂志, 2019, 1(10).(2019-10-10)[2019-10-31].https: //doi.org/10.19556/j.0258-7033.20190531-02.
|
| [10] |
黄青云. 畜牧微生物学[M]. 北京: 中国农业出版社, 2009.
|
| [11] |
ARNTZEN M Ø, VÁRNAI A, MACKIE R I, et al. Outer membrane vesicles from Fibrobacter succinogenes S85 contain an array of carbohydrate-active enzymes with versatile polysaccharide-grading capacity[J]. Environmental Microbiology, 2017, 19(7): 2701-2714. DOI:10.1111/1462-2920.13770 |
| [12] |
XU Q, MORRISON M, NELSON K E, et al. A novel family of carbohydrate-binding modules identified with Ruminococcus albus proteins[J]. The FEBS Letters, 2004, 566(1/2/3): 11-16. |
| [13] |
MORAÏS S, MIZRAHI I. Islands in the stream:from individual to communal fiber degradation in the rumen ecosystem[J]. FEMS Microbiology Reviews, 2019, 43(4): 362-379. DOI:10.1093/femsre/fuz007 |
| [14] |
ARTZI L, BAYER E A, MORAÏS S. Cellulosomes:bacterial nanomachines for dismantling plant polysaccharides[J]. Nature Reviews Microbiology, 2017, 15(2): 83-95. DOI:10.1038/nrmicro.2016.164 |
| [15] |
EZER A, MATALON E, JINDOU S, et al. Cell surface enzyme attachment is mediated by family 37 carbohydrate-binding modules, unique to Ruminococcus albus[J]. Journal of Bacteriology, 2008, 190(24): 8220-8222. DOI:10.1128/JB.00609-08 |
| [16] |
BENSOUSSAN L Z, MORAÏS S, DASSA B, et al. Broad phylogeny and functionality of cellulosomal components in the bovine rumen microbiome[J]. Environmental Microbiology, 2017, 19(1): 185-197. |
| [17] |
DASSA B, BOROVOK I, RUIMY-ISRAELI V, et al. Rumen cellulosomics:divergent fiber-degrading strategies revealed by comparative genome-wide analysis of six ruminococcal strains[J]. PLoS One, 2014, 9(7): e99221. DOI:10.1371/journal.pone.0099221 |
| [18] |
ISRAELI-RUIMY V, BULE P, JINDOU S, et al. Complexity of the Ruminococcus flavefaciens FD-1 cellulosome reflects an expansion of family-related protein-protein interactions[J]. Scientific Reports, 2017, 7: 42355. DOI:10.1038/srep42355 |
| [19] |
MILLER M E B, ANTONOPOULOS D A, RINCON M T, et al. Diversity and strain specificity of plant cell wall degrading enzymes revealed by the draft genome of Ruminococcus flavefaciens FD-1[J]. Plos One, 2009, 4(8): e6650. DOI:10.1371/journal.pone.0006650 |
| [20] |
MILLER M E B, ANTONOPOULOS D A, RINCON M T, et al. Diversity and strain specificity of plant cell wall degrading enzymes revealed by the draft genome of Ruminococcus flavefaciens FD-1[J]. PLoS One, 2009, 4(8): e6650. DOI:10.1371/journal.pone.0006650 |
| [21] |
SUEN G, WEIMER P J, STEVENSON D M, et al. The complete genome sequence of Fibrobacter succinogenes S85 reveals a cellulolytic and metabolic specialist[J]. PLoS One, 2011, 6(4): e18814. DOI:10.1371/journal.pone.0018814 |
| [22] |
BURNET M C, DOHNALKOVA A C, NEUMANN A P, et al. Evaluating models of cellulose degradation by Fibrobacter succinogenes S85[J]. PLoS One, 2015, 10(12): e0143809. DOI:10.1371/journal.pone.0143809 |
| [23] |
KOBAYASHI Y, SHINKAI T, KOIKE S. Ecological and physiological characterization shows that Fibrobacter succinogenes is important in rumen fiber digestion-review[J]. Folia Microbiologica, 2008, 53(3): 195-200. DOI:10.1007/s12223-008-0024-z |
| [24] |
DEHORITY B A. Rumen Microbiology[M]. Nottingham: Nottingham University Press, 2003.
|
| [25] |
AKIN D E, BORNEMAN W S. Role of rumen fungi in fiber degradation[J]. Journal of Dairy Science, 1990, 73(10): 3023-3032. DOI:10.3168/jds.S0022-0302(90)78989-8 |
| [26] |
FLIEGEROVA K, KAERGER K, KIRK P, et al.Rumen fungi[M]//PUNIYA A K, SINGH R, KAMRA D N.Rumen microbiology: from evolution to revolution.New Delhi: Springer, 2015: 97-112.
|
| [27] |
HAITJEMA C H, GILMORE S P, HENSKE J K, et al. A parts list for fungal cellulosomes revealed by comparative genomics[J]. Nature Microbiology, 2017, 2: 17087. DOI:10.1038/nmicrobiol.2017.87 |
| [28] |
雷威, 苏建明, 章怀云. 瘤胃厌氧真菌纤维素酶的研究进展[J]. 畜牧与饲料科学, 2005, 26(3): 12-14. DOI:10.3969/j.issn.1672-5190.2005.03.006 |
| [29] |
LYND K R, WEIME P J, VAN ZYL W H, et al. Microbial cellulose utilization:fundamentals and biotechnology[J]. Microbiology and Molecular Biology Reviews, 2002, 66(3): 506-577. |
| [30] |
LOMBARD V, RAMULU H G, DRULA E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2014, 42(D1): D490-D495. DOI:10.1093/nar/gkt1178 |
| [31] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria:potential for new insights from genomic analysis[J]. Nature Reviews Microbiology, 2008, 6(2): 121-131. DOI:10.1038/nrmicro1817 |
| [32] |
LIN L L, THOMSON J A. Cloning, sequencing and expression of a gene encoding a 73 kDa xylanase enzyme from the rumen anaerobe Butyrivibrio fibrisolvens H17c[J]. Molecular and General Genetics MGG, 1991, 228(1): 55-61. |
| [33] |
YOSHIDA S, MACKIE R I, CANN I K. Biochemical and domain analyses of FSUAxe6B, a modular acetyl xylan esterase, identify a unique carbohydrate binding module in Fibrobacter succinogenes S85[J]. Journal of Bacteriology, 2010, 192(2): 483-493. DOI:10.1128/JB.00935-09 |
| [34] |
JAMI E, MIZRAHI I. Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [35] |
AVGUŠTIN G, FLINT H J, WHITEHEAD T R. Distribution of xylanase genes and enzymes among strains of Prevotella (Bacteroides) ruminicola from the rumen[J]. FEMS Microbiology Letters, 1992, 78(7/8): 137-143. |
| [36] |
SESHADRI R, LEAHY S C, ATTWOOD G T, et al. Cultivation and sequencing of rumen microbiome members from the Hungate1000 collection[J]. Nature Biotechnology, 2018, 36(4): 359-367. |
| [37] |
AARTI C, ARASU M V, AGASTIAN P. Lignin degradation:a microbial approach[J]. South Indian Journal of Biological Sciences, 2015, 1(3): 119-127. DOI:10.22205/sijbs/2015/v1/i3/100405 |
| [38] |
邢岩.降解木质素厌氧菌株筛选及其转化秸秆作用研究[D].硕士学位论文.长春: 吉林大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10183-1012366847.htm
|
| [39] |
许浩.水牛瘤胃对不同木质素单体组成粗饲料中纤维降解的研究[D].硕士学位论文.武汉: 华中农业大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10504-1017194817.htm
|
| [40] |
朱宁.木质纤维素降解酶系在草本类生物质上的协作机制[D].博士学位论文.北京: 中国农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10019-1017000866.htm
|
| [41] |
MENON V, RAO M L. Trends in bioconversion of lignocellulose:biofuels, platform chemicals & biorefinery concept[J]. Progress in Energy and Combustion Science, 2012, 38(4): 522-550. DOI:10.1016/j.pecs.2012.02.002 |
| [42] |
WANG G R, DUAN Y L. Studies on lignocellulose degradation by rumen microorganism[J]. Advanced Materials Research, 2013, 853: 253-259. DOI:10.4028/www.scientific.net/AMR.853.253 |
| [43] |
HUH J H, KIM D J, ZHAO X Q, et al. Widespread activation of antibiotic biosynthesis by S-adenosylmethionine in Streptomycetes[J]. FEMS Microbiology Letters, 2004, 238(2): 439-447. DOI:10.1111/j.1574-6968.2004.tb09787.x |
| [44] |
高巍.瘤胃微生物对植物细胞壁降解的相对贡献及其附着与降解的关系研究[D].博士学位论文.北京: 中国农业大学, 2004. http://d.wanfangdata.com.cn/Thesis/Y658880
|
| [45] |
LEE S S, HA J K, CHENG K J. Relative contributions of bacteria, protozoa, and fungi to in vitro degradation of orchard grass cell walls and their interactions[J]. Applied and Environmental Microbiology, 2000, 66(9): 3807-3813. DOI:10.1128/AEM.66.9.3807-3813.2000 |
| [46] |
NEWBOLD C J, DE LA FUENTE G, BELANCHE A, et al. The role of ciliate protozoa in the rumen[J]. Frontiers in Microbiology, 2015, 6: 1313. |
| [47] |
BERNALIER A G, FONTY F, BONNEMOY F, et al. Inhibition of the cellulolytic activity of Neocallimastix frontalis by Ruminococcus flavefaciens[J]. Journal of General Microbiology, 1993, 139(4): 873-880. DOI:10.1099/00221287-139-4-873 |
| [48] |
ROGER V, FONTY G, KOMISARCZUK-BONY S, et al. Effects of physicochemical factors on the adhesion to cellulose avicel of the ruminal bacteria Ruminococcus flavefaciens and Fibrobacter succinogenes subsp. succinogenes[J]. Applied and Environmental Microbiology, 1990, 56(10): 3081-3087. DOI:10.1128/AEM.56.10.3081-3087.1990 |
| [49] |
MOSONI P, FONTY G, GOUET P. Competition between ruminal cellulolytic bacteria for adhesion to cellulose[J]. Current Microbiology, 1997, 35(1): 44-47. DOI:10.1007/s002849900209 |
| [50] |
EMERSON E L, WEIMER P J. Fermentation of model hemicelluloses by Prevotella strains and Butyrivibrio fibrisolvens in pure culture and in ruminal enrichment cultures[J]. Applied Microbiology Biotechnology, 2017, 101(10): 4269-4278. DOI:10.1007/s00253-017-8150-7 |
| [51] |
DEMANÈCHE S, DAVID M M, NAVARRO E, et al. Evaluation of functional gene enrichment in a soil metagenomic clone library[J]. Journal of Microbiological Methods, 2009, 76(1): 105-107. DOI:10.1016/j.mimet.2008.09.009 |
| [52] |
GILBERT J A, DUPONT C L. Microbial metagenomics:beyond the genome[J]. Annual Review of Marine Science, 2011, 3: 347-371. DOI:10.1146/annurev-marine-120709-142811 |
| [53] |
HESS M, SCZYRBA A, EGAN R, et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen[J]. Science, 2011, 331(6016): 463-467. DOI:10.1126/science.1200387 |
| [54] |
DAI X, ZHU Y X, LUO Y F, et al. Metagenomic insights into the fibrolytic microbiome in yak rumen[J]. PLoS One, 2012, 7(7): e40430. DOI:10.1371/journal.pone.0040430 |
| [55] |
UFARTÉ L, POTOCKI-VERONESE G, CECCHINI D, et al. Highly promiscuous oxidases discovered in the bovine rumen microbiome[J]. Frontiers in Microbiology, 2018, 9: 861. DOI:10.3389/fmicb.2018.00861 |
| [56] |
COMTET-MARRE S, CHAUCHEYRAS-DURAND F, BOUZID O, et al. FibroChip, a functional DNA microarray to monitor cellulolytic and hemicellulolytic activities of rumen microbiota[J]. Frontiers in Microbiology, 2018, 9: 215. DOI:10.3389/fmicb.2018.00215 |
| [57] |
田彦.基于高通量测序的中国荷斯坦奶牛瘤胃宏转录组研究[D].博士学位论文.北京: 中国科学院北京基因组研究所, 2015. http://cdmd.cnki.com.cn/Article/CDMD-80167-1015446359.htm
|
| [58] |
EMERSON J B, ADAMS R I, ROMÁN C M B, et al. Schrödinger's microbes:tools for distinguishing the living from the dead in microbial ecosystems[J]. Microbiome, 2017, 5: 86. DOI:10.1186/s40168-017-0285-3 |
| [59] |
SOREK R, COSSART P. Prokaryotic transcriptomics:a new view on regulation, physiology and pathogenicity[J]. Nature Reviews Genetics, 2010, 11(1): 9-16. DOI:10.1038/nrg2695 |
| [60] |
MARX I J, VAN WYK N, SMIT S, et al. Comparative secretome analysis of Trichoderma asperellum S4F8 and Trichoderma reesei Rut C30 during solid-state fermentation on sugarcane bagasse[J]. Biotechnology for Biofuels, 2013, 6: 172. DOI:10.1186/1754-6834-6-172 |
| [61] |
ADAV S S, CHEOW E S H, RAVINDRAN A, et al. Label free quantitative proteomic analysis of secretome by Thermobifida fusca on different lignocellulosic biomass[J]. Journal of Proteomics, 2012, 75(12): 3694-3706. DOI:10.1016/j.jprot.2012.04.031 |
| [62] |
DEUSCH S, CAMARINHA-SILVA A, CONRAD J, et al. A structural and functional elucidation of the rumen microbiome influenced by various diets and microenvironments[J]. Frontiers in Microbiology, 2017, 8: 1605. DOI:10.3389/fmicb.2017.01605 |
| [63] |
SNELLING T J, WALLACE R J. The rumen microbial metaproteome as revealed by SDS-PAGE[J]. BMC Microbiology, 2017, 17: 9. DOI:10.1186/s12866-016-0917-y |
| [64] |
谢骁, 孙会增, 刘建新. 宏组学在哺乳动物消化道微生物研究中的应用[J]. 现代牧业, 2017, 1(1): 1-8. DOI:10.3969/j.issn.1008-3111.2017.01.002 |
| [65] |
HASSA J, MAUS I, OFF S, et al. Metagenome, metatranscriptome, and metaproteome approaches unraveled compositions and functional relationships of microbial communities residing in biogas plants[J]. Applied Microbiology and Biotechnology, 2018, 102(12): 5045-5063. DOI:10.1007/s00253-018-8976-7 |




