2. 北京农学院奶牛营养学北京市重点实验室, 北京 102206
2. Beijing Key Laboratory for Dairy Cow Nutrition, Beijing University of Agriculture, Beijing 102206, China
反刍动物能将人类不能直接利用的纤维性植物原料转化成肉和奶等优质的畜产品,对人类社会发展具有重要意义。随着世界人口的增长以及居民生活水平的提高,人类社会对优质畜产品的需求越来越多,反刍动物生产的重要性也越来越大。然而,反刍动物在消化植物纤维的同时会向环境中排放甲烷等温室气体,反刍动物每年向环境中排放甲烷约1亿t,约占全球每年甲烷排放总量的20%[1]。甲烷的温室效应约为二氧化碳的25倍,反刍动物生产对环境的影响引起了越来越多的关注,各国科学家围绕反刍动物瘤胃甲烷排放展开了大量研究,通过适当的措施调控反刍动物瘤胃甲烷排放具有重要意义。
1 甲烷生成机制甲烷生成通常被认为是产甲烷古菌在严格厌氧的条件下独有的生命现象。但有研究报道,除产甲烷古菌外,蓝藻细菌和真核生物也具有产生甲烷的能力,甚至可以在有氧的情况下产生甲烷[2-4]。产甲烷古菌是一种生态多样性的微生物,广泛存在于多种陆生及水生厌氧环境中,包括湿地、海洋沉积物、淡水沉积物以及动物胃肠道等。甲烷是产甲烷菌厌氧呼吸的终产物[5],作为主要的甲烷产生源头,大气中70%的甲烷是由产甲烷菌产生的[6]。
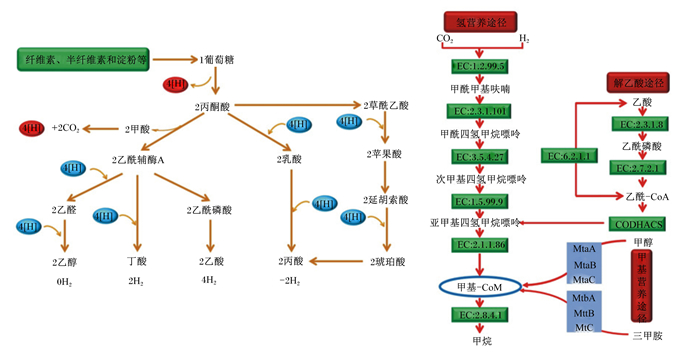
甲烷生成是生物质厌氧降解的终端过程,通常发生在氧、硝酸盐、Fe3+及硫酸盐等末端电子受体不足或快速耗尽的环境中[5-6]。甲烷生成的底物主要有二氧化碳、乙酸盐和甲基化合物。根据反应底物的不同,甲烷生成可分为3条途径:二氧化碳还原途径、解乙酸途径和甲基营养途径。3条途径的最后1步反应均为甲基辅酶M被甲基辅酶M还原酶还原生成甲烷。氢气是二氧化碳还原途径的主要电子供体,因此该途径也被称为氢营养途径,此外,甲酸、甲醇及一氧化碳也可作为该途径的电子供体[5]。氢营养途径是最常见的甲烷生成途径,甲烷杆菌目(Methanobacteriales)、甲烷球菌目(Methanococcales)、甲烷微菌目(Methanomicrobiales)、甲烷八叠球菌目(Methanosarcinales)、甲烷火菌目(Methanopyrales)及甲烷胞菌目(Methanocellales)的产甲烷菌均可通过该途径生成甲烷。在解乙酸途径中,乙酸裂解为羧基和甲基,羧基被氧化为二氧化碳,甲基被还原为甲烷。作为最不常见的途径,仅存在于Methanosarcinales,但由解乙酸途径生成的甲烷约占全球生物甲烷总量的2/3[5]。在甲基营养途径中,甲醇、甲胺及甲基硫化物等甲基化合物的甲基团传递给辅酶M,生成的甲基辅酶M最终被甲基辅酶M还原酶还原生成甲烷。对于常见的甲基营养型甲烷菌(主要来自Methanosarcinales),甲基还原所需的电子是通过额外的甲基被氧化成二氧化碳得到,但是Methanomicrococcus blatticola和Methanosphaera的甲基营养型甲烷菌以氢气作为电子供体[5-7]。最近研究发现,Methanomassiliicoccales的甲烷菌也以氢气作为电子供体,其甲烷生成途径属于氢气依赖型甲基营养途径[8]。甲基营养型甲烷菌主要存在于海洋沉积物中,以及动物胃肠道和一些极端环境中[5]。
2 反刍动物瘤胃甲烷生成反刍动物瘤胃微生物发酵碳水化合物产生甲烷,不但加剧全球温室效应,而且降低饲料利用率。反刍动物以甲烷形式所损失的能量占饲料总能的2%~12%[9]。研究人员围绕甲烷生成及其调控措施展开了大量研究,然而瘤胃微生物发酵碳水化合物生成挥发性脂肪酸的过程部分依赖于可排出代谢氢的甲烷生成过程,因此单纯抑制瘤胃甲烷生成的调控措施往往不能起到长期调控的目的[10-11]。产乙酸作用和丙酸生成等一些瘤胃内可与甲烷生成途径竞争代谢氢的内在代谢过程,在瘤胃甲烷调控方面的潜在作用越来越多的引起了公众的关注[12-13]。一方面,这些代谢过程通过与甲烷生成途径竞争代谢氢来抑制甲烷生成,不会因为瘤胃中的氢无法被及时排出而影响发酵;另一方面,乙酸和作为瘤胃葡萄糖前体物的丙酸均为反刍动物的能量来源物质,通过增加乙酸和丙酸的产量来竞争性抑制瘤胃甲烷生成可提高饲料能量的利用率。
碳水化合物是反刍动物主要的能量来源,纤维素、半纤维素和淀粉等多糖首先在瘤胃内水解为葡萄糖等单糖。各种单糖在瘤胃微生物的作用下进一步代谢为挥发性脂肪酸、二氧化碳及氢气。氢气是瘤胃发酵过程中重要的中间产物,在葡萄糖分解为丙酮酸以及丙酮酸氧化脱羧生成乙酰辅酶A的过程中产生(图 1)。为了保证瘤胃发酵的正常进行,产生的氢气需要被及时从瘤胃排出[10]。甲烷生成是瘤胃主要的排出氢气的途径,细菌、原虫以及真菌产生的氢气被传递给产甲烷古菌通过氢营养途径还原二氧化碳生成甲烷。瘤胃产甲烷古菌在数量和多样性上不如瘤胃细菌丰富,且在全世界范围内的反刍动物中高度保守[14]。Henderson等[14]对来自35个国家的瘤胃和前肠样品(379头牛、106头绵羊、59头鹿、52头山羊和72头其他物种)的微生物群落组成进行了全面的全球普查。研究发现,尽管样品的来源千差万别,但主要的古菌群体却惊人地相似。Methanobrevibacter gottschalkii和Methanobrevibacter ruminantium出现在所有样品中,且占比高达74%。Methanosphaera和Methanomassiliicoccaceae的2个古菌群体占比也比较高,这5个主要的产甲烷古菌群体约占整个古菌群落的90%。瘤胃中大约78%的产甲烷古菌通过氢营养途径产生甲烷,22%的产甲烷古菌通过甲基营养途径产生甲烷,解乙酸途径在瘤胃中较为少见[15]。甲烷短杆菌(Methanobrevibacter)是瘤胃中主要氢营养型甲烷菌[16],Methanosphaera、Methanimicrococcus和甲烷细菌属(Methanobacterium)也是瘤胃中重要氢营养型甲烷菌属[13]。瘤胃中的甲基营养型甲烷菌主要包括Methanosarcinales、Methanosphaera和Methanomassiliicoccaceae[5]。
除甲烷生成外,瘤胃中的丙酸生成过程以及由氢气和二氧化碳生成乙酸的过程都可消耗氢气。硝酸盐和硫酸盐等一些无机盐也可作为电子受体消耗氢气,但是这些物质在瘤胃中的含量通常不多[17-19],而且含量过多可能会增加反刍动物中毒的风险[20]。
3 反刍动物瘤胃甲烷生成相关微生物瘤胃甲烷的生成是瘤胃内各种微生物共同作用的结果,产甲烷古菌是直接的甲烷产生微生物,而原虫、细菌及真菌等其他微生物也在瘤胃甲烷生成过程中发挥着重要作用。
瘤胃原虫根据其结构和活性不同,主要有2种类型:Holotrich原虫,具有完全被纤毛覆盖的柔性表膜,主要消化可溶性底物;Entodiniomorphid原虫,具有坚硬的表膜,纤毛仅位于口部附近区域,能够消化微粒状物质[22]。尽管瘤胃原虫不能产生甲烷,但其可通过自身的氢化酶产生大量氢气供氢营养型产甲烷古菌使用,因此在瘤胃甲烷生成中发挥重要作用。此外,原虫的表面和体内附着以及寄生有产甲烷古菌,这种共生关系也使原虫成为瘤胃甲烷生成重要的参与者[22]。Methanobrevibacter和Methanomicrobium被认为是最主要的2个与原虫具有共生关系的产甲烷古菌属[23-24]。尽管原虫在瘤胃中普遍存在,但原虫对于瘤胃并不重要,且驱除原虫可以降低9%~37%的甲烷排放[25-26]。然而,也有一些研究表明,驱除原虫对瘤胃甲烷产生的影响并不明显[27-28]。Newbold等[29]通过Meta分析研究发现,通过驱除原虫平均可降低11%的甲烷产量,然而产甲烷古菌的丰度并没有显著降低。
同原虫类似,真菌通过产生大量氢气参与瘤胃甲烷生成,此外真菌发酵也可产生二氧化碳、甲酸和乙酸等代谢终产物[30]。目前已知的瘤胃中的真菌属有6个,Neocallimastix、Caecomyces、Piromyces、Anaeromyces、Orpinomyces和Cyllamyces[31]。一些产甲烷古菌可能也与真菌有共生关系,然而这种关系并没有得到证实[32]。由于真菌能产生氢气,通常认为真菌的丰度可能与甲烷生成有关,但Kittelmann等[33]研究发现,绵羊瘤胃甲烷排放与真菌群落结构没有相关性。Newbold等[29]通过Meta分析研究发现,驱除原虫虽然对产甲烷古菌的丰度没有影响,但降低了真菌的丰度,而真菌丰度的降低是否与甲烷产量的降低有关尚不清楚。
细菌是瘤胃内最多样化的微生物类群,能分解纤维、淀粉、蛋白质和糖等多种物质,瘤胃中最丰富的3个细菌门是厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)[14]。属于Firmicutes的纤维分解菌瘤胃球菌属(Ruminococcus)和优杆菌属(Eubacterium)能够产生氢气,纤维杆菌属(Fibrobacter)不产生氢气,而Bacteroidetes是纯粹的氢气消耗菌[34]。Kittelmann等[33]通过分析236份来自于118头不同甲烷排放量绵羊的瘤胃液样品,发现瘤胃微生物的群落结构与绵羊甲烷排放水平相关。一种类型的低甲烷排放量绵羊瘤胃中具有较高含量的丙酸生成菌Quinella ovalis;另一种类型的低甲烷排放量绵羊瘤胃中具有较高含量的乳酸和琥珀酸生成菌,包括Fibrobacter spp.、Kandleria vitulina、Olsenella spp.、Prevotella bryantii和Sharpea azabuensis;高甲烷排放量绵羊的瘤胃中具有较高含量属于瘤胃球菌属(Ruminococcus)的一些菌种,以及瘤胃球菌科(Ruminococcaceae)、毛螺旋菌科(Lachnospiraceae)、Catabacteriaceae、粪球菌属(Coprococcus)和普雷沃菌属(Prevotella)等。Danielsson等[35]和Wallace等[36]均研究发现,高甲烷排放动物个体的瘤胃中Proteobacteria的含量较少。琥珀酸弧菌科(Succinivibrionaceae)是Proteobacteria的优势科,有研究发现肠道中较高含量的Succinivibrionaceae是导致Tammar袋鼠相比反刍动物甲烷生成效率低的原因[37]。Prevotella是一种多功能的菌属,该属的部分菌种在高甲烷排放个体中含量较高,而部分菌种又在低甲烷排放个体中含量较高[16, 35]。通常情况下,高甲烷排放的动物个体肠道中有更多的氢气产生菌,而低甲烷排放的动物个体肠道中有更多的氢气消耗菌。
4 反刍动物瘤胃甲烷测定方法准确测定反刍动物甲烷排放量对于研究甲烷生成机制及其减排措施具有重要意义。呼吸舱法以其高的准确性及可重复性被认为是甲烷测定的“金标”方法,但由于其成本高、技术要求严格且对动物应激较大,因此限制了该方法的广泛使用[38]。六氟化硫示踪法相比于呼吸舱法,可直接在生产条件下对动物的甲烷排放进行测定,但该方法的准确性受当地天气变化影响较大且存在残留问题,因此该方法的广泛使用亦受到限制[39]。此外,直接测定法还包括头箱法、面罩法、便携式收集舱法、甲烷/二氧化碳比例法、GreenFeed体系法、嗅探器法、甲烷激光探测器等短期测定方法,以及适用于大群体测定的塑料大棚法和微气象法[40]。直接测定法虽然设备成本高、技术要求苛刻且操作难度大,但直接测定法是研究甲烷排放的基础方法,是其他方法参考和对比的标准。
间接测定甲烷产量的方法主要有体外法和统计模型法。体外法通过模拟体内瘤胃环境来研究气体生成,故而其影响因素较多[41]。统计模型法通常根据营养物质或能量摄入量建立线性或非线性模型估测甲烷排放量,实用性强。国家水平以及全球水平甲烷排放量的测定均采用统计模型法估测[40]。随着技术水平的不断改进,模型法预测的准确性越来越高,并且发展了针对不同种类反刍动物的专用模型(表 1)。此外,间接测定法还包括根据乳中特定脂肪酸的浓度预测甲烷产量的方法以及乳中红外光谱法[42]。统计模型法是一种非常有效的甲烷估测方法,但代表性强的统计模型需要以直接测定法为基础。
|
|
表 1 甲烷产量预测模型 Table 1 Models used to predict CH4 production |
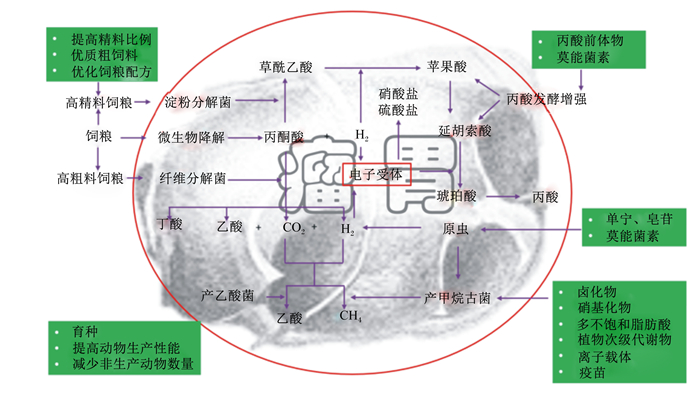
瘤胃甲烷产量受动物个体、饲粮组成、瘤胃发酵模式、瘤胃微生物组成及活性等多种因素影响。研究人员围绕反刍动物瘤胃甲烷调控展开了大量研究,主要的调控措施包括:调整饲粮结构,改变瘤胃发酵模式;使用甲烷抑制剂,抑制甲烷生成途径及甲烷生成相关微生物活性;增加其他电子受体,竞争性抑制甲烷产生。此外,提高动物的生长性能,优化畜群结构减少非生产动物的数量,以及选育低甲烷排放品种等也是调控瘤胃甲烷排放的有效措施(图 2)。通常情况下,不同调控措施之间可相互影响,共同发挥调控作用。
|
图 2 反刍动物瘤胃甲烷排放调控措施 Fig. 2 Strategies to mitigate CH4 emission in rumen[40] |
调整饲粮结构,改善瘤胃发酵模式和瘤胃微生物组成及活性进而调控瘤胃甲烷排放。饲粮组成对瘤胃甲烷生成具有重要影响。随饲粮精料比例的增加,瘤胃中乙酸比例降低,丙酸比例升高,瘤胃发酵模式以丙酸型发酵为主,而丙酸是瘤胃中仅次于甲烷的氢利用物质,可竞争性抑制瘤胃甲烷产生[10]。高精料饲粮会降低瘤胃pH,抑制产甲烷古菌及原虫活性进而降低甲烷产量[47]。然而,增加饲粮精料比例不但增加了饲养成本,而且易引起亚急性或急性瘤胃酸中毒、蹄叶炎等营养代谢病。Meale等[48]通过体外法研究发现,墨西哥丁香(Gliricidia sepium)和臂形草(Brachiaria ruziziensis)具有降低甲烷排放的潜力。Machado等[49]通过体外法研究了多种海藻类植物发现,钥形毛藻(Asparagopsis)和鞘藻(Oedogonium)具有显著降低甲烷排放的作用。Wang等[12]研究发现,增加饲粮中非粗料来源纤维的含量,有降低体外甲烷产量的趋势,并增加了发酵液中丙酸的比例。因此,开发利用新型优质饲料比单纯增加饲粮精料水平更有应用前景。
使用甲烷抑制剂,抑制甲烷生成途径及甲烷生成相关微生物活性。甲烷抑制剂主要包括植物次级代谢物、脂类、卤代物、离子载体及硝基物等。单宁等植物次级代谢物一方面可抑制产甲烷古菌活性抑制甲烷生成,另一方面可减少原虫数量抑制甲烷生成[50]。脂类可通过抑制产甲烷古菌活性抑制甲烷生成,且不饱和脂肪酸还可通过生物氢化作用竞争性抑制甲烷生成[51]。卤代物和离子载体对甲烷的抑制作用主要是通过毒害产甲烷古菌直接降低甲烷产量[52-53]。离子载体还可刺激产琥珀酸菌和丙酸菌生长,通过增加丙酸产量从而竞争性抑制甲烷生成[40]。3-硝基丙醇可在不损害动物生产性能和健康的情况下持续降低甲烷产量并增加丙酸产量[54],被认为是目前最有潜力及应用价值的甲烷抑制剂。此外,科学家通过免疫法使用产甲烷古菌疫苗抑制瘤胃甲烷产生[55],但由于大部分瘤胃产甲烷菌无法纯培养,因而利用纯培养产甲烷古菌开发的疫苗可能导致瘤胃未培养产甲烷菌数量增加。甲烷抑制剂普遍存在成本高、毒副作用及生物残留等诸多弊端,且抑制剂可使产甲烷古菌产生抗性,因此甲烷抑制剂长期作用效果有待观察。
增加其他电子受体,竞争性抑制甲烷生成。硝酸盐在瘤胃中对氢的亲和力大于二氧化碳,可通过与产甲烷古菌竞争氢进而减少瘤胃甲烷生成[49],硝酸盐自身被还原为亚硝酸盐,进一步生成氨。Lee等[56]研究发现,饲粮中添加硝酸盐能降低肉牛12%的甲烷排放。Van Zijderveld等[57]在奶牛饲粮中添加硝酸盐,降低了16%的甲烷排放。硝酸盐和亚硝酸盐具有一定的毒性,饲喂硝酸盐会增加其在组织和奶中的残留[58-59]。此外,饲粮中添加延胡索酸和苹果酸等丙酸前体物,可通过增加丙酸生成竞争性抑制甲烷生成[60]。
6 小结消耗氢气生成甲烷从而促进碳水化合物的降解吸收是反刍动物在物种进化过程中形成的正常生理机制,各种直接抑制甲烷生成的调控措施在瘤胃微生物复杂的自我调节下很难长期发挥作用。丙酸生成是瘤胃内可与甲烷生成途径竞争代谢氢的内在代谢过程,通过竞争性抑制甲烷产生从而增加丙酸产量,不会因为瘤胃中的氢无法被及时排出而影响发酵,同时作为瘤胃葡萄糖前体物的丙酸是反刍动物重要的能量来源物质。因此,竞争性抑制甲烷生成,在调控甲烷生成的同时提高饲料能量利用率是一种具有应用前景的甲烷调控模式。
| [1] |
CONRAD R. The global methane cycle:recent advances in understanding the microbial processes involved[J]. Environmental Microbiology Reports, 2009, 1(5): 285-292. DOI:10.1111/j.1758-2229.2009.00038.x |
| [2] |
LENHART K, BUNGE M, RATERING S, et al. Evidence for methane production by saprotrophic fungi[J]. Nature Communications, 2012, 3: 1046. DOI:10.1038/ncomms2049 |
| [3] |
LIU J G, CHEN H, ZHU Q A, et al. A novel pathway of direct methane production and emission by eukaryotes including plants, animals and fungi:an overview[J]. Atmospheric Environment, 2015, 115: 26-35. DOI:10.1016/j.atmosenv.2015.05.019 |
| [4] |
BIŽIĆ M, KLINTZSCH T, IONESCU D, et al. Cyanobacteria, the most ancient and abundant photoautotrophs on earth produce the greenhouse gas methane during photosynthesis[J]. BioRxiv, 2019, 398958. |
| [5] |
LIU Y C, WHITMAN W B. Metabolic, phylogenetic, and ecological diversity of the methanogenic archaea[J]. Annals of the New York Academy of Sciences,, 2008, 1125(1): 171-189. DOI:10.1196/annals.1419.019 |
| [6] |
LYU Z, SHAO N N, AKINYEMI T, et al. Methanogenesis[J]. Current Biology, 2018, 28(13): R727-R732. DOI:10.1016/j.cub.2018.05.021 |
| [7] |
THAUER R K, KASTER A K, SEEDORF H, et al. Methanogenic archaea:ecologically relevant differences in energy conservation[J]. Nature Reviews Microbiology, 2008, 6(8): 579-591. DOI:10.1038/nrmicro1931 |
| [8] |
SÖLLINGER A, URICH T. Methylotrophic methanogens everywhere-physiology and ecology of novel players in global methane cycling[J]. Biochemical Society Transaction, 2019, 47(6): 1895-1907. DOI:10.1042/BST20180565 |
| [9] |
JOHNSON K A, JOHNSON D E. Methane emissions from cattle[J]. Journal of Animal Science, 1995, 73(8): 2483-2492. DOI:10.2527/1995.7382483x |
| [10] |
MCALLISTER T A, NEWBOLD C J. Redirecting rumen fermentation to reduce methanogenesis[J]. Australian Journal of Experimental Agriculture, 2008, 48(2): 7-13. DOI:10.1071/EA07218 |
| [11] |
DENMAN S E, FERNANDEZ G M, SHINKAI T, et al. Metagenomic analysis of the rumen microbial community following inhibition of methane formation by a halogenated methane analog[J]. Frontiers in Microbiology, 2015, 6: 1087. |
| [12] |
WANG K, NAN X M, CHU K K, et al. Shifts of hydrogen metabolism from methanogenesis to propionate production in response to replacement of forage fiber with non-forage fiber sources in diets in vitro[J]. Frontiers in Microbiology, 2018, 9: 2764. DOI:10.3389/fmicb.2018.02764 |
| [13] |
LAN W, YANG C L. Ruminal methane production:associated microorganisms and the potential of applying hydrogen-utilizing bacteria for mitigation[J]. Science of the Total Environment, 2019, 654: 1270-1283. DOI:10.1016/j.scitotenv.2018.11.180 |
| [14] |
HENDERSON G, COX F, GANESH S, et al. Rumen microbial community composition varies with diet and host, but a core microbiome is found across a wide geographical range[J]. Scientific Reports, 2015, 5: 14567. DOI:10.1038/srep14567 |
| [15] |
SESHADRI R, LEAHY S C, ATTWOOD G T, et al. Cultivation and sequencing of rumen microbiome members from the Hungate1000 collection[J]. Nature Biotechnology, 2018, 36(4): 359-367. DOI:10.1038/nbt.4110 |
| [16] |
KITTELMANN S, SEEDORF H, WALTERS W A, et al. Simultaneous amplicon sequencing to explore co-occurrence patterns of bacterial, archaeal and eukaryotic microorganisms in rumen microbial communities[J]. PLoS One, 2013, 8(2): e47879. DOI:10.1371/journal.pone.0047879 |
| [17] |
NEWBOLD C J, LÓPEZ S, NELSON N, et al. Propionate precursors and other metabolic intermediates as possible alternative electron acceptors to methanogenesis in ruminal fermentation in vitro[J]. British Journal of Nutrition, 2005, 94(1): 27-35. DOI:10.1079/BJN20051445 |
| [18] |
VAN ZIJDERVELD S M, GERRITS W J J, APAJALAHTI J A, et al. Nitrate and sulfate:effective alternative hydrogen sinks for mitigation of ruminal methane production in sheep[J]. Journal of Dairy Science, 2010, 93(12): 5856-5866. DOI:10.3168/jds.2010-3281 |
| [19] |
BEAUCHEMIN K A, UNGERFELD E M, ECKARD R J, et al. Review:fifty years of research on rumen methanogenesis:lessons learned and future challenges for mitigation[J]. Animal, 2020, 14(Suppl.1): S2-S16. |
| [20] |
PATRA A K. Prediction of enteric methane emission from buffaloes using statistical models[J]. Agriculture, Ecosystems & Environment, 2014, 195: 139-148. |
| [21] |
SHI W B, MOON C D, LEAHY S C, et al. Methane yield phenotypes linked to differential gene expression in the sheep rumen microbiome[J]. Genome Research, 2014, 24(9): 1517-1525. DOI:10.1101/gr.168245.113 |
| [22] |
BELANCHE A, DE LA FUENTE G, NEWBOLD C J. Study of methanogen communities associated with different rumen protozoal populations[J]. FEMS Microbiology Ecology, 2014, 90(3): 663-677. DOI:10.1111/1574-6941.12423 |
| [23] |
JANSSEN P H, KIRS M. Structure of the archaeal community of the rumen[J]. Applied and Environmental Microbiology, 2008, 74(12): 3619-3625. DOI:10.1128/AEM.02812-07 |
| [24] |
PATRA A, PARK T, KIM M, et al. Rumen methanogens and mitigation of methane emission by anti-methanogenic compounds and substances[J]. Journal of Animal Science and Biotechnology, 2017, 8: 13. DOI:10.1186/s40104-017-0145-9 |
| [25] |
HOOK S E, WRIGHT A D G, MCBRIDE B W. Methanogens:methane producers of the rumen and mitigation strategies[J]. Archaea, 2010, 2010: 945785. |
| [26] |
MORGAVI D P, FORANO E, MARTIN C, et al. Microbial ecosystem and methanogenesis in ruminants[J]. Animal, 2010, 4(7): 1024-1036. DOI:10.1017/S1751731110000546 |
| [27] |
HEGARTY R S, BIRD S H, VANSELOW B A, et al. Effects of the absence of protozoa from birth or from weaning on the growth and methane production of lambs[J]. British Journal of Nutrition, 2008, 100(6): 1220-1227. DOI:10.1017/S0007114508981435 |
| [28] |
BIRD S H, HEGARTY R S, WOODGATE R. Persistence of defaunation effects on digestion and methane production in ewes[J]. Australian Journal of Experimental Agriculture, 2008, 48(2): 152-155. DOI:10.1071/EA07298 |
| [29] |
NEWBOLD C J, DE LA FUENTE G, BELANCHE A, et al. The role of ciliate protozoa in the rumen[J]. Frontiers in Microbiology, 2015, 6: 1313. |
| [30] |
GRUNINGER R J, PUNIYA A K, CALLAGHAN T M, et al. Anaerobic fungi (phylum Neocallimastigomycota):advances in understanding their taxonomy, life cycle, ecology, role and biotechnological potential[J]. FEMS Microbiology Ecology, 2014, 90(1): 1-17. |
| [31] |
ISHAQ S L, KIM C J, REIS D, et al. Fibrolytic bacteria isolated from the rumen of north american moose (Alces alces) and their use as a probiotic in neonatal lambs[J]. PLoS One, 2015, 10(12): e0144804. DOI:10.1371/journal.pone.0144804 |
| [32] |
WEI Y Q, LONG R J, YANG H, et al. Fiber degradation potential of natural co-cultures of Neocallimastix frontalis and Methanobrevibacter ruminantium isolated from yaks (Bos grunniens) grazing on the Qinghai Tibetan Plateau[J]. Anaerobe, 2016, 39: 158-164. DOI:10.1016/j.anaerobe.2016.03.005 |
| [33] |
KITTELMANN S, PINARES-PATINO C S, SEEDORF H, et al. Two different bacterial community types are linked with the low-methane emission trait in sheep[J]. PLoS One, 2014, 9(7): e103171. DOI:10.1371/journal.pone.0103171 |
| [34] |
TAPIO I, SNELLING T J, STROZZI F, et al. The ruminal microbiome associated with methane emissions from ruminant livestock[J]. Journal of Animal Science and Biotechnology, 2017, 8: 7. DOI:10.1186/s40104-017-0141-0 |
| [35] |
DANIELSSON R, DICKSVED J, SUN L, et al. Methane production in dairy cows correlates with rumen methanogenic and bacterial community structure[J]. Frontiers in Microbiology, 2017, 8: 226. |
| [36] |
WALLACE R J, ROOKE J A, MCKAIN N, et al. The rumen microbial metagenome associated with high methane production in cattle[J]. BMC Genomics, 2015, 16: 839. DOI:10.1186/s12864-015-2032-0 |
| [37] |
POPE P B, SMITH W, DENMAN S E, et al. Isolation of Succinivibrionaceae implicated in low methane emissions from tammar wallabies[J]. Science, 2011, 333(6042): 646-648. DOI:10.1126/science.1205760 |
| [38] |
GRAINGER C, CLARKE T, MCGINN S M, et al. Methane emissions from dairy cows measured using the sulfur hexafluoride (SF6) tracer and chamber techniques[J]. Journal of Dairy Science, 2007, 90(6): 2755-2766. DOI:10.3168/jds.2006-697 |
| [39] |
WILLIAMS S R O, MOATE P J, HANNAH M C, et al. Background matters with the SF6 tracer method for estimating enteric methane emissions from dairy cows:a critical evaluation of the SF6 procedure[J]. Animal Feed Science and Technology, 2011, 170(3/4): 265-276. |
| [40] |
PATRA A K. Recent Advances in measurement and dietary mitigation of enteric methane emissions in ruminants[J]. Frontiers in Veterinary Science, 2016, 3: 39. |
| [41] |
RYMER C, HUNTINGTON J A, WILLIAMS B A, et al. In vitro cumulative gas production techniques:history, methodological considerations and challenges[J]. Animal Feed Science and Technology, 2005, 123-124: 9-30. DOI:10.1016/j.anifeedsci.2005.04.055 |
| [42] |
NEGUSSIE E, DE HAAS Y, DEHARENG F, et al. Invited review:large-scale indirect measurements for enteric methane emissions in dairy cattle:a review of proxies and their potential for use in management and breeding decisions[J]. Journal of Dairy Science, 2017, 100(4): 2433-2453. DOI:10.3168/jds.2016-12030 |
| [43] |
PATRA A K, LALHRIATPUII M, DEBNATH B C. Predicting enteric methane emission in sheep using linear and non-linear statistical models from dietary variables[J]. Animal Production Science, 2016, 56(2/3): 574-584. |
| [44] |
PATRA A K, LALHRIATPUII M. Development of statistical models for prediction of enteric methane emission from goats using nutrient composition and intake variables[J]. Agriculture Ecosystems & Environment, 2016, 215: 89-99. |
| [45] |
MORAES L E, STRATHE A B, FADEL J G, et al. Prediction of enteric methane emissions from cattle[J]. Global Change Biology, 2014, 20(7): 2140-2148. DOI:10.1111/gcb.12471 |
| [46] |
PATRA A K. Prediction of enteric methane emission from cattle using linear and non-linear statistical models in tropical production systems[J]. Mitigation and Adaptation Strategies for Global Change, 2016, 1-22. |
| [47] |
KUMAR S, DAGAR S S, PUNIYA A K, et al. Changes in methane emission, rumen fermentation in response to diet and microbial interactions[J]. Research in Veterinary Science, 2013, 94(2): 263-268. DOI:10.1016/j.rvsc.2012.09.007 |
| [48] |
MEALE S J, CHAVES A V, BAAH J, et al. Methane production of different forages in in vitro ruminal fermentation[J]. Asian-Australasian Journal of Animal Sciences, 2011, 25(1): 86-91. DOI:10.5713/ajas.2011.11249 |
| [49] |
MACHADO L, MAGNUSSON M, PAUL N A, et al. Effects of marine and freshwater macroalgae on in vitro total gas and methane production[J]. PLoS One, 2014, 9(1): e85289. DOI:10.1371/journal.pone.0085289 |
| [50] |
PATRA A K, SAXENA J. A new perspective on the use of plant secondary metabolites to inhibit methanogenesis in the rumen[J]. Phytochemistry, 2010, 71(11/12): 1198-1222. |
| [51] |
WILLIAMS S R O, MOATE P J, DEIGHTON M H, et al. Methane emissions of dairy cows cannot be predicted by the concentrations of C8 : 0 and total C18 fatty acids in milk[J]. Animal Production Science, 2014, 54(10): 1757-1761. DOI:10.1071/AN14292 |
| [52] |
CHEN M, WOLIN M J. Effect of monensin and lasalocid-sodium on the growth of methanogenic and rumen saccharolytic bacteria[J]. Applied and Environmental Microbiology, 1979, 38(1): 72-77. |
| [53] |
MARTINEZ-FERNANDEZ G, DENMAN S E, YANG C L, et al. Methane inhibition alters the microbial community, hydrogen flow, and fermentation response in the rumen of cattle[J]. Frontiers in Microbiology, 2016, 7: 1122. |
| [54] |
HRISTOV A N, OH J, GIALLONGO F, et al. An inhibitor persistently decreased enteric methane emission from dairy cows with no negative effect on milk production[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(34): 10663-10668. DOI:10.1073/pnas.1504124112 |
| [55] |
WRIGHT A D G, KENNEDY P, O'NEILL C J, et al. Reducing methane emissions in sheep by immunization against rumen methanogens[J]. Vaccine, 2004, 22(29/30): 3976-3985. |
| [56] |
LEE C, ARAUJO R C, KOENIG K M, et al. Effects of encapsulated nitrate on growth performance, nitrate toxicity, and enteric methane emissions in beef steers:backgrounding phase[J]. Journal of Animal Science, 2017, 95(8): 3700-3711. |
| [57] |
Van ZIJDERVELD S M, GERRITS W J J, DIJKSTRA J, et al. Persistency of methane mitigation by dietary nitrate supplementation in dairy cows[J]. Journal of Dairy Science, 2011, 94(8): 4028-4038. DOI:10.3168/jds.2011-4236 |
| [58] |
GUYADER J, DOREAU M, MORGAVI D P, et al. Long-term effect of linseed plus nitrate fed to dairy cows on enteric methane emission and nitrate and nitrite residuals in milk[J]. Animal, 2016, 10(7): 1173-1181. DOI:10.1017/S1751731115002852 |
| [59] |
DOREAU M, ARBRE M, POPOVA M, et al. Linseed plus nitrate in the diet for fattening bulls:effects on methane emission, animal health and residues in offal[J]. Animal, 2017, 12(3): 501-507. |
| [60] |
LI X Z, LONG R J, YAN C G, et al. Rumen microbial responses in fermentation characteristics and production of CLA and methane to linoleic acid in associated with malate or fumarate[J]. Animal Feed Science and Technology, 2010, 155(2/3/4): 132-139. |