2. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Beijing Institute of Animal Husbandry and Veterinary Sciences, Chinese Academy of Agricultural Sciences, Beijing 100193, China
动物机体各个部位存在各种共生细菌,条件致病菌、致病微生物与宿主之间形成相互依存,相互作用,维持稳定且营养丰富的生态系统动态平衡。有些微生物共生体(微生物组)的共有基因组为宿主动物提供了多种优势,例如不同结构的碳水化合物的消化吸收与代谢、维生素的生物合成以及对先天性和适应性免疫系统的调节作用等。
奶牛乳腺内环境中,以传统宿主-病原体相互作用的角度看待乳腺与微生物之间的关系,即当病原微生物的感染或环境性病原菌侵入时,导致乳腺防御能力下降,引起乳腺组织炎性反应及乳成分的改变,造成严重危害。据报道,引起奶牛乳房炎的病原菌有150多种,主要包含细菌、病毒、真菌和支原体等。其中细菌为主要的致病菌,例如金黄色葡萄球菌、链球菌、大肠杆菌占所有病原菌的83.74%[1-2]。然而,近年来随着宏基因组学的快速发展与广泛应用,越来越多的研究表明,临床上健康的乳腺分泌物也可以包含各种细菌群基因组的标记物,且其中绝大多数与乳腺炎无关[3]。因此,“乳腺共生微生物”的概念运用而生,其生态特性对于更好地了解乳腺炎的发病机理可能具有重要意义,为治疗和预防乳腺炎提供了新的视角与机遇。
目前研究结果表明,最佳的乳腺菌群多样性与免疫稳态有关,而乳腺炎或患有乳腺炎病史的微生物菌群多样性显著减少[4-6]。那么,乳房微生物群多样性的破坏(生物代谢)是否具有决定乳腺炎易感性的作用尚不清楚。而且,关于各种生物和非生物因子对乳房内微生物菌落多样性形成的作用研究较少。因此,本文旨在综述目前关于乳房各个生态部位中微生物菌群的研究进展,并强调了微生物群落代谢网络特点及高度动态的微生物生态系统研究方向,并对乳腺抗菌方面的应用潜力及开发进行了展望。
1 乳头部菌群奶牛乳头和乳池在泌乳活动中发挥了重要作用,具有排出和储存乳汁的作用。乳头管作为预防病原菌侵袭的第1道防线,内衬有角质蛋白,不但封闭管道,还可有效抑制细菌和病原菌的生长;此外,乳头管底部,靠近乳头乳池处,含有特殊的生理结构——弗斯登堡静脉环(图 1),可有效抵御外来病原菌的侵袭以及防止乳汁的溢出[7]。其次,乳头作为病原菌入侵的重要窗口,其生理结构是防止乳房炎的有效结构。研究发现,基于传统培养方法和DNA提取等方法,奶牛乳头细菌群落具有较高的微生物多样性[8],作为共生菌群,主要包括病原体及与皮肤相关的条件致病菌;在门水平上的细菌主要包括放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形杆菌门(Proteobacteria),可以留在奶牛乳头的皮肤上;在属水平上的细菌主要包括不动杆菌属(Acinetobacter)、气球菌属(Aerococcus)、棒状杆菌属(Corynebacterium)、肠杆菌属(Enterobacter)、法克菌属(Facklamia)、乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、微球菌属(Micrococcus)、丙酸杆菌属(Propionibacterium)、葡萄球菌属(Staphylococcus)和链球菌属(Streptococcus)等。曾学琴[9]等通过对乳房炎奶牛乳头和乳汁中微生物群落多样性分析,结果表明,在门水平上乳头丰度最高的是细菌厚壁菌门,而乳汁中丰度最高的细菌是变形菌门;且患有乳房炎奶牛的乳头样品均含有拟杆菌门细菌,而乳汁中几乎没有,但梭杆菌门细菌仅存在于乳头中。因此,奶牛乳头的菌落结构与乳房炎的发生发展密切相关。
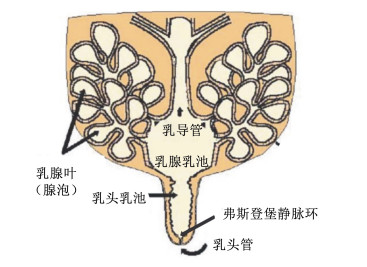
|
图 1 奶牛乳腺乳区解剖示意图 Fig. 1 Anatomy of mamary gland of dairy cows[10] |
长期以来,乳头端过度角化病一直被认为是发展临床型乳房炎的潜在危险因素之一[11]。过度角化是指乳头末端的角质层(即角蛋白衬里)的厚度增加,通常是由于长期刺激(例如,由于挤奶习惯造成的创伤)而引起的。多项研究表明,乳头过度角化病与较高的微生物负荷和隐性乳房炎的发生呈正相关[12-13]。Derakhshani等[14]评估奶牛的生理状态和乳头过度角化对乳头管和乳汁中微生物群特征的影响,结果表明,乳头过度角化病与乳房任一部位的微生物群总体多样性之间没有很强的相关性,然而鞘氨醇杆菌属(Sphingobacterium)除外,乳头过度角化病与Sphingobacterium丰度呈显著正相关关系,而Sphingobacterium是进一步与临床型乳房炎的潜在发病率相关的细菌属。因此,乳头的过度角化严重危害了乳头管的完整性,并通过机会型或者致病型细菌群抑制乳腺内细菌的定植能力。
此外,环境性病原菌也是影响乳腺健康的主要外源性致病菌,经乳头感染而引起奶牛的乳房炎。研究发现,大肠杆菌、变形杆菌、肺炎克雷伯菌、产气肠杆菌等为主要的环境性病原体[15]。据报道,不动杆菌、气球菌和棒状杆菌是奶牛乳头顶端皮肤上最常见的菌属[16],而环境中的不动杆菌和气球菌是人类皮肤的正常定居者[17],但是它们是否与奶牛乳房健康和隐性乳房炎相关尚未见报道。
2 乳头管部菌群结构奶牛的乳头管长0.65~1.30 cm,一端与乳头乳池相连,另一端开口于乳头顶端的乳头孔;乳头管的大小和括约肌的紧缩度与奶牛泌乳速度密切相关[18]。乳头管由于其复杂的生化和结构特性,可以有效阻止环境微生物进入乳腺。但是,如果乳头管的角蛋白塞部分丢失或挤奶后乳头管的括约肌扩张,则会损害乳房的第1道防御屏障,并通过多种来自乳房外部环境中的微生物,增加奶牛对乳腺炎的易感性[19]。致病菌破坏了乳头管并进入乳房的乳头池,大多数乳腺炎病原体能够以相对较快的速度在乳房内生态系统中生存和增殖,并引发不同程度的炎症反应[20]。
目前,由于在牛场挤奶厅条件下,对乳头管进行无菌采样比较困难,只有少数研究报道了该部位的微生物群落多样性[21]。研究发现,葡萄球菌属是乳头管菌群中最常见的定居者,即使在挤奶前后对乳头消毒后也是如此[22]。此外,一些与肠道相关的细菌,如梭菌科(Clostridiaceae)和鞭毛藻科(Lachnospiraceae),也被认为是乳头管中微生物群的主要成员。Falentin等[21]研究发现,健康母牛的乳头管和乳池中的Corynebacterium、瘤胃球菌属(Ruminococcus)、Aerococcus、双歧杆菌属(Bifidobacterium)和Facklamia是常见的微生物群。有研究报道,将患有临床型乳腺炎病史的奶牛乳头管的菌群结构与健康奶牛相比,临床型乳腺炎病史的奶牛乳头管含有更高比例的细菌[14]。梭状芽胞杆菌属(Clostridia)和Bifidobacterium在健康乳区的比例较高,因此,杆菌与梭状芽胞杆菌的比例在判定乳房健康中起重要作用。据报道,从牛奶或乳头管中分离出的乳酸菌可以黏附并内化奶牛乳腺上皮细胞,并调节细胞促炎性细胞因子的产生[23],有助于充分认识乳腺免疫防御机制,对预防与治疗奶牛乳腺炎具有重要的意义。
因此,乳头和乳头管等部位定居的微生物群落组成和功能直接影响着乳房内微生物的结构组成,与乳房的健康密不可分。大量研究结果表明,某些金黄色葡萄球菌物种在乳头管的微生物定植可为研发治疗乳房炎的药物提供重要的依据[24-26]。此外,通过对乳汁、乳头皮肤、粪便和床垫材料中的微生物群落比较分析结果证实,乳头皮肤是牛奶微生物群中最重要的来源[27];而畜群粪便和床垫铺料的微生物群是乳内微生物群第二大流行来源[28]。这些发现强调了乳房的各个部位,特别是乳头皮肤和乳头管的微生物群,以及各种环境源(如垫料和挤奶设备等)在塑造乳腺内微生物群中的重要作用。
3 乳腺内微生物的潜在起源与环境因素乳腺对微生物定植的防御机制受到多种因素的调节(图 2),包括:奶牛的基因型(特别是编码先天性和适应性免疫系统各个组成部分的基因)、母牛生理状况的变化、不同泌乳周期的代谢状况和饲养管理[29],例如干奶期抗微生物的治疗、挤奶后乳头消毒和挤奶卫生等[27]。上述各个因素在调节乳房的免疫稳态和抵抗乳腺感染的过程中都起着重要的作用。因此,这些因素在对乳腺内微生物群落的组成及功能等方面也具有直接或间接影响。
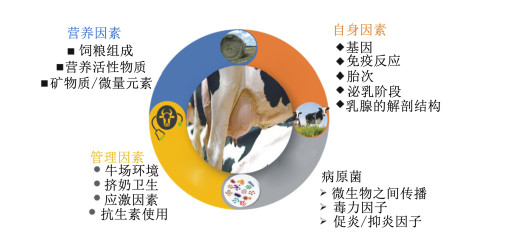
|
图 2 奶牛乳腺健康影响因素概图 Fig. 2 Determinants of udder healthy of dairy cows |
目前,鉴定乳腺炎易感性遗传标志物的主要方法是候选基因内的遗传变异,最重要的是对乳腺炎病原体所发生的全身和局部免疫反应[30],即与抗原识别后,白细胞募集和病原体消除是最重要的评判标准。例如,由于编码主要组织相容性复合物(major histocompatibility complex,MHC)分子的基因[也称为牛白细胞抗原(BoLA)]在调节先天性和获得性免疫应答中的关键作用而受到越来越多的关注[31-32]。特别是MHC基因和共生菌群之间的潜在联系,也得到广泛研究。研究表明,MHC基因型的多态性可以影响胃肠道上共生菌群的组成,例如某些Ⅱ类MHC基因型可以增加小鼠对沙门氏菌诱导的肠道感染的抵抗力[33],但在奶牛乳腺炎中关于MHC基因与共生菌群之间的联系研究较少。此外,在最近的一项研究中,BoLA DRB3.2的多态性与分娩后立即收集的初乳样品中独特的微生物群谱密切相关[34],在这项研究中,每种已鉴定的BoLA-DRB 3.2单倍型(n=3)与初乳菌群内特定操作分类单元的富集相关,如S. chromogenes、S. sciuri和S. succinus物质。因此,与乳腺炎易感性密切相关的候选基因可能通过调节乳内微生物群落,参与调节乳房稳态以及保护乳房免受主要乳腺炎病原体的侵袭而发挥作用。
奶牛的营养管理对乳房稳态及乳腺健康具有重要的意义。例如在围产期,奶牛的营养供应和干物质采食量(DMI)落后于机体所需的能量,负能量平衡(NEB)的这种不可避免的状态会导致各种新陈代谢疾病的发展,从而导致免疫系统受损[35]。机体内非酯化脂肪酸(NEFA)和酮体浓度升高与关键免疫功能受损密切相关,这些免疫功能包括吞噬活动、巨噬细胞和中性粒细胞的活性氧(ROS)产生、中性粒细胞的募集及其易位内皮、炎性细胞因子的分泌、肝细胞产生补体因子和急性期蛋白、激活免疫系统以及分泌免疫球蛋白等。但是,目前关于围产期代谢紊乱对乳房微生物群组成的潜在影响知之甚少。
此外,亚急性瘤胃酸中毒(SARA)是一种因高精料饲喂而引起的瘤胃和后肠微生物群失调所发生的代谢紊乱疾病[36]。研究发现,在发生SARA的初产奶牛泌乳初期的乳汁微生物群与病原菌和条件性致病菌[包括嗜麦芽单胞菌属(Stenotrophomonas)、Streptococcus、假单胞菌属(Pseudomonas)和产碱杆菌属(Alcaligenes)]的富集显著相关[37],证实全身和局部炎症反应的诱导是SARA调节乳房免疫稳态的潜在机制。还有研究报道,在SARA期间,反刍动物胃肠道中游离脂多糖(LPS,革兰氏阴性细菌外细胞膜的免疫原性成分)的脱落和积累会触发系统性炎症反应[38]。那么,SARA对胃肠道上皮屏障的破坏可使得游离的LPS从肠腔转移到血液循环和淋巴系统,与特定的急性期蛋白结合称为LPS结合蛋白[39-40];所得的LPS结合蛋白是一种免疫调节复合物,能够从血液循环/淋巴系统转移到包括乳房在内的各种身体部位,并通过激活核因子-κB(NF-κB)促炎途径来调节局部免疫力。但是,与SARA相关的乳房微生物群组成的变化是炎症性免疫反应激活的结果,还是由于细菌从胃肠道转移到乳房引起的,仍未得到答案,需要进一步研究。
奶牛的居住条件在塑造乳房微生物群的组成中起着核心作用,而乳头和乳头管内微生物群落的组成与牛场环境密切相关[28]。据报道,虽然挤奶前乳头消毒不影响室内奶牛乳汁的微生物群,但在室外放牧季节,经过挤奶前乳头消毒的奶牛乳汁微生物群比未处理组更为多样化[41]。在挤奶厅收集的乳汁样本中,牛奶菌群中与肠道相关的菌属如Ruminococcus、Aerococcus和普雷沃菌属(Prevotella)的比例较高;而在牧场收集的乳汁样本中,革兰氏阴性菌[如假单胞菌(Pseudomonas)]和Acinetobacter的比例较高。这些发现为更好地了解环境因素(尤其是牛场管理和挤奶卫生)在塑造整体乳房微生物群中的作用提供了宝贵的见解。床垫管理是乳腺炎控制程序的另一个重要方面,它可能会影响奶牛环境的微生物状况,进而影响乳房的微生物定植;据报道,在使用稻草垫料的农场中,冬季采集的牛奶和乳头采集的棉签样本均比夏季采集的样本富含物种更丰富的微生物群[42]。这项发现强调了季节性因素(如环境温度和湿度)对奶牛周围环境微生物群组成以及乳房微生物群的潜在贡献。
4 微生物菌群失调与乳房健康的潜在影响越来越多的证据表明,临床型乳房炎的发生与乳腺内微生物多样性的减少和菌群组成的改变密切相关。研究发现,与健康奶牛相比,梭杆菌门仅在乳房炎奶牛乳头中检测到,而在健康奶牛乳头中未检测到;乳房炎奶牛乳头Staphylococcus丰度高于正常奶牛,肠球菌属(Enterococcus)仅在乳房炎奶牛牛奶中检测到,正常奶牛牛奶中未检测到[9]。而且,乳腺炎病原体通常有多种毒力因子,使它们能够抵抗机体介导的免疫反应而在乳房内定植[43]。在最初的定植之后,病原体可以迅速地在乳房的生态系统中生长并消除其常驻共生菌群。然而,在乳房生态系统中,致病菌的优势通常不会持续存在,而且由于免疫细胞的活动和通过定期挤奶进行的清除,或者在治疗干预的帮助下(如乳腺内注射抗菌剂),这种优势通常会自行消除。因此,共生细菌的恢复程度一方面取决于用于治疗乳腺炎的抗菌剂的活性,另一方面也取决于侵入性病原体的毒力因子,如生物膜的形成和内化乳腺上皮细胞的能力等[44]。因此,充分了解乳腺微生物群落不仅能加深我们对乳房各个部位之间的生态关系的理解,而且能使我们更全面、更准确地评估各种乳腺炎治疗的有效性。
5 小结综上所述,乳腺内的共生菌群栖息于乳房的不同部位,包括乳头顶端、乳导管和乳房内的生态系统,可以通过直接的微生物-微生物交互作用、间接的免疫刺激或两者兼用来调节奶牛对乳腺炎病原体的易感性。因此,更好地了解能够影响乳房内环境稳定的各种因素,为开发治疗奶牛乳房炎的新思路及新方法奠定基础。
虽然目前对乳房的微生物生态系统的了解尚处于婴儿期,但是乳房内微生物群的最佳多样性及由1个健康的乳房组成共生菌群和机会菌群之间的平衡,对于维持促炎反应和抗炎反应之间的平衡从而维持乳房内环境稳定是至关重要的。研究奶牛的生理状态、乳房的解剖特征、与免疫应答相关的遗传特性以及环境因素会改变乳房微生物群的组成等,可以更好地了解乳房不同部位的共生菌群相互作用及与乳腺炎病原体和免疫系统相互作用,为更好地治疗奶牛乳腺炎及相关药物的研发提供有利保障。
| [1] |
GOMES F, SAAVEDRA M J, HENRIQUES M. Bovine mastitis disease/pathogenicity:evidence of the potential role of microbial biofilms[J]. Pathogens and Disease, 2016, 74(3): ftw006. DOI:10.1093/femspd/ftw006 |
| [2] |
BARKEMA H W, SCHUKKEN Y H, ZADOKS R N. Invited review:the role of cow, pathogen, and treatment regimen in the therapeutic success of bovine Staphylococcus aureus mastitis[J]. Journal of Dairy Science, 2006, 89(6): 1880-1895. |
| [3] |
TONG J J, ZHANG H, ZHANG Y H, et al. Microbiome and metabolome analyses of milk from dairy cows with subclinical Streptococcus agalactiae mastitis—potential biomarkers[J]. Frontiers in Microbiology, 2019, 10: 2547. DOI:10.3389/fmicb.2019.02547 |
| [4] |
THOMAS F C, MUDALIAR M, TASSI R, et al. Mastitomics, the integrated omics of bovine milk in an experimental model of Streptococcus uberis mastitis:3.Untargeted metabolomics[J]. Molecular Biosystems, 2016, 12(9): 2762-2779. DOI:10.1039/C6MB00289G |
| [5] |
FALENTIN H, RAULT L, NICOLAS A, et al. Bovine teat microbiome analysis revealed reduced alpha diversity and significant changes in taxonomic profiles in quarters with a history of mastitis[J]. Frontiers in Microbiology, 2016, 7: 480. |
| [6] |
CATOZZI C, BONASTRE A S, FRANCINO O, et al. The microbiota of water buffalo milk during mastitis[J]. PLoS One, 2017, 12(9): e0184710. DOI:10.1371/journal.pone.0184710 |
| [7] |
FRANDSON T L, SPURGEON R D.Anatomy and physiology of farm animals[M]. 5th ed.Philadelphia: Hardcover Hardcover, 1992.
|
| [8] |
任艳.复合乳酸菌乳头清洗剂对奶牛隐性乳房炎的防治研究[D].博士学位论文.呼和浩特: 内蒙古农业大学, 2018: 60-75. REN Y.Study on the application of Lactobacilli-based teat disinfectant for preventing subclinical mastitis of dairy cows[D]. Ph.D.Thesis.Huhhot: Inner Mongolia Agricultural University, 2018: 60-75.(in Chinese) |
| [9] |
曾学琴, 柳陈坚, 杨雪, 等. 高通量测序法检测奶牛乳房炎关联微生物群落结构及多样性[J]. 浙江农业学报, 2019, 31(9): 1437-1445. ZENG X Q, LIU C J, YANG X, et al. Microbial community structure and diversity of mastitis cows by 16S rRNA high-throughput sequencing[J]. Acta Agriculturae Zhejiangensis, 2019, 31(9): 1437-1445 (in Chinese). DOI:10.3969/j.issn.1004-1524.2019.09.06 |
| [10] |
FUQUAY J W.Encyclopedia of dairy sciences[M]. 2nd ed.London: Elsevier Ltd., 2011.
|
| [11] |
WICAKSONO A, SUDARNIKA E, PISESTYANI H, et al. Role of teat dipping after milking for subclinical mastitis control and improving production of dairy cow[J]. Buletin of Animal Science, 2019, 43(2): 135-140. |
| [12] |
PADUCH J H, MOHR E, KRÖMKER V. The association between teat end hyperkeratosis and teat canal microbial load in lactating dairy cattle[J]. Veterinary Microbiology, 2012, 158(3/4): 353-359. |
| [13] |
HAVERKAMP H, PADUCH J H, KLOCKE D, et al. Prevalence of teat end hyperkeratosis in lactating dairy cattle and their association with animal variables[J]. International Journal of Environmental Engineering, 2017, 3(9): 75-82. |
| [14] |
DERAKHSHANI H, FEHR K B, SEPEHRI S, et al. Invited review:microbiota of the bovine udder:contributing factors and potential implications for udder health and mastitis susceptibility[J]. Journal of Dairy Science, 2018, 101(12): 10605-10625. DOI:10.3168/jds.2018-14860 |
| [15] |
KITCHEN B J. Review of the progress of dairy science: bovine mastitis:milk compositional changes and related diagnostic tests[J]. Journal of Dairy Research, 1981, 48: 167-188. DOI:10.1017/S0022029900021580 |
| [16] |
BRAEM G, DE VLIEGHER S, VERBIST B, et al. Culture-independent exploration of the teat apex microbiota of dairy cows reveals a wide bacterial species diversity[J]. Veterinary Microbiology, 2012, 157(3/4): 383-390. |
| [17] |
FYHRQUIST N, RUOKOLAINEN L, SUOMALAINEN A, et al. Acinetobacter species in the skin microbiota protect against allergic sensitization and inflammation[J]. Journal of Allergy and Clinical Immunology, 2014, 134(6): 1301-1309. DOI:10.1016/j.jaci.2014.07.059 |
| [18] |
李庆章. 奶牛乳腺发育与泌乳生物学[M]. 北京: 科学出版社, 2014. LI Q Z. Mammary gland development and lactation biology of dairy cows[M]. Beijing: Science Press, 2014 (in Chinese). |
| [19] |
JAMALI H, BARKEMA H W, JACQUES M, et al. Invited review:incidence, risk factors, and effects of clinical mastitis recurrence in dairy cows[J]. Journal of Dairy Science, 2018, 101(6): 4729-4746. DOI:10.3168/jds.2017-13730 |
| [20] |
RAINARD P. Mammary microbiota of dairy ruminants:fact or fiction?[J]. Veterinary Research, 2017, 48(1): 25. DOI:10.1186/s13567-017-0429-2 |
| [21] |
FALENTIN H, RAULT L, NICOLAS A, et al. Bovine teat microbiome analysis revealed reduced alpha diversity and significant changes in taxonomic profiles in quarters with a history of mastitis[J]. Frontiers in Microbiology, 2016, 7: 480. |
| [22] |
WATTENBURGER K, SCHMIDT R, PLACHETA L, et al. Evaluation of 4 different teat disinfection methods prior to collection of milk samples for bacterial culture in dairy cattle[J]. Journal of Dairy Science, 2020, 103(5): 4579-4587. DOI:10.3168/jds.2019-17338 |
| [23] |
BOUCHARD D S, SERIDAN B, SARAOUI T, et al. Lactic acid bacteria isolated from bovine mammary microbiota:potential allies against bovine mastitis[J]. PLoS One, 2015, 10(12): e0144831. DOI:10.1371/journal.pone.0144831 |
| [24] |
QUIRK T, FOX L K, HANCOCK D D, et al. Intramammary infections and teat canal colonization with coagulase-negative staphylococci after postmilking teat disinfection:species-specific responses[J]. Journal of Dairy Science, 2012, 95(4): 1901-1912. |
| [25] |
WANG L L, SI W, XUE H P, et al. A fibronectin-binding protein (FbpA) of Weissella cibaria inhibits colonization and infection of Staphylococcus aureus in mammary glands[J]. Cellular Microbiology, 2017, 19(8): e12731. DOI:10.1111/cmi.12731 |
| [26] |
SVENNESEN L, LUND T B, SKARBYE A P, et al. Expert evaluation of different infection types in dairy cow quarters naturally infected with Staphylococcus aureus or Streptococcus agalactiae[J]. Preventive Veterinary Medicine, 2019, 167: 16-23. DOI:10.1016/j.prevetmed.2019.03.016 |
| [27] |
SKARBYE A P, THOMSEN P T, KROGH M A, et al. Effect of automatic cluster flushing on the concentration of Staphylococcus aureus in teat cup liners[J]. Journal of Dairy Science, 2020, 103(6): 5431-5439. DOI:10.3168/jds.2019-17785 |
| [28] |
DOYLE C J, GLEESON D, O'TOOLE P W, et al. Impacts of seasonal housing and teat preparation on raw milk microbiota:a high-throughput sequencing study[J]. Applied and Environmental Microbiology, 2016, 83(2): e02694-16. |
| [29] |
TURGEON S L, BRISSON G. Symposium review:the dairy matrix-bioaccessibility and bioavailability of nutrients and physiological effects[J]. Journal of Dairy Science, 2020, 103(7): 6727-6736. DOI:10.3168/jds.2019-17308 |
| [30] |
PIGHETTI G M, ELLIOTT A A. Gene polymorphisms:the keys for marker assisted selection and unraveling core regulatory pathways for mastitis resistance[J]. Journal of Mammary Gland Biology and Neoplasia, 2011, 16(4): 421-432. DOI:10.1007/s10911-011-9238-9 |
| [31] |
DERAKHSHANI H, PLAIZIER J C, DE BUCK J, et al. Association of bovine major histocompatibility complex (BoLA) gene polymorphism with colostrum and milk microbiota of dairy cows during the first week of lactation[J]. Microbiome, 2018, 6: 203. |
| [32] |
WU H C, JIANG K F, ZHANG T, et al. The expression of major histocompatibility complex class Ⅰ in endometrial epithelial cells from dairy cow under a simulating hypoxic environment[J]. Research in Veterinary Science, 2018, 118: 61-65. DOI:10.1016/j.rvsc.2018.01.021 |
| [33] |
KUBINAK J L, STEPHENS W Z, SOTO R, et al. MHC variation sculpts individualized microbial communities that control susceptibility to enteric infection[J]. Nature Communications, 2015, 6: 8642. |
| [34] |
DERAKHSHANI H, TUN H M, CARDOSO F C, et al. Linking peripartal dynamics of ruminal microbiota to dietary changes and production parameters[J]. Frontiers in Microbiology, 2017, 7: 2143. |
| [35] |
ESPOSITO G, IRONS P C, WEBB E C, et al. Interactions between negative energy balance, metabolic diseases, uterine health and immune response in transition dairy cows[J]. Animal Reproduction Science, 2014, 144(3/4): 60-71. |
| [36] |
KHAFIPOUR E, KRAUSE D O, PLAIZIER J C. A grain-based subacute ruminal acidosis challenge causes translocation of lipopolysaccharide and triggers inflammation[J]. Journal of Dairy Science, 2009, 92(3): 1060-1070. DOI:10.3168/jds.2008-1389 |
| [37] |
ZHANG R Y, HUO W J, ZHU W Y, et al. Characterization of bacterial community of raw milk from dairy cows during subacute ruminal acidosis challenge by high-throughput sequencing[J]. Journal of the Science of Food and Agriculture, 2015, 95(5): 1072-1089. |
| [38] |
BUCCIONI A, PALLARA G, PASTORELLI R, et al. Effect of dietary chestnut or quebracho tannin supplementation on microbial community and fatty acid profile in the rumen of dairy ewes[J]. BioMed Research International, 2017, 2017: 4969076. |
| [39] |
XUE F G, PAN X H, JIANG L S, et al. GC-MS analysis of the ruminal metabolome response to thiamine supplementation during high grain feeding in dairy cows[J]. Metabolomics, 2018, 14(5): 1-12. |
| [40] |
PAN X H, XUE F G, NAN X M, et al. Illumina sequencing approach to characterize thiamine metabolism related bacteria and the impacts of thiamine supplementation on ruminal microbiota in dairy cows fed high-grain diets[J]. Frontiers in Microbiology, 2017, 8: 1818. |
| [41] |
ADDIS M F, TANCA A, UZZAU S, et al. Moroni P.The bovine milk microbiota:insights and perspectives from-omics studies[J]. Molecular BioSystems, 2016, 12(8): 2359-2372. |
| [42] |
KATHARINE A L, SIMON C A, JAMES E B, et al. Recycling manure as cow bedding:potential benefits and risks for UK dairy farms[J]. The Veterinary Journal, 2015, 206(2): 123-130. |
| [43] |
VANDERHAEGHEN W, PIEPERS S, LEROY F, et al. Invited review:effect, persistence, and virulence of coagulase-negative Staphylococcus species associated with ruminant udder health[J]. Journal of Dairy Science, 2013, 97(9): 5275-5293. |
| [44] |
ALLARD M, STER C, JACOB C L, et al. The expression of a putative exotoxin and an ABC transporter during bovine intramammary infection contributes to the virulence of Staphylococcus aureus[J]. Veterinary Microbiology, 2013, 162(2/3/4): 761-770. |




