2. 中国农业科学院北京畜牧兽医研究所, 农业农村部奶产品质量安全风险评估实验室(北京), 北京 100193
2. Laboratory of Quality and Safety Risk Assessment for Dairy Products of Ministry of Agriculture and Rural Affairs, Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing 100193, China
反刍动物瘤胃微生物能利用尿素分解产生的氨合成机体可利用的微生物蛋白,所以尿素可作为一种经济的蛋白质饲料替代品[1-2]。瘤胃尿素的分解依赖于瘤胃细菌脲酶,脲酶催化尿素水解的速度是尿素自然分解速度的1014倍[3],尿素饲喂不当容易造成动物氨中毒[4],这严重制约了尿素在动物生产上的广泛应用。脲酶抑制剂是脲酶活性调控研究的前沿。
细菌脲酶是一种镍金属酶[5],通常由结构蛋白(UreA、UreB、UreC)和辅助蛋白(UreD/H、UreF、UreG、UreE)组成[6]。不同微生物来源的脲酶的结构蛋白具有差异,如产气克雷伯氏菌脲酶由UreA、UreB、UreC 3个亚基聚合形成(UreABC)3[7],耶尔森氏菌脲酶为[(UreABC)3]4[8],幽门螺杆菌脲酶为[(UreAB)3]4[9]。细菌脲酶的活性中心位于序列高度保守的UreC亚基[10],每1个活性中心包含2个镍离子,2个镍离子由1个羧化的赖氨酸连接[11],活性中心底物的进入和产物的排出由1个柔性的flap区域调控[12]。脲酶活性中心及flap区域是活性研究的重要靶点。
目前已经报道了多种脲酶抑制剂,其中多为化学合成的化合物。乙酰氧肟酸是目前唯一被批准商用的脲酶抑制剂[13],但是在瘤胃中稳定性差、对动物的健康有影响[14],所以开发安全、高效的脲酶抑制剂是目前研究的重点。自然界蕴含丰富的天然产物资源,其中包括大量具有生物活性、化学结构多样的植物来源的天然化合物,这些化合物在疾病治疗、粮食资源等方面应用广泛[15]。在这种背景下,研究植物来源的天然产物作为脲酶抑制剂调控脲酶活性在动物生产中具有重要价值。本研究拟运用分子对接的方法对植物源化合物库进行筛选,并进一步结合瘤胃细菌脲酶活性试验进行评价,以期获得对瘤胃脲酶具有抑制效果的天然化合物,为天然化合物抑制瘤胃细菌脲酶的研究提供科学依据。
1 试验材料 1.1 试剂材料试验用瘤胃液来自中国农业科学院昌平基地荷斯坦奶牛[泌乳中期,体重(550±23) kg];试验用植物源化合物库信息及植物源天然化合物来源于上海陶素生化科技有限公司,纯度>98%;刀豆脲酶、尿素、4-羟乙基哌嗪乙磺酸(HEPES)粉末、四水合酒石酸钾钠、还原性谷胱甘肽(GSH)、L-半胱氨酸(L-Cys)、硼酸(BA)购于西格玛奥德里奇(上海)贸易有限公司,分析纯;偏磷酸购于国药集团化学试剂有限公司,分析纯;纳氏试剂购于上海阿拉丁生化科技股份有限公司,分析纯;二甲基亚砜(DMSO)购于北京索莱宝科技有限公司;尿素氮含量测试试剂盒购于南京建成生物工程研究所;试验用气体购于北京市北温气体制造厂;试验均使用超纯水。
试验用到的仪器和软件包括Auto Dock vina软件、Discovery Studio 4.5 Client软件、超纯水仪(F6BA99920)、pH计(SG2)、台式高速冷冻离心机(sigma 3-18k)、超声波细胞粉碎机(UP-250)、生化振荡培养箱(HZQ-F100)、磁力搅拌器(IKA big squid)、酶标仪(Thermo Electron Varioskan Flash)、厌氧培养箱(PLAS-LAB 855-AC & 855-ACB)。
1.2 溶液配制50 mmol/L HEPES缓冲液:称取11.9 g HEPES粉末,适量去离子水溶解后,调节pH至7.5,定容至1 L。
50 mmol/L尿素缓冲液:称取0.3 g尿素,用HEPES缓冲液溶解,定容至100 mL。
瑞香素溶液:10 mg瑞香素中加入561.4 μL DMSO,即配制成浓度为100 mmol/L的瑞香素母液,后续使用根据所需浓度进行稀释。
DMSO溶液:DMSO溶液中DMSO的含量与对应试验组化合物中DMSO含量一致。
硫醇试剂溶液:称取L-Cys 1.21 g,HEPES缓冲液定容至100 mL,称取GSH 3.07 g,HEPES缓冲液定容至100 mL,2种硫醇试剂母液浓度均为100 mmol/L,后续使用根据所需进行稀释。
BA溶液:称取BA 0.62 g,HEPES缓冲液定容至100 mL,BA溶液母液浓度为100 mmol/L,后续使用根据所需进行稀释。
2 试验方法 2.1 天然化合物的筛选本试验使用的瘤胃细菌脲酶结构参考实验室之前的同源建模[16]。利用Auto Dock vina软件对瘤胃细菌脲酶进行去除水分子、加氢及计算电荷等处理,对植物源化合物库中的化合物进行同样处理,而后选择细菌脲酶UreC亚基的第308位氨基酸残基至第336位氨基酸残基的区域和活性中心区域进行对接,选择最佳结合模型。进一步测定对接结果较好的4种天然化合物对瘤胃细菌脲酶的抑制效果。
2.2 4种天然化合物对瘤胃细菌脲酶活性的影响 2.2.1 瘤胃细菌脲酶的提取挑选3头荷斯坦瘘管奶牛,采集瘤胃内容物100 mL,通过4层无菌纱布过滤,将滤液(瘤胃液)分装到50 mL无菌离心管中,并置于液氮中保存。
瘤胃液用流水解冻后,300×g离心5 min,收集上清液到新的离心管中,12 000×g离心10 min,弃上清,用25 mL HEPES缓冲液重悬沉淀,12 000×g离心10 min,弃上清,用25 mL HEPES缓冲液重悬沉淀,将其置于冰上,用超声波细胞粉碎机进行破碎(150 W,超声5 s,停5 s,超声10 min),12 000×g离心10 min,收集上清,即瘤胃细菌脲酶。离心过程均为4 ℃。
2.2.2 4种天然化合物对瘤胃细菌脲酶的抑制效果的测定本试验测定脲酶活性采用纳氏试剂法。试验组中,取40 μL瘤胃细菌脲酶于96孔板中,加入40 μL化合物溶液(终浓度10 μmol/L)和100 μL尿素缓冲液(终浓度28 mmol/L);试验阴性对照组中,取40 μL瘤胃细菌脲酶于96孔板中,加入40 μL化合物溶液(终浓度10 μmol/L)和100 μL HEPES缓冲液;脲酶组中,取40 μL瘤胃细菌脲酶于96孔板中,加入40 μL DMSO溶液和100 μL尿素缓冲液;脲酶阴性对照组中,取40 μL瘤胃细菌脲酶于96孔板中,加入40 μL DMSO溶液和100 μL HEPES缓冲液。以上每组设置3个重复,样品加好后,置于生化振荡培养箱中,37 ℃孵育30 min,加入10 μL酒石酸钾钠溶液,混合均匀,加入10 μL纳氏试剂溶液,混合均匀后,室温放置10 min。而后置于酶标仪中,设置420 nm波长,测定吸光度(OD)值。剩余活性计算公式如下:

|
根据4种天然化合物对瘤胃细菌脲酶的抑制结果,对其中抑制效果最好的化合物瑞香素作进一步的评价。
2.3 瑞香素对瘤胃细菌脲酶和刀豆脲酶的半抑制浓度(IC50)值的测定测定瑞香素对瘤胃细菌脲酶的IC50值采用纳氏试剂法,并且以刀豆脲酶作为对照,瑞香素对瘤胃细菌脲酶IC50值测定浓度设置为0、10、20、80、100、160、200 μmol/L,瑞香素对刀豆脲酶IC50值测定浓度设置为0、2、10、20、30、40、45 μmol/L,每个浓度3个重复,测定体系及相应对照设置、剩余活性计算同2.2.2。通过GraphPad Prism 6进行曲线拟合,得到IC50值。
2.4 瑞香素对瘤胃细菌脲酶的抑制类型瑞香素对瘤胃脲酶动力学测定采用纳氏试剂法,瑞香素设0、80、160、200 μmol/L 4个终浓度,尿素设0、0.875、1.750、3.500、7.000、14.000 mmol/L 6个终浓度,每个处理3个重复,其中尿素0 mmol/L为试验阴性对照组,测定体系同2.2.2。根据测得的OD值计算出相应处理下的反应速率(V),在GraphPad Prism 6中进行反应速率的倒数关于尿素浓度(S)的倒数的线性拟合,进一步根据米氏方程计算得到酶动力学参数——米氏常数(Km)和最大反应速率(Vmax),方程如下:
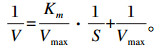
|
本试验选择L-Cys、GSH 2种硫醇试剂和BA这种脲酶Ni2+竞争性抑制剂。脲酶活性测定选择纳氏试剂法,每个处理中添加40 μL瘤胃细菌脲酶和100 μL尿素缓冲液,其中对照组中添加40 μL DMSO溶液,瑞香素组中添加40 μL瑞香素溶液,L-Cys、GSH、BA组中添加相应的溶液40 μL,L-Cys/GSH/BA+瑞香素组中各添加20 μL,瑞香素终浓度为200 μmol/L,L-Cys、GSH、BA终浓度都为0.25 mmol/L,用HEPES缓冲液代替尿素缓冲液作为相对应的阴性对照组,计算方法同2.2.2。利用GraphPad Prism 6进行作图分析。
2.6 瑞香素与瘤胃脲酶的结合位点分析利用Discovery Studio 4.5 Client软件对瑞香素与瘤胃脲酶活性中心对接的结果进行作图,分析瑞香素与瘤胃脲酶相互结合的氨基酸位点及作用力。
2.7 瑞香素对瘤胃微生物尿素氮利用的影响瘤胃液的采集同2.2.1,经过纱布过滤后,用20 mL无菌注射器注射入进过厌氧及灭菌后的血清瓶中,冰上保存。
厌氧稀释液及厌氧培养基配制参考实验室之前的配方[17]。瑞香素溶液和尿素溶液用厌氧稀释液配制,浓度分别为3.8 mmol/L和0.4 mol/L。在厌氧培养箱中将20 mL新鲜瘤胃液与40 mL厌氧培养基混合均匀后分装到螺口培养管中,每管9 mL,试验组中加入500 μL瑞香素溶液,对照组中则加入500 μL DMSO溶液(DMSO含量与瑞香素溶液一致,用厌氧稀释液配制),每组设置3个重复,室温下反应30 min后,加入500 μL尿素溶液,置于厌氧培养箱中39 ℃培养,分别于0、2、4、6、12 h采集200 μL培养液,采集的样品12 000×g,4 ℃离心5 min,收集上清,加入400 μL 25%偏磷酸,于-20 ℃保存。尿素氮含量利用尿素氮测试盒(二乙酰肟比色法)进行测定。
2.8 数据分析试验数据利用SPSS 22.0统计软件进行统计学分析,结果以平均值±标准差表示。在统计分析前,先利用软件中分析-探索-Shapiro-Wilk检验对数据进行正态性检验,服从正态分布的2组数据间的比较采用分析-比较均值-独立样本t检验进行差异性比较;服从正态分布的多组数据间的比较采用分析-比较均值-单因素ANOVA,方差齐时用LSD检验进行多重比较。P < 0.05表示结果差异显著。
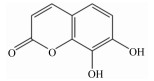
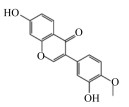
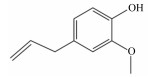
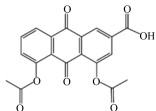
3 结果与分析 3.1 分子对接筛选天然化合物库通过分子对接对植物来源化合物库进行多轮筛选,最后得到4种天然化合物作为瘤胃脲酶候选抑制剂,表 1列出了这4种化合物分子对接得分以及化学结构式。
|
|
表 1 4种天然化合物分子对接得分及化学结构式信息 Table 1 Molecular docking score and chemical formula information of four natural compounds |
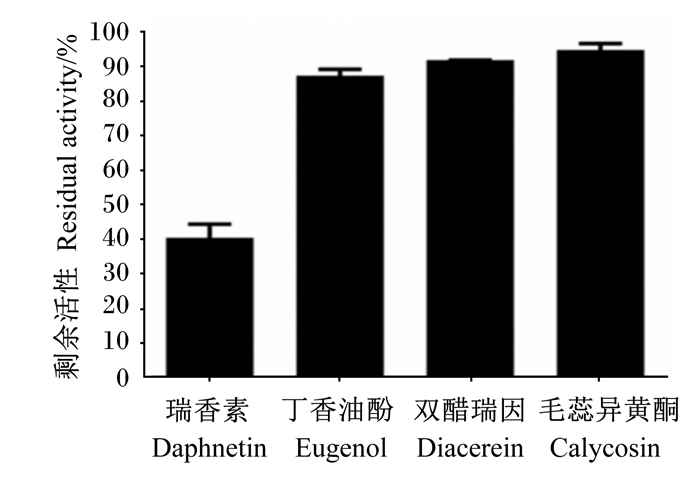
测定分子对接筛选出的4种天然化合物对瘤胃脲酶的抑制效果,结果如图 1所示,在浓度一致的情况下,瑞香素对瘤胃脲酶的抑制效果最好。
|
图 1 4种天然化合物作用后瘤胃细菌脲酶的剩余活性 Fig. 1 Residual activity of ruminal microbial urease interacted by four natural compounds |
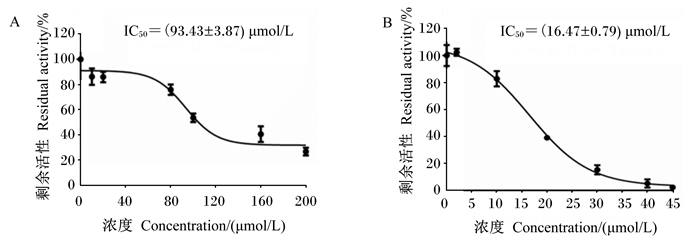
为了表征瑞香素对脲酶的抑制效果,通过测定不同浓度的瑞香素对瘤胃脲酶和刀豆脲酶的抑制率,并计算瑞香素对2种脲酶的IC50值。结果如图 2所示,瑞香素对瘤胃脲酶的IC50值为(93.43±3.87) μmol/L(图 2-A),瑞香素对刀豆脲酶的IC50值为(16.47±0.79) μmol/L(图 2-B)。
|
图 2 不同瑞香素浓度作用后瘤胃细菌脲酶(A)、刀豆脲酶(B)剩余活性 Fig. 2 Residual activity of ruminal microbial urease (A) and jack been urease (B) with different concentrations of daphnetin |
根据测定结果计算,在不存在瑞香素的情况下,瘤胃脲酶动力学参数Km和Vmax分别是0.15 μmol/L和9.09 μmol/(L·min),当瑞香素浓度为80 μmol/L时,Km的值为0.23 μmol/L,Vmax的值为7.49 μmol/(L·min);当瑞香素浓度为160 μmol/L时,Km的值为0.66 μmol/L,Vmax的值为5.40 μmol/(L·min);瑞香素浓度为200 μmol/L时,Km的值为1.02 μmol/L,Vmax的值为3.83 μmol/(L·min) (表 2),即随着瑞香素浓度的升高,Km的值逐渐增大,而Vmax的值逐渐减小。根据双倒数作图结果(图 3)可知,斜率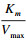
|
|
表 2 不同瑞香素浓度对瘤胃细菌脲酶动力学参数的影响 Table 2 Effects of kinetic parameters of rumen bacterial urease by different concentrations of daphnetin |
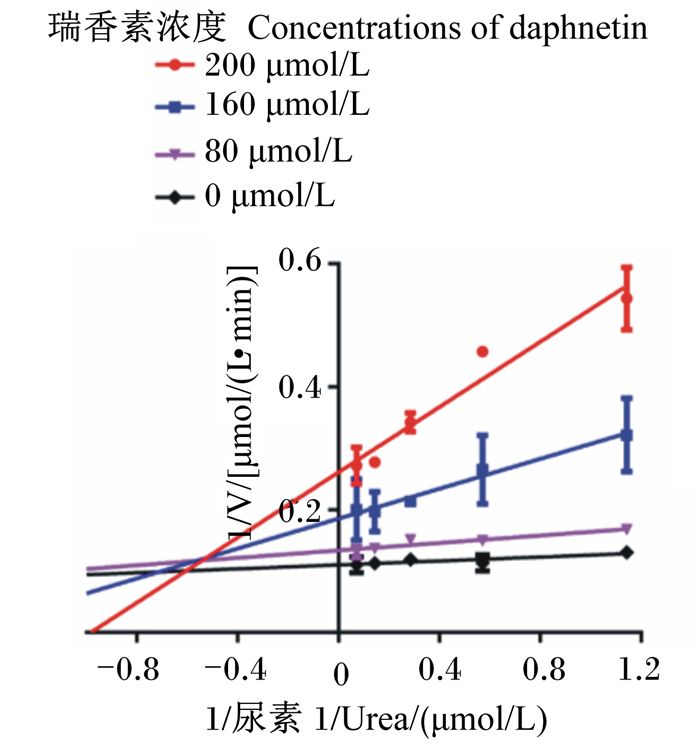
|
图 3 Lineweaver-Burk双倒数作图法测定瑞香素对瘤胃细菌脲酶的酶动力学参数 Fig. 3 Kinetic parameters of rumen bacterial urease tested through Lineweaver-Burk plots of different concentrations of daphnetin |
2种硫醇试剂L-Cys、GSH以及Ni2+竞争性抑制剂BA对瘤胃脲酶均具有一定的抑制效果,但是抑制效果均显著低于瑞香素(图 4)。当瑞香素与L-Cys或GSH联合作用时,瑞香素对瘤胃脲酶的抑制效果显著降低(P < 0.05)(图 4-A和图 4-B),当瑞香素与BA联合作用时,瑞香素对瘤胃脲酶的抑制效果变化不显著(P>0.05)(图 4-C)。这一结果说明,瑞香素对瘤胃脲酶的抑制可能是通过与活性中心周围的巯基相互作用。
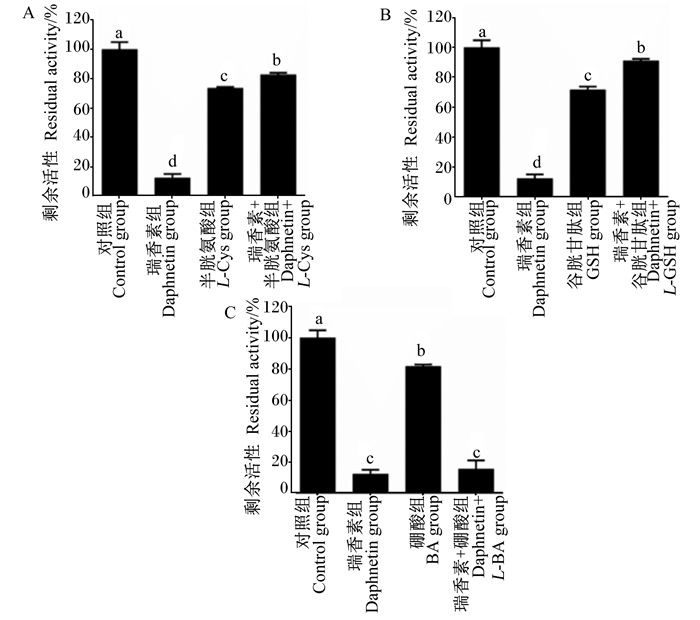
|
标注相同小写字母表示差异不显著(P>0.05),标注不同小写字母表示差异显著(P < 0.05) The same lowercase letters indicated no significant difference (P>0.05), while different lowercase letters indicated significant difference (P < 0.05). 图 4 L-Cys(A)、GSH(B)、BA(C)对瘤胃细菌脲酶活性的影响 Fig. 4 Effects of L-Cys(A)、GSH(B)、BA(C) on ruminal microbial urease activity |
通过Discovery Studio 4.5 Client软件分析得到瑞香素与瘤胃脲酶的最佳结合模式(图 5)。结果显示,瑞香素上的2个羟基分别与氨基酸残基Ala-166和His-245形成氢键,其中1个羟基还与氨基酸残基Lys-216产生离子相互作用(盐桥),瑞香素的苯环结构与氨基酸残基Ala-362形成疏水结合Pi-Alkyl作用,瑞香素的杂环结构能与氨基酸残基Ala-362和Cys-318形成疏水结合Pi-Alkyl作用,并且还能与Ala-362形成碳氢键。
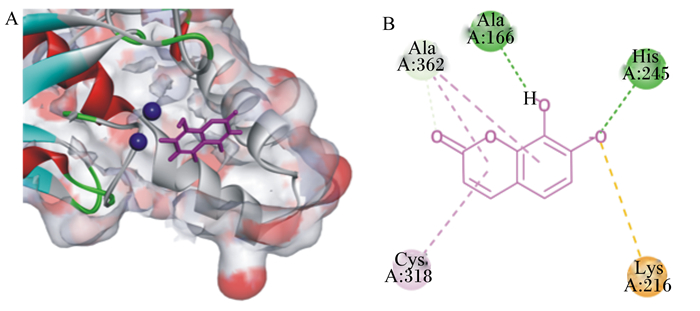
|
A图中深蓝色球体为瘤胃细菌优势脲酶活性中心镍离子,玫红色结构为瑞香素。B图中圆形球体表示脲酶氨基酸残基;实线表示瑞香素;虚线表示瑞香素与氨基酸之间的相互作用,其中深绿色表示氢键,浅绿色表示碳氢键,橙色表示离子相互作用,玫红色表示Pi-Alkyl作用。 The dark blue spheres were Ni2+ of ruminal microbial predominant urease active site and rose part was daphnetin in figure A. In figure B, the round spheres represent urease amino acid residues; the solid line indicates daphnetin; the dotted line represents the interaction between daphnetin and amino acids, where the dark green represents hydrogen bonds, the light green represents carbon-hydrogen bonds, the orange represents ion interactions, and the rose represents Pi-Alkyl interaction. 图 5 瑞香素与瘤胃细菌优势脲酶活性中心结合3D模式图(A)和二维对接图(B) Fig. 5 3D pattern (A) and 2D docking mode (B) of daphnetin binding to rumen bacteria predominant urease active center |
由图 6结果可知,在瘤胃微生物体外厌氧培养试验中,添加瑞香素与只添加等量DMSO的对照组相比,0 h时,2组培养体系中尿素含量差异不显著(P>0.05),而在2、4、6 h时,瑞香素体系中尿素含量显著高于对照组(P < 0.05)。这一结果说明,添加瑞香素在一定时间内能有效地降低瘤胃微生物对尿素的分解。
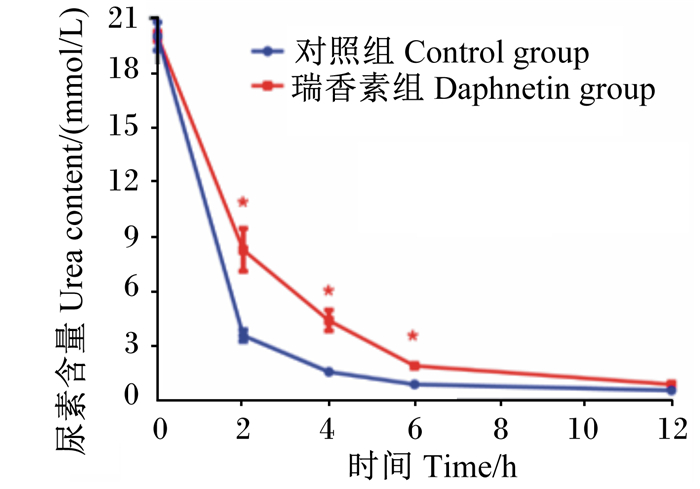
|
标注*表示差异显著(P < 0.05),未标注*表示差异不显著(P>0.05)。 Label * indicated significant difference (P < 0.05), while no label * indicated no significant difference (P>0.05). 图 6 瑞香素对瘤胃微生物体外厌氧培养体系中尿素含量的影响 Fig. 6 Effects of daphnetin on urea content in anaerobic culture system of rumen microorganisms in vitro |
分子对接是目前药物筛选常用的方法[18],这一方法在对脲酶有抑制效果的天然化合物研究中也有诸多应用。如Kataria等[19]利用刀豆脲酶作为目标蛋白,通过分子对接技术从多种天然酚类化合物中筛选出了桑色素、白藜芦醇等化合物。有研究发现,桑色素及其类似物对幽门螺杆菌脲酶具有较好的抑制效果[20],白藜芦醇能通过抑制幽门螺杆菌脲酶而影响幽门螺杆菌在胃肠环境的生存和定植[21],也有研究表明,白藜芦醇的衍生物对刀豆脲酶具有抑制作用[22]。这些研究提示在众多天然化合物种中筛选有效的脲酶抑制剂,分子对接技术是一种有效的方法。虽然脲酶的活性中心具有高度保守的特点,但是同一种化合物对不同微生物脲酶的抑制效果具有一定的差异。Li等[23]研究发现,小檗碱对幽门螺杆菌脲酶的IC50值为2.40 mg/mL,而对刀豆脲酶的IC50值大于3.06 mg/mL,这提示我们靶向性的筛选脲酶抑制剂很有必要。本试验利用瘤胃优势脲酶基因簇的结构基因[24]进行同源建模,将得到的蛋白结构通过分子对接对植物来源化合物库进行筛选,并测定了天然化合物对瘤胃细菌脲酶的抑制效果,最终确定瑞香素对瘤胃细菌脲酶抑制效果最好。
香豆素及其衍生物是分布广泛的天然化合物,具有多种生物活性,一直以来备受化学家和药物化学家的青睐。研究发现,这些天然化合物中有多种具有脲酶抑制效果[25]。瑞香素是一种天然香豆素衍生物,具有抗菌、抗炎、抗肿瘤等特性[26-27]。幽门螺杆菌产生的脲酶有利于幽门螺杆菌在人体胃肠环境中的生存,从而引发多种疾病,瑞香素能有效降低幽门螺杆菌活性[28],并且也有研究发现瑞香素能抑制幽门螺杆菌脲酶活性[29]。本试验研究发现,瑞香素对瘤胃细菌脲酶以及刀豆脲酶均具有抑制效果,并且对刀豆脲酶的抑制效果更好,猜测可能和本试验所用瘤胃细菌脲酶不纯有关。这一现象也出现在天然化合物黄连碱的研究中,Li等[30]研究显示,黄连碱对刀豆脲酶的IC50值低于对幽门螺杆菌脲酶。在后续天然化合物研究中可以考虑对瘤胃细菌脲酶进行提纯后分析。
随着脲酶结构以及抑制剂-脲酶结构的解析,脲酶抑制剂对脲酶产生抑制作用的结构机制有:一方面是脲酶抑制剂进入脲酶活性口袋,与活性中心的镍离子发生相互作用,阻碍底物与镍离子的结合[31];另一方面则是脲酶抑制剂与flap区域发生相互作用,限制这个皮瓣结构的运动而产生抑制作用[32]。有研究表明,含硫醇化合物是一种脲酶活化剂,它能与脲酶氨基酸中的巯基发生相互作用[33],脲酶flap区域的关键氨基酸是1个半胱氨酸,当硫醇化合物和脲酶抑制剂同时存在时,硫醇可通过与半胱氨酸的巯基结合而阻碍脲酶抑制剂与半胱氨酸的相互作用,从而降低脲酶抑制剂对脲酶的抑制效果[34]。本试验结果显示,存在硫醇试剂时,瑞香素的抑制效果降低,说明瑞香素可能是通过与瘤胃细菌脲酶flap区域的半胱氨酸发生相互作用而产生抑制效果,同时分子对接结果也显示瑞香素能与半胱氨酸形成比较强的相互作用。氨基酸残基Lys-216是连接活性中心2个镍离子的关键氨基酸,氨基酸残基His245与其中的1个镍离子发生配位[16]。本试验分子对接结果显示了瑞香素结构中的1个羟基能与这2个关键氨基酸残基发生相互作用,而BA作用下瑞香素对瘤胃脲酶的抑制效果没有明显降低,猜测可能受到瘤胃脲酶纯度较低的影响。很多属于天然化合物的脲酶抑制剂也能通过与脲酶flap区域的半胱氨酸以及与活性中心的关键氨基酸残基发生相互作用而对脲酶产生抑制效果,如槲皮素[35]、芦荟树脂和枸杞中的天然脲酶抑制剂成分[36]、两面针中对脲酶具有抑制效果的天然化合物[37]等。
有研究发现,瘤胃微生物产生的脲酶水解尿素是一个很快的过程,细菌脲酶能在30 min到2 h内快速水解来自饲粮或者血液循环而来的尿素[38],本研究对瘤胃微生物体外厌氧培养的结果显示,培养体系中尿素含量在2 h显著降低。一些脲酶抑制剂如乙酰氧肟酸、正丁基硫代磷酸三胺、铋盐类化合物[39]等能降低尿素的分解,但是这些化合物也存在诸多问题,如乙酰氧肟酸在瘤胃内易降解,瘤胃微生物对正丁基硫代磷酸三胺能产生适应性,还有些化合物对人和动物机体有损害。本试验得到的天然化合物瑞香素在降低尿素分解方面具有很大的潜力,但是瑞香素在瘤胃内的稳定性、微生物对瑞香素是否具有适应性等还需要进一步的研究。
5 结论① 植物源化合物库中对瘤胃细菌脲酶抑制效果最好的化合物是瑞香素,该化合物对瘤胃细菌脲酶的IC50值为93.43 μmol/L,是瘤胃细菌脲酶的混合型抑制剂。
② 瑞香素能通过与瘤胃细菌脲酶活性中心的多个氨基酸残基以及flap区域的半胱氨酸相互作用而阻碍脲酶对尿素的分解。
③ 瑞香素能降低瘤胃微生物体外厌氧培养过程中尿素的分解。
| [1] |
KERTZ A F. Review: urea feeding to dairy cattle: ahistorical perspective and review[J]. The Professional Animal Scientist, 2010, 26(3): 257-272. DOI:10.15232/S1080-7446(15)30593-3 |
| [2] |
JIN D, ZHAO S G, ZHENG N, et al. Differences in ureolytic bacterial composition between the rumen digesta and rumen wall based on ureC gene classification[J]. Frontiers in Microbiology, 2017, 8: 385. |
| [3] |
ZAMBELLI B, MUSIANI F, BENINI S, et al. Chemistry of Ni2+ in urease: sensing, trafficking, and catalysis[J]. Accounts of Chemical Research, 2011, 44(7): 520-530. DOI:10.1021/ar200041k |
| [4] |
PATRA A K. Urea/ammonia metabolism in the rumen and toxicity in ruminants[M]//PUNIYA A, SINGH R, KAMRA D. Rumen microbiology: from evolution to revolution. New Delhi: Springer, 2015: 329-341.
|
| [5] |
KRAJEWSKA B. Urease-aided calcium carbonate mineralization for engineering applications: a review[J]. Journal of Advanced Research, 2018, 13: 59-67. DOI:10.1016/j.jare.2017.10.009 |
| [6] |
ALFANO M, CAVAZZA C. Structure, function, and biosynthesis of nickel-dependent enzymes[J]. Protein Science, 2020, 29(5): 1071-1089. DOI:10.1002/pro.3836 |
| [7] |
MACOMBER L, MINKARA M S, HAUSINGER R P, et al. Reduction of urease activity by interaction with the flap covering the active site[J]. Journal of Chemical Information and Modeling, 2015, 55(2): 354-361. DOI:10.1021/ci500562t |
| [8] |
RIGHETTO R D, ANTON L, ADAIXO R, et al. High-resolution cryo-EM structure of urease from the pathogen Yersinia enterocolitica[J]. Nature Communications, 2020, 11(1): 5101. DOI:10.1038/s41467-020-18870-2 |
| [9] |
MARONEY M J, CIURLI S. Nonredox nickel enzymes[J]. Chemical Reviews, 2014, 114(8): 4206-4228. DOI:10.1021/cr4004488 |
| [10] |
NIM Y S, WONG K B. The maturation pathway of Nickel urease[J]. Inorganics, 2019, 7(7): 85. DOI:10.3390/inorganics7070085 |
| [11] |
MAZZEI L, CIANCI M, BENINI S, et al. Kinetic and structural studies reveal a unique binding mode of sulfite to the Nickel center in urease[J]. Journal of Inorganic Biochemistry, 2016, 154: 42-49. DOI:10.1016/j.jinorgbio.2015.11.003 |
| [12] |
ROBERTS B P, MILLER B R 3rd, ROITBERG A E, et al. Wide-open flaps are key to urease activity[J]. Journal of the American Chemical Society, 2012, 134(24): 9934-9937. DOI:10.1021/ja3043239 |
| [13] |
SHI W K, DENG R C, WANG P F, et al. 3-Arylpropionylhydroxamic acid derivatives as Helicobacter pylori urease inhibitors: synthesis, molecular docking and biological evaluation[J]. Bioorganic & Medicinal Chemistry, 2016, 24(19): 4519-4527. |
| [14] |
REGO Y F, QUEIROZ M P, BRITO T O, et al. A review on the development of urease inhibitors as antimicrobial agents against pathogenic bacteria[J]. Journal of Advanced Research, 2018, 13: 69-100. DOI:10.1016/j.jare.2018.05.003 |
| [15] |
DE FÁTIMA A, TERRA B S, DA SILVA C M, et al. From nature to market: examples of natural products that became drugs[J]. Recent Patents on Biotechnology, 2014, 8(1): 76-88. DOI:10.2174/1872208307666131220163108 |
| [16] |
ZHANG Z Y, LI M, ZHANG X Y, et al. A novel urease inhibitor of ruminal microbiota screened through molecular docking[J]. International Journal of Molecular Sciences, 2020, 21(17): 6006. DOI:10.3390/ijms21176006 |
| [17] |
HAILEMARIAM S, ZHAO S G, WANG J Q. Complete genome sequencing and transcriptome analysis of Nitrogen metabolism of Succinivibrio dextrinosolvens strain Z6 isolated from dairy cow rumen[J]. Frontiers in Microbiology, 2020, 11: 1826. DOI:10.3389/fmicb.2020.01826 |
| [18] |
DE RUYCK J, BRYSBAERT G, BLOSSEY R, et al. Molecular docking as a popular tool in drug design, an in silico travel[J]. Advances and Applications in Bioinformatics and Chemistry: AABC, 2016, 9: 1-11. |
| [19] |
KATARIA R, KHATKAR A. Molecular docking of natural phenolic compounds for the screening of urease inhibitors[J]. Current Pharmaceutical Biotechnology, 2019, 20(5): 410-421. DOI:10.2174/1389201020666190409110948 |
| [20] |
KATARIA R, KHATKAR A. Molecular docking, synthesis, kinetics study, structure-activity relationship and ADMET analysis of morin analogous as Helicobacter pylori urease inhibitors[J]. BMC Chemistry, 2019, 13(1): 45. DOI:10.1186/s13065-019-0562-2 |
| [21] |
PAULO L, OLEASTRO M, GALLARDO E, et al. Antimicrobial properties of resveratrol: a review[M]//MÉNDEZ-VILAS A. Science against microbial pathogens: communicating current research and technological advances. Spain: Formatex Research Center, 2011: 1225-1235.
|
| [22] |
KATARIA R, KHATKAR A. Contribution of resveratrol in the development of novel urease inhibitors: synthesis, biological evaluation and molecular docking studies[J]. Combinatorial Chemistry & High Throughput Screening, 2019, 22(4): 245-255. |
| [23] |
LI C L, XIE J H, CHEN X Y, et al. Comparison of Helicobacter pylori urease inhibition by rhizoma coptidis, cortex phellodendri and berberine: mechanisms of interaction with the sulfhydryl group[J]. Planta Medica, 2016, 82(4): 305-311. |
| [24] |
ZHANG X Y, ZHAO S G, HE Y, et al. Substitution of residues in UreG to investigate UreE interactions and Nickel binding in a predominant urease gene cluster from the ruminal metagenome[J]. International Journal of Biological Macromolecules, 2020, 161: 1591-1601. DOI:10.1016/j.ijbiomac.2020.07.260 |
| [25] |
ALOMARI M, TAHA M, IMRAN S, et al. Design, synthesis, in vitro evaluation, molecular docking and ADME properties studies of hybrid bis-coumarin with thiadiazole as a new inhibitor of urease[J]. Bioorganic Chemistry, 2019, 92: 103235. |
| [26] |
WANG Y K, CHEN J, CHEN J D, et al. Daphnetin ameliorates glucocorticoid-induced osteoporosis via activation of Wnt/GSK-3β/β-catenin signaling[J]. Toxicology and Applied Pharmacology, 2020, 409: 115333. |
| [27] |
WANG G Z, WANG X Y, XUN Z K, et al. The prospect of daphnetin against drug-resistant bacteria: a mini review[J]. Research & Reviews: Journal of Microbiology and Biotechnology, 2019, 8(3): 34-35. |
| [28] |
WANG G Z, PANG J, HU X X, et al. Daphnetin: a novel anti-Helicobacter pylori agent[J]. International Journal of Molecular Sciences, 2019, 20(4): 850. |
| [29] |
JADHAV S G, MESHRAM R J, GOND D S, et al. Inhibition of growth of Helicobacter pylori and its urease by coumarin derivatives: molecular docking analysis[J]. Journal of Pharmacy Research, 2013, 7(8): 705-711. |
| [30] |
LI C L, HUANG P, WONG K, et al. Coptisine-induced inhibition of Helicobacter pylori: elucidation of specific mechanisms by probing urease active site and its maturation process[J]. Journal of Enzyme Inhibition and Medicinal Chemistry, 2018, 33(1): 1362-1375. |
| [31] |
MAZZEI L, CIANCI M, CONTALDO U, et al. Urease inhibition in the presence of N-(n-butyl) thiophosphoric triamide, a suicide substrate: structure and kinetics[J]. Biochemistry, 2017, 56(40): 5391-5404. |
| [32] |
MAZZEI L, CIANCI M, MUSIANI F, et al. Inactivation of urease by catechol: kinetics and structure[J]. Journal of Inorganic Biochemistry, 2017, 166: 182-189. |
| [33] |
DÍAZ-SÁNCHEZ Á G, ALVAREZ-PARRILLA E, MARTÍNEZ-MARTÍNEZ A, et al. Inhibition of urease by disulfiram, an FDA-approved thiol reagent used in humans[J]. Molecules, 2016, 21(12): 1628. |
| [34] |
TAN L H, LI C L, CHEN H B, et al. Epiberberine, a natural protoberberine alkaloid, inhibits urease of Helicobacter pylori and jack bean: susceptibility and mechanism[J]. European Journal of Pharmaceutical Sciences, 2017, 110: 77-86. |
| [35] |
XIAO Z P, WANG X D, PENG Z Y, et al. Molecular docking, kinetics study, and structure-activity relationship analysis of quercetin and its analogous as Helicobacter pylori urease inhibitors[J]. Journal of Agricultural and Food Chemistry, 2012, 60(42): 10572-10577. |
| [36] |
REHMAN N U, KHAN A, AL-HARRASI A, et al. Natural urease inhibitors from Aloe vera resin and Lycium shawii and their structural-activity relationship and molecular docking study[J]. Bioorganic Chemistry, 2019, 88: 102955. |
| [37] |
LU Q, LI C L, WU G S. Insight into the inhibitory effects of Zanthoxylum nitidum against Helicobacter pylori urease and Jack bean urease: kinetics and mechanism[J]. Journal of Ethnopharmacology, 2020, 249: 112419. |
| [38] |
PATRA A K, ASCHENBACH J R. Ureases in the gastrointestinal tracts of ruminant and monogastric animals and their implication in urea-N/ammonia metabolism: a review[J]. Journal of Advanced Research, 2018, 13: 39-50. |
| [39] |
GETAHUN D, ALEMNEH T, AKEBEREGN D, et al. Urea metabolism and recycling in ruminants[J]. Biomedical Journal of Scientific & Technical Research, 2019, 20(1): 14790-14796. |