2. 福建省家畜传染病防治与生物技术重点实验室, 龙岩 364000;
3. 福建省龙岩市动物疫病预防控制中心, 龙岩 364000;
4. 福建龙岩金和动物饲料有限公司, 龙岩 364000
2. Key Laboratory of Fujian University Preventive Veterinary Medicine and Biotechnology, Longyan 364000, China;
3. Animal Disease Prevention and Control Center of Longyan City, Longyan 364000, China;
4. Fujian Longyan Jinhe Animal Feed Co., Ltd., Longyan 364000, China
随着减抗、无抗时代的来临,更多的抗生素替代品被大量应用在动物饲粮中,其中菌酶协同发酵饲料作为一种新型的绿色饲料添加剂在“禁抗替抗”中具有良好的应用前景。菌酶协同发酵其微生物益生菌与酶解技术相结合的功能,是发酵饲料重大的技术突破点,是将来生物饲料发展的一个重要方向[1]。大量研究证实,在饲粮中添加复合酶特别是非淀粉多糖酶(NSP)对畜禽生产及品质具有积极的影响。研究表明,在肉鸡饲粮中添加NSP,能弥补机体分泌NSP不足的缺陷,消除NSP的黏度效应,间接提高饲粮营养物质的消化率,从而促进肉鸡生长[2-3]。另有报道表明,菌酶协同发酵饲料能够促进鸡肠上皮细胞的形成,提高营养物质的消化率,维持肠道微生态平衡,改善鸡的肠道健康并预防肠道病原菌感染[4]。但已有的研究中探讨菌酶协同发酵饲料影响畜禽生长及肠道功能的机理研究报道较少。
此外,随着人们生活水平的提高,消费者越来越注重鸡肉的质量,对肉质有了更高的要求。然而,随着动物生产性能的不断提高,其风味品质呈现出逐渐降低的趋势[5-6]。因此,既能充分发挥肉鸡的生长潜力又能够改善鸡肉风味品质的各类型发酵饲料添加剂在肉鸡饲养中越来越受到人们的重视。研究表明,发酵饲料通过好氧厌氧发酵对底物进行深层发酵,对肉鸡的生长性能、消化代谢、肉品质、肌肉脂肪酸组成等方面均有不同程度的改善,还能显著提高肉鸡的屠宰率、腿肌率以及胸肌率[7]。
高通量测序已经成为一种常规的试验技术用于生命科学领域,包括转录水平和肠道菌群分析等。转录组测序(RNA-Seq)通过高通量测序来全面快速地获得特定细胞或组织在某一状态下几乎所有转录的序列信息和表达信息,准确地分析基因的表达差异[8];而基于16S rRNA基因高通量测序已成为动物肠道菌群组成及多样性分析的常用技术[9]。因此,本研究将河田鸡作为研究对象,研究菌酶协同发酵饲料对肉鸡肌肉风味物质含量、肠道转录组及菌群组成的影响,探讨其作用肉鸡肠道功能的机理,以期为菌酶协同发酵饲料在肉鸡生产中的推广应用提供理论依据。
1 材料与方法 1.1 试验材料NSP组成及其活性:木聚糖酶活性10 000~20 000 U/g,β-葡聚糖酶活性1 000~1 500 U/g,甘露聚糖酶活性200~300 U/g,纤维素酶活性2 000~4 000 U/g,果胶酶活性100~300 U/g。
发酵装置:选择带有单向透气孔的PE膜,该膜5~7层厚,透气口径9~12 mm,透氧量2.25~4.42 cm3/(m2·d·Pa),排气压力6 644~7 350 Pa。
菌酶协同发酵饲料:采用某公司自动化生产工艺设备进行生产,具体流程如下:1)发酵复合菌株(乳酸菌6.2×109~7.7×109 CFU/g、酵母菌3.3×108~4.6×108 CFU/g和芽孢杆菌2.5×108~4.2×108 CFU/g的复合菌剂)提前用25~30 ℃红糖水活化;2)发酵原料(玉米、豆粕、麸皮、糖蜜豆皮、米糠粕)与1.0%复合多维、0.07% NSP和前述活化好的发酵复合菌株共同混合,并补充水分至30%,搅拌均匀;3)混合好的物料装入带有单向透气孔的PE膜袋中密封,在25 ℃下发酵5~7 d得成品。发酵产品中主要营养成分含量:粗蛋白质≥16.0%、粗纤维12.5%、粗灰分≤12.0%、有机酸≥13.8%、酸溶蛋白12.0%、钙2.5%~3.5%、总磷≥10.1%、氯化钠0.1%~0.8%、赖氨酸≥0.35%,最终活菌总数7.3×109~8.1×109 CFU/g。
1.2 试验动物及饲养管理试验在福建省长汀县众益农牧有限公司饲养的长汀河田肉鸡养殖场内进行。试验选取体重相近、健康无病的60日龄河田肉鸡18 000羽,随机分为2组,每组6个重复,每个重复1 500羽,饲养于单层平养鸡笼内。对照组饲喂玉米-豆粕型基础饲粮(组成及营养水平见表 1),试验组在基础饲粮中添加5.0%菌酶协同发酵饲料,混合均匀饲喂,试验期90 d。自由采食,自由饮水,饲养管理按场内统一技术规程进行。试验结束后,每组随机选取体重相似的肉鸡6只(每个重复1只)进行屠宰,取左侧胸肌、髂胫外侧肌同一部位的腿肌样品用于风味物质含量测定。再用无菌剪刀将所屠宰的6组肉鸡分别沿腹底部向上剖开腹腔,剪切鸡的整个肠道,其中3组整个肠道分别装入无菌封口塑料袋中;另外3组肠道再分别剪切下十二指肠、空肠、回肠、盲肠和直肠组织,挑起各肠道内容物于无菌离心管中。2类样品均先液氮预冻-80 ℃保存,再用干冰运输送至北京奥维森基因科技有限公司,整个肠道样品用于转录组测序,各肠道不同区段内容物用于菌群组成分析。
|
|
表 1 基础饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of the basal diet (DM basis) |
分别使用氨基酸分析仪(茚三酮衍生系统,兰博PCR3+)和全自动凯氏定氮仪(海能K1160)参照中国食品安全国家标准食品中GB 5009.124—2016《食品中氨基酸的测定》和GB 5009.5—2016《食品中蛋白质的测定》的方法测定肌肉中的氨基酸和蛋白质含量;采用高效液相色谱仪(安捷伦1260)参照GBDB 37/T 3816—2019 《畜禽肌肉中肌苷酸含量测定高效液相色谱法》和GB 5009.169—2016《食品中牛磺酸的测定》的方法测定肌肉中的肌苷酸和牛磺酸含量;采用气相色谱仪(Agilent Technologies 7890B)参照GB 5009.128.2016 《食品中胆固醇的测定》和GB 5009.168—2016《食品中脂肪酸的测定》的方法测定肌肉中胆固醇和不饱和脂肪酸含量;以上指标测定均送至中国厦门谱尼测试有限公司检测。
1.4 转录组测序分析将2组肉鸡整个肠道样本分别放置液氮中,边研磨边粉碎,再取粉碎后部分样品采用RNA 6000 Nano Kit试剂盒(Agilent公司,型号5067-1511)提取总RNA,通过Nanodrop和Agilent 2100检测RNA纯度[吸光度(OD)260/280、OD260/230]及RNA片段长度,样品检测合格后委托北京奥维森基因科技有限公司完成文库的构建和测序工作,建好的测序文库采用Illumina HiSeq 4000高通量测序平台进行转录组测序。去除带有测序接头(adapter)、N(不确定碱基)含量≥10%和低质量碱基(Q≤20)含量大于50%的测序原始数据后,采用Tophat2软件将测序序列和转录组序列进行比对,随后采用Cufflinks软件组装比对结果,对测序结果进行RNA-Seq相关性检查。采用HTSeq软件对各样品进行基因表达水平的分析,使用FPKM值为1作为判断基因是否表达的阙值,只分析FPKM>1的基因[10],并使用DESeq进行差异表达分析,筛选条件为qvalue < 0.05[11]。基于Wallenius non-central hyper-genometic发布的方法,通过GOseq软件对差异表达基因(DEGs)进行GO富集分析[12];将测序结果与KEGG数据库比对,通过超几何检验,找出差异表达基因当中最显著富集的通路,对差异表达基因进行功能注释和分类[13]。
1.5 肠道菌群分析分别取3组鸡肠道十二指肠、空肠、回肠、盲肠和直肠组织内容物,采用MoBio PowerSoil DNA Isolation Kit(100)提取其细菌总DNA。获得总DNA后委托北京奥维森基因科技有限公司进行具体分析工作,采用Miseq PE300高通量测序平台进行建库,应用16S rRNA基因V3~V4可变区进行高通量测序,经过滤数据中的嘈杂序列,去杂、拼接获得高质量有效序列再通过Usearch软件,按照97%的相似度CD-HIT分类方法进行操作分类单元(OTU)聚类,以丰度最大的序列为代表序列通过RDP Classifier与GreenGene数据库对OTU代表序列进行物种注释(阙值为0.8~1.0)[14],计算OTU数量、Shannon指数、Simpson指数和Chao l指数等α多样性指数[15],并进行主成分分析(PCA)(R软件,vegan包)[16]和线性判别分析(LEFSe)(LDA score>4)[17],探讨肠道各区段菌群组成的差异性。
1.6 统计分析所得数据以平均值±标准误表示,采用Statistics 17.0软件中的Independent-Sample t Test独立样本的t检验分析试验组与对照组之间的差异,P < 0.05表示差异显著。
2 结果与分析 2.1 菌酶协同发酵饲料对肉鸡肌肉风味物质含量的影响由表 2可知,与对照组相比,试验组肉鸡的各项风味物质含量均有增长或改善,其中,试验组胸肌的总氨基酸、风味氨基酸、肌苷酸、不饱和脂肪酸含量和腿肌的总氨基酸、风味氨基酸、蛋白质、胆固醇、牛磺酸和不饱和脂肪酸含量差异均达显著水平(P < 0.05),表明菌酶协同发酵饲料对肉鸡肌肉风味具有良好的改善作用。
|
|
表 2 菌酶协同发酵饲料对肉鸡肌肉风味物质含量的影响 Table 2 Effects of fermented feed by bacteria coupled fermentation with enzymes on contents of flavor compounds in muscle of broiler |
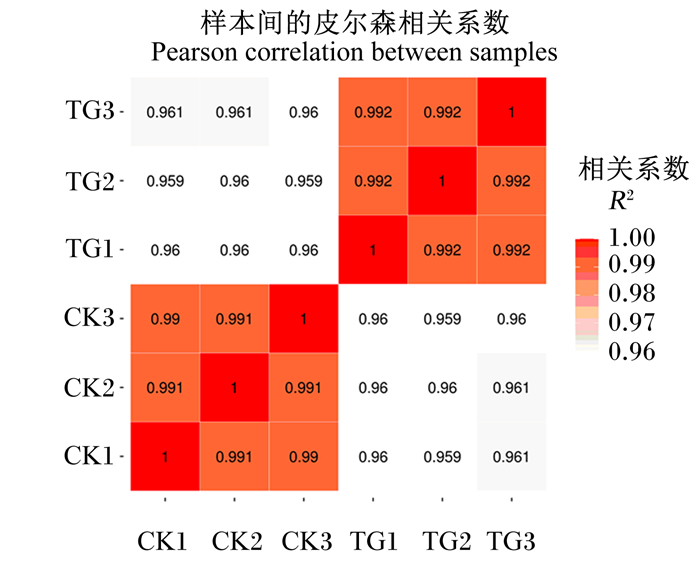
对照组和试验组各样品总RNA提取效果较好,其OD260/280在2.027~2.063,OD260/230在1.450~2.254,RNA完整值(RIN)在9.8~10.0,28S/18S在2.2~4.0,样品质量满足建库测序要求。RNA样品反转录成cDNA,将构建好的cDNA文库,通过Illumina HiSeq 4000测序平台进行转录组测序。测序得到的下机数据去除带有接头(adapter)、N(不确定碱基)含量≥10%和低质量碱基(Q≤20)含量超过50% reads的原始序列,得到质控序列(clean reads)。6个样本均获得了7.00 G以上容量的原始数据,GC含量都在50.00%以上,平均错误率均为0.03%,Q20与Q30分别都在97.00%及93.00%以上(表 3),表明转录组测序所获得数据的质量较好,可用于后续分析。2组样本之间的相关系数(R2)大于0.960,表明结果可靠且样本选择合理(图 1)。
|
|
表 3 2组样品测序数据统计及质量检验结果 Table 3 Sequencing data statistics and quality test results of two groups of samples |
|
CK1、2、3和TG1、2、3分别为对照组和试验组样品。 CK1, 2, 3 and TG1, 2, 3 were samples in control and test groups, respectively. 图 1 2组样品间相关系数 Fig. 1 Correlation coefficient between two groups of samples |
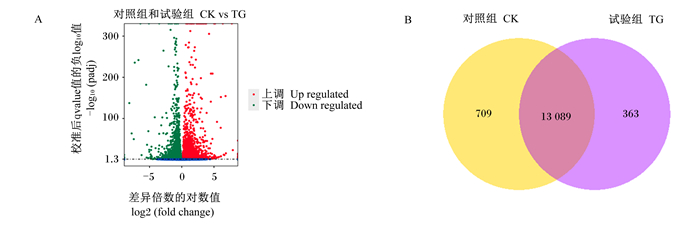
对基因表达量进行表转化处理后,根据|log2(fold change)|>1且qvalue < 0.05的原则筛选差异表达基因。与对照组比较,试验组共筛选出7 751个差异表达基因,其中试验组有3 833个基因上调表达(图 2-A),占总差异表达49.45%,3 918个基因下调表达,占总差异表达50.55%。以FPKM>1作为判断基因表达的标准,2个样品共同表达基因13 089个,试验组特有的表达基因363个(图 2-B)。
|
图 2 2组样品差异表达基因火山图(A)和韦恩图(B) Fig. 2 Volcano diagram (A) and Venn diagram (B) of DEGs of samples in two groups |
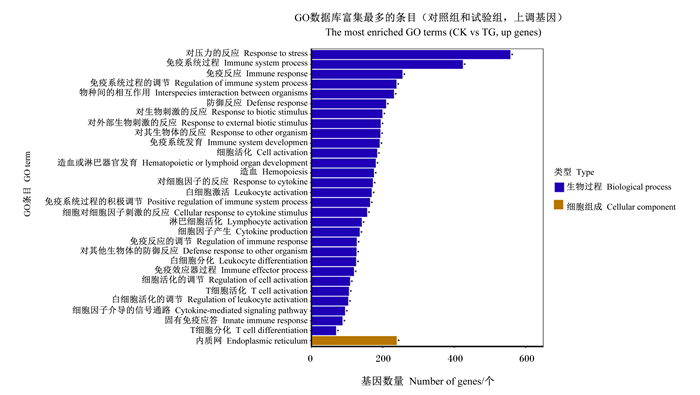
对2组样品差异表达基因进行GO富集分析,结果显示,总共有12 782个上调差异表达基因被富集到3 167个GO term。对富集最显著的30个GO term功能富集分析表明,富集的基因条目集中在与生物过程(biological process)相关的条目,个别在细胞组成(cellular component)相关的条目。在生物过程类别中,富集到差异表达基因最多的类别是对压力的反应(response to stress)、免疫系统过程(immune system process)和免疫反应(immune response)(图 3)。
|
图 3 上调差异表达基因的GO功能富集分析 Fig. 3 GO functional enrichment analysis of up-regulated DEGs |
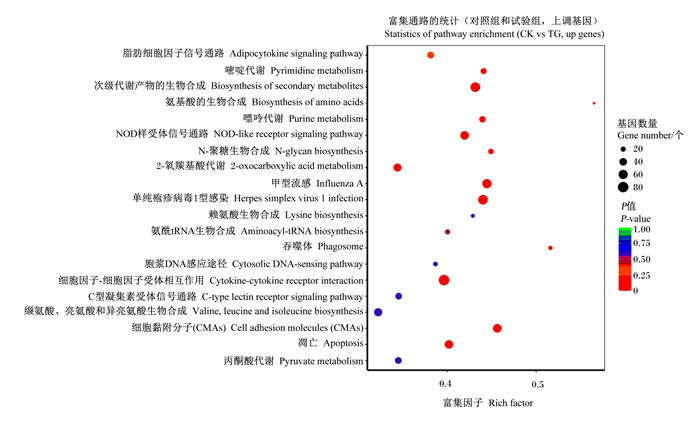
2组样品上调差异表达基因KEEG富集分析表明,共有2 772个基因被注释到162条KEGG代谢通路中,其中2组富集最显著的20条KEGG代谢通路如图 4所示。与新陈代谢有关的信号通路11个,包括脂肪细胞因子信号通路(adipocytokine signaling pathway)、嘧啶代谢(pyrimidine metabolism)、次级代谢产物的生物合成(biosynthesis of secondary metabolites)、氨基酸的生物合成(biosyn-thesis of amino acids)、嘌呤代谢(purine metabo-lism)、N-聚糖生物合成(N-glycan biosynthesis)、2-氧羰基酸代谢(2-oxocarboxylic acid metabolism)、赖氨酸生物合成(lysine biosynthesis)、氨酰tRNA生物合成(aminoacyl-tRNA biosynthesis)、丙酮酸代谢(pyruvate metabolism)及缬氨酸、亮氨酸和异亮氨酸生物合成(valine, leucine and isoleucine biosynthesis),其中富集差异表达基因数目最多的信号通路为次级代谢产物的生物合成,共富集到差异表达基因66个,与对照组差异显著(P < 0.05);富集程度最高的信号通路是氨基酸的生物合成,共富集到差异表达基因13个,与对照组差异显著(P < 0.05)。与免疫有关信号通路9个,包括NOD样受体信号通路(NOD-like receptor signaling pathway)、甲型流感(influenza A)、单纯疱疹病毒1型感染(herpes simplex virus 1 infection)、吞噬体(phagosome)、胞浆DNA感应途径(cytosolic DNA-sensing pathway)、细胞因子-细胞因子受体相互作用(cytokine-cytokine receptor interaction)、C型凝集素受体信号通路(c-type lectin receptor signaling pathway)、细胞黏附分子(cell adhesion molecules)和凋亡(apoptosis),其中富集差异表达基因数最多的信号通路为细胞因子-细胞因子受体相互作用(cytokine-cytokine receptor interaction),共富集到差异表达基因80个,与对照组差异显著(P < 0.05);富集程度最高的信号通路是吞噬体(phagosome),共富集到差异表达基因16个,与对照组差异显著(P < 0.05)。
|
图 4 上调差异表达基因的KEGG功能归类 Fig. 4 KEGG functional classification of up-regulated DEGs |
通过对测序所得的序列进行预处理,去除低质量序列和冗余序列,2组样品30个样本Index完全匹配的有效序列量见表 4所示。各样本菌群组成差异性较大,2组肠道各区段菌群的数量均为盲肠>直肠>回肠>空肠>十二指肠;与对照组相比,试验组各对应肠道区段OTUs数目和Chao1指数增加,表明各样本的菌群数量增加;试验组各对应肠道区段Shannon和Simpson指数略小于对照组,表明各样本的菌群的种类略有下降。总之,益生菌发酵饲料的添加改变了肉鸡肠道不同区段的微生物菌群组成。
|
|
表 4 各样本的测序数据及OTUs、多样性指数统计 Table 4 Sequencing data, OTUs and diversity index statistics of each sample |
从细菌的有效序列长度图可以看出,细菌序列主要分布在400~440 bp(图 5-A)。利用Mothur软件做稀释曲线分析,从各样本中细菌的稀释度(rarefaction)曲线(图 5-B)可以看出,30条曲线均趋向平坦,2组样本不同肠道区段样本曲线均较为集中的聚类在一起,表明所测序数据量合理,大多数细菌类群包括在各样本文库中,能够比较真实地反映样本的微生物群落多样性。
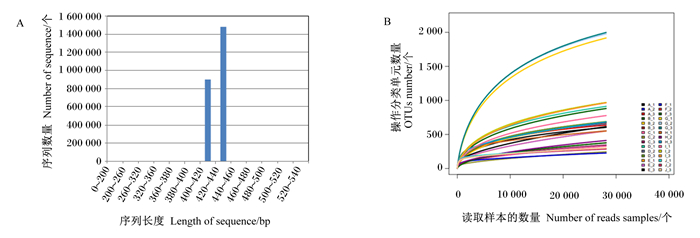
|
图 5 各样本序列长度分布图(A)和稀释度曲线(B) Fig. 5 Sequence length distribution (A) and rarefaction curves (B) of each sample |
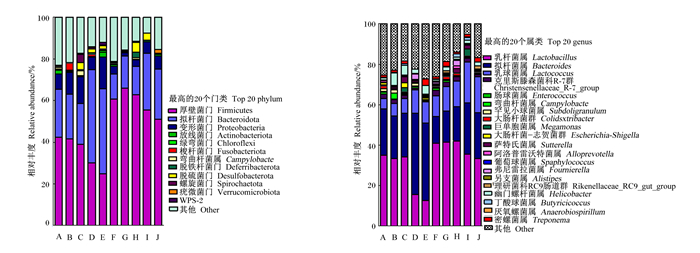
对照组和试验组肉鸡各肠道不同区段样本均值的OTUs物种注释结果如图 6所示,2组样品中均存在着占很大比例的未能培养(uncultured)和未定义(unidentified)菌群,从门水平上看,2组样品菌群中相对丰度最高的为厚壁菌门(Firmicutes)(24%~66%)、拟杆菌门(Bacteroidota)(12%~45%)和变形菌门(Proteobacteria)(1%~16%);与对照组相比,试验组各对应肠道区段样本菌群的厚壁菌门相对丰度增加,拟杆菌门、变形菌门和放线菌门(Actinobacteriota)相对丰度降低。从属的水平上看,2组样品菌群的优势菌属均为乳杆菌属(Lactobacillus)(12%~43%)、拟杆菌属(Bacteroides)(13%~41%)和乳球菌属(Lactococcus)(5%~21%)。与对照组相比,试验组各对应肠道区段样本菌群的乳杆菌属、乳球菌属、克里斯滕森菌科R-7群(Christensenellaceae_R-7_group)、另支菌属(Alistipes)等有益菌属相对丰度增加,拟杆菌属、肠球菌属(Enterococcus)、弯曲杆菌属(Campylobacte)、罕见小球菌属(Subdoligranulum)、大肠杆菌-志贺菌群(Escherichia-Shigella)、萨特氏菌属(Sutterella)、密螺菌属(Treponema)等病原菌属相对丰度降低。
|
A:对照组十二指肠;B:对照组空肠;C:对照组回肠;D:对照组盲肠;E:对照组直肠;F:试验组十二指肠;G:试验组空肠;H:试验组回肠;I:试验组盲肠;J:试验组直肠。下图同。 A: duodenum of control group; B: jejunum of control group; C: ileum of control group; D: cecum of control group; E: rectum of control group; F: duodenum of test group; G: jejunum of test group; H: ileum of test group; I: cecum of test group; J: rectum of test group. The same as below. 图 6 2组样本在门、属水平上主要微生物菌群比较 Fig. 6 Comparison of major microbial flora of samples in 2 groups on phylum level and genus level |
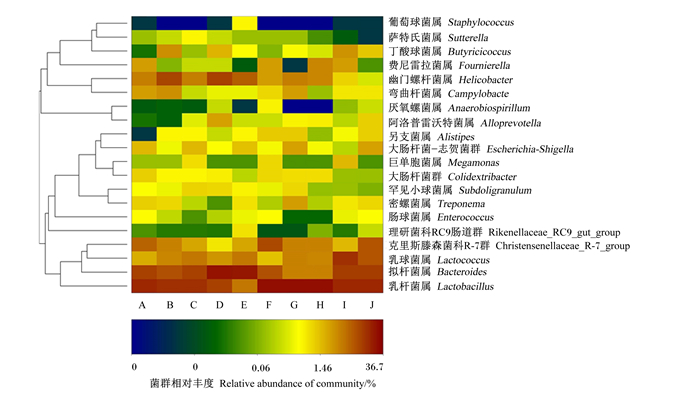
使用R语言vegan包,通过vegdist和hclust进行距离计算和聚类分析,对各样本菌群相对丰度前20的属进行聚类分析到Heatmap(图 7),结果表明,各样本菌群组成差异较大,其中相对丰度最高的是乳杆菌属、拟杆菌属、乳球菌属和克里斯滕森菌科R-7群。与对照组相比,试验组中乳杆菌属、乳球菌属、克里斯滕森菌科R-7群和另支菌属等益生菌群的相对丰度增加,拟杆菌属、大肠杆菌-志贺菌群、厌氧螺菌属(Anaerobiospirillum)、弯曲杆菌属和幽门螺杆菌属(Helicobacter)等肠道病原菌相对丰度降低,所得结果与前述的各样本OTUs物种注释结果一致。
|
图 7 各样本属水平微生物菌群Heatmap分析 Fig. 7 Heatmap analysis of microbial flora on genus level of each sample |
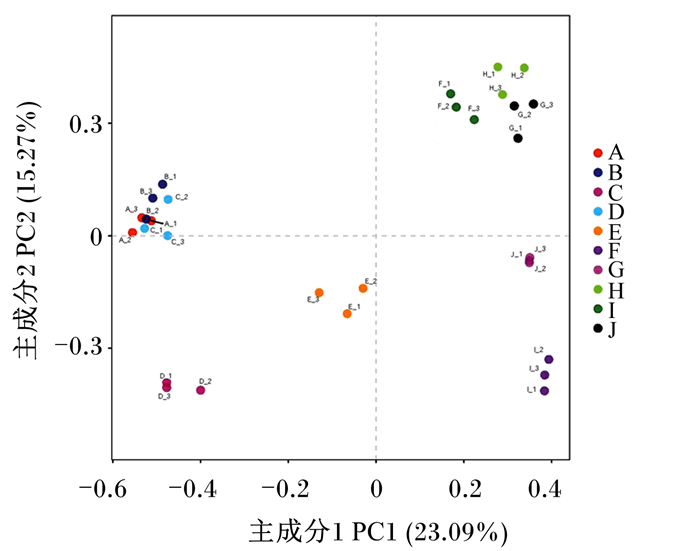
采用R语言PCA统计分析和作图,对照组和试验组肉鸡各肠道不同区段菌群的组成差异性PCA结果如图 8所示。同一肠道区段3个取样样本菌群能较好地聚类在一起,但2组间各对应肠道区段样本的菌群组成差异明显,无法较好地聚类在一起,表明试验组添加菌酶协同发酵饲料饲喂后其肠道菌群的组成明显改变。另外,2组样品的十二指肠、空肠和回肠菌群组成均较为相近,可以较好地聚类在一起,但盲肠、直肠与它们的菌群组成差异较大,无法聚类在一起。
|
图 8 各样本属水平微生物菌群PCA Fig. 8 PCA of microbial flora on genus level of each sample |
2组样品各样本LEFSe分析表明(图 9),共富集了13个属菌群,其中弯曲杆菌属、拟杆菌属、肠球菌属、萨特氏菌属、罕见小球菌属、大肠杆菌-志贺菌群、厌氧螺菌属和密螺菌属肠道常见病原菌群在对照组肠道样本中的相对丰度最高,而乳球菌属、另支菌属、弗尼雷拉菌属(Fournierella)、乳杆菌属和克里斯滕森菌科R-7群在试验组中相对丰度最高,所得结果与前述结果基本一致。
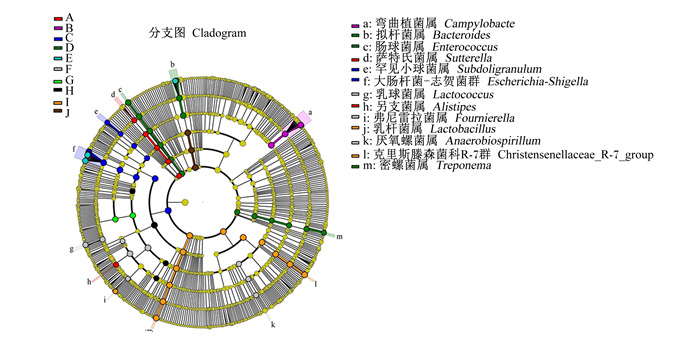
|
图 9 各样本微生物菌群的LEFSe分析 Fig. 9 LEFSe analysis of microbial flora of each sample |
近年来,菌酶协同发酵成为了生物饲料发酵技术研究的热点,菌酶协同作用是微生物参与的生命活动过程和酶参与的生物化学过程的有机结合,在协同过程中涉及到多种基质、菌种和酶,其机理极其复杂。研究已证实,饲粮中添加菌酶协同发酵饲料对畜禽的生长性能具有良好的促进作用[18-19],但对畜禽肉品质的影响及机理的研究报道较少。本研究表明,与对照组相比,添加菌酶协同发酵饲料饲喂,肉鸡肌肉的各项风味物质含量、肉品质指标均有明显的增长或改善。推测原因一方面是复合微生物有益菌群如乳酸菌产生了乳酸、脂肪酸等代谢物[20],由畜禽吸收并进一步沉积到肌肉中,促进畜禽肌肉中风味物质的生成;另一方面,NSP的加入,饲料原料中的淀粉多糖得到了切割、分解,黏度降低,包裹的营养物质被溶解释放,促进了消化吸收[21],有利于肌苷酸、牛磺酸等一些代谢中间产物的风味物质形成;最后,发酵饲料的添加降低了肌肉中的胆固醇类物质含量,提高了不饱和脂肪酸的含量,改善肌肉营养成分,表明发酵饲料及其代谢产物可能可以直接或间接地参与畜禽机体内的胆固醇、脂肪酸合成与代谢调控,进一步影响肉鸡肌肉中脂肪酸的组成,从而影响肉品质。
转录组测序表明,压力的反应、免疫系统过程和免疫反应是试验组肉鸡整个肠道上调差异表达基因的GO功能富集的最多的类别。KEGG富集分析表明,上调差异表达基因被注释到162条代谢通路中,其中与新陈代谢有关11个、与免疫相关的9个,这些结果表明,菌酶协同发酵饲料的添加对肉鸡的氨基酸代谢、次级产物代谢及免疫功能产生了重要的影响。试验组在脂肪细胞因子信号通路、嘧啶代谢、氨基酸的生物合成、嘌呤代谢等代谢通路上调差异表达,有利于肉鸡氨基酸、胆固醇、牛磺酸和不饱和脂肪酸等风味物质的形成。试验组添加菌酶协同发酵饲料后可能激活肉鸡体内产生吞噬体[22],进而通过细胞因子-细胞因子受体相互作用、C型凝集素受体信号通路和细胞黏附分子信号通路进行信号传递[23],刺激机体产生细胞因子[24],并通过增强甲型流感、单纯疱疹病毒1型感染等病毒代谢通路免疫反应最终起到综合免疫作用。
鸡的肠道内多种微生物共生在宿主体内形成了一个复杂而动态的微生物群落,对宿主健康和生产性能具有重要的生理学意义,主要表现在免疫屏障、免疫系统发育调节、宿主营养吸收和解毒方面的作用[25]。研究表明,畜禽饲养中添加益生菌发酵制剂,可以起到增加肠道有益菌数量、维持肠道免疫稳态、增强抵抗力、降低病原菌数量、改善其生长性能的功效[26-27]。本研究肠道各部位菌群的组成分析表明,肉鸡肠道每1个区段都有自己独特的菌群组成,从而发挥其不同的代谢功能,从菌群数量和种类上看,盲肠>直肠>十二指肠>回肠>空肠,其中十二指肠、回肠、空肠的菌群组成较为一致,但与盲肠、直肠的菌群组成差异较大。盲肠是肉鸡中菌群数量和种类最高的肠道区段,而十二指肠由于pH低,加上胰腺和胆分泌物的抑制和稀释作用,其菌群组成数量最少[28]。肉鸡肠道各区段菌群组成从门水平上看,相对丰度最多的2个门是厚壁菌门和拟杆菌门,研究认为两者丰度之比(F/B)是评价畜禽肠道菌群组成的重要指标,F/B增加,有利于提高厚壁菌门细菌获取能量的能力,从而促进畜禽生长[29]。沙门氏菌、大肠杆菌、霍乱弧菌等变形菌门细菌在肉鸡肠道内定植会导致肠道感染引发肠道及肠道外疾病,是影响畜禽肠道健康的重要因素[30]。从属水平上看,乳杆菌属和乳球菌属是肉鸡肠道主要优势益生菌,其在肉鸡肠道定植,产生挥发性脂肪酸和抑菌因子,降低肠道pH,限制了病原菌增殖,具有预防感染和腹泻的功效[31]。克里斯滕森菌科R-7群、另支菌属能够调节脂质代谢、调节炎症,对某些疾病有保护作用,对调节肠道健康、促进畜禽生长起到了一定的作用[32]。拟杆菌属、肠球菌属、弯曲杆菌属、罕见小球菌属、大肠杆菌-志贺菌群、葡萄球菌属、幽门螺杆菌属、厌氧螺菌属和密螺菌属都是畜禽肠道中常见的病原菌属,如弯曲杆菌属为空肠中的主要研究对象,是“人畜共患病”的食源性病原菌之一,常引起血液炎症,导致畜禽痉挛、疼痛和腹泻[33];葡萄球菌属易引起肉鸡坏死性肠炎[34];密螺菌属细菌易引起的猪血痢[35]等。本研究证实,添加益生菌发酵饲料,试验组肠道各区段微生物菌群的相对丰度上升,但菌群的多样性下降,推测原因可能是乳酸菌等益生菌的繁殖降低了肠道pH,抑制了一些肠道菌和病原菌的生长,增强了畜禽的免疫功能[36],所得结果与转录组测序结论一致。试验组各肠道不同区段的厚壁菌门相对丰度显著增加,拟杆菌门相对丰度降低,乳杆菌属、乳球菌属、克里斯滕森菌科R-7群、另支菌属相对丰度增加,拟杆菌属、肠球菌属、弯曲杆菌属等肉鸡肠道常见病原菌属相对丰度减少,有利于改善肉鸡肠道健康,保持菌群稳态,提高免疫功能,促进氨基酸、牛磺酸、不饱和脂肪酸等代谢物的形成,最终降低畜禽肠道疾病的发病率,促进肉鸡生长,提高肉鸡肌肉风味品质的含量。
4 结论饲粮中添加5.0%的菌酶协同发酵饲料可以提高肉鸡胸肌、腿肌的总氨基酸、风味氨基酸、肌苷酸、不饱和脂肪酸等风味物质的含量,在转录组水平上加强氨基酸、次级产物及免疫功能等代谢通路基因的上调差异表达,改善肠道菌群结构、保持菌群稳态,对病原菌免疫、调节肉鸡肠道健康起到了一定的作用。
| [1] |
徐伟佳, 王智伟, 袁凯鑫, 等. 菌酶协同发酵饲料研究进展[J]. 家畜生态学报, 2020, 41(11): 84-86. XU W J, WANG Z W, YUAN K X, et al. Advances of microbial coupling with enzyme fermented feed[J]. Journal of Domestic Animal Ecology, 2020, 41(11): 84-86 (in Chinese). DOI:10.3969/j.issn.1673-1182.2020.11.017 |
| [2] |
NARASIMHA J, NAGALAKSHMI D, REDDY Y R, et al. Effect of supplementing non starch polysaccharide degrading enzymes to corn soybean meal based diets varying in energy on performance, egg quality, nutrient utilization and gut health in layers[J]. Indian Journal of Poultry Science, 2016, 51(1): 29-35. DOI:10.5958/0974-8180.2016.00006.4 |
| [3] |
DE KEYSER K, KUTERNA L, KACZMAREK S, et al. High dosing NSP enzymes for total protein and digestible amino acid reformulation in a wheat/corn/soybean meal diet in broilers[J]. Journal of Applied Poultry Research, 2016, 25(2): 239-246. DOI:10.3382/japr/pfw006 |
| [4] |
李爽, 王海燕, 刘琼, 等. 菌酶协同发酵饲料的特点及其在畜牧养殖上的应用[J]. 中国饲料, 2020(21): 21-24, 28. LI S, WANG H Y, LIU Q, et al. Characteristics of bacteria-enzyme co-fermented feed and its application in animal husbandry[J]. China Feed, 2020(21): 21-24, 28 (in Chinese). DOI:10.15906/j.cnki.cn11-2975/s.20202104 |
| [5] |
PETRACCI M, CAVANI C. Muscle growth and poultry meat quality issues[J]. Nutrients, 2012, 4(1): 1-12. |
| [6] |
闫俊书, 刘培峰, 施振旦. 家禽肌肉肌苷酸含量影响因素及其相关基因的研究进展[J]. 中国家禽, 2017, 39(10): 41-46. YAN J S, LIU P F, SHI Z D. Research progress on influence factors and related genes of inosine monophosphate content in poultry muscle[J]. China Poultry, 2017, 39(10): 41-46 (in Chinese). DOI:10.16372/j.issn.1004-6364.2017.10.010 |
| [7] |
叶成智, 宦海琳, 周维仁, 等. 发酵饲料对肉鸡生长性能、肌肉理化特性及风味的影响[J]. 饲料工业, 2021, 42(11): 7-13. YE C Z, HUAN H L, ZHOU W R, et al. Effects of fermented feed on growth performance, muscle physicochemical properties and flavor of broilers[J]. Feed Industry, 2021, 42(11): 7-13 (in Chinese). DOI:10.13302/j.cnki.fi.2021.11.002 |
| [8] |
AZAMAN S N A, SATHARASINGHE D A, TAN S W, et al. Identification and analysis of microRNAs in Chlorella sorokiniana using high-throughput sequencing[J]. Genes, 2020, 11(10): 1131. DOI:10.3390/genes11101131 |
| [9] |
WU X Y, ZHANG H H, CHEN J, et al. Comparison of the fecal microbiota of dholes high-throughput Illumina sequencing of the V3-V4 region of the 16S rRNA gene[J]. Applied Microbiology and Biotechnology, 2016, 100(8): 3577-3586. DOI:10.1007/s00253-015-7257-y |
| [10] |
WANG L K, FENG Z X, WANG X, et al. DEGseq: an R package for identifying differentially expressed genes from RNA-Seq data[J]. Bioinformatics, 2010, 26(1): 136-138. DOI:10.1093/bioinformatics/btp612 |
| [11] |
WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics[J]. Nature Reviews Genetics, 2009, 10(1): 57-63. DOI:10.1038/nrg2484 |
| [12] |
YANG X H, CHEN L, YANG Y, et al. Transcriptome analysis reveals that exogenous ethylene activates immune and defense responses in a high late blight resistant potato genotype[J]. Scientific Reports, 2020, 10(1): 21294. DOI:10.1038/s41598-020-78027-5 |
| [13] |
WANG Z R, SONG M Y, WANG Z, et al. Metabolome and transcriptome analysis of flavor components and flavonoid biosynthesis in fig female flower tissues (Ficus carica L.) after bagging[J]. BMC Plant Biology, 2021, 21(1): 396. DOI:10.1186/s12870-021-03169-1 |
| [14] |
EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [15] |
MILLER G E, ENGEN P A, GILLEVET P M, et al. Lower neighborhood socioeconomic status associated with reduced diversity of the colonic microbiota in healthy adults[J]. PLoS One, 2016, 11(2): e0148952. DOI:10.1371/journal.pone.0148952 |
| [16] |
JIA Y L, LIAO Z, CHEW H, et al. Effect of Pennisetum giganteum z.x.lin mixed nitrogen-fixing bacterial fertilizer on the growth, quality, soil fertility and bacterial community of pakchoi (Brassica chinensis L.)[J]. PLoS One, 2020, 15(2): e0228709. DOI:10.1371/journal.pone.0228709 |
| [17] |
LI K, DAN Z, GESANG L B, et al. Comparative analysis of gut microbiota of native Tibetan and Han populations living at different altitudes[J]. PLoS One, 2016, 11(5): e0155863. DOI:10.1371/journal.pone.0155863 |
| [18] |
孙智媛, 梅力文, 黄兴国, 等. 菌酶协同发酵饲料及其在动物生产中应用的研究进展[J]. 中国畜牧杂志, 2021, 57(8): 42-47. SUN Z Y, MEI L W, HUANG X G, et al. Research progress on fermented feed by microbial cooperating with enzyme and its application to animal production[J]. Chinese Journal of Animal Science, 2021, 57(8): 42-47 (in Chinese). DOI:10.19556/j.0258-7033.20200828-01 |
| [19] |
LIN B S, YAN J B, ZHONG Z L, et al. A study on the preparation of microbial and nonstarch polysaccharide enzyme synergistic fermented maize cob feed and its feeding efficiency in finishing pigs[J]. BioMed Research International, 2020, 2020: 8839148. |
| [20] |
王红明, 丁雪婧, 陈俭, 等. 饲料中添加甘露寡糖对珍珠龙胆石斑鱼生长性能、血清免疫指标、转录组及肠道菌群的影响[J]. 动物营养学报, 2021, 33(12): 6982-6998. WANG H M, DING X J, CHEN J, et al. Effects of dietary mannan-oligosaccharides on growth performance, serum immune indices, transcriptome and intestinal microflora of Epinephelus fuscoguttatus ♀×Epinephelus lanceolatus ♂[J]. Chinese Journal of Animal Nutrition, 2021, 33(12): 6982-6998 (in Chinese). |
| [21] |
CHEN L, GAO L X, HUANG Q H, et al. Viscous and fermentable nonstarch polysaccharides affect intestinal nutrient and energy flow and hindgut fermentation in growing pigs[J]. Journal of Animal Science, 2017, 95(11): 5054-5063. DOI:10.2527/jas2017.1662 |
| [22] |
DEAN P, HEUNIS T, HÄRTLOVA A, et al. Regulation of phagosome functions by post-translational modifications: a new paradigm[J]. Current Opinion in Chemical Biology, 2019, 48: 73-80. DOI:10.1016/j.cbpa.2018.11.001 |
| [23] |
HARJUNPÄÄ H, LLORT ASENS M, GUENTHER C, et al. Cell adhesion molecules and their roles and regulation in the immune and tumor microenvironment[J]. Frontiers in Immunology, 2019, 10: 1078. DOI:10.3389/fimmu.2019.01078 |
| [24] |
PAN W Y, WANG Q X, CHEN Q M. The cytokine network involved in the host immune response to periodontitis[J]. International Journal of Oral Science, 2019, 11(3): 30. DOI:10.1038/s41368-019-0064-z |
| [25] |
周雪雁, 李琼毅, 丁功涛, 等. 鸡肠道微生物菌群的建立发育、分布和生理学意义[J]. 微生物学报, 2020, 60(4): 641-652. ZHOU X Y, LI Q Y, DING G T, et al. Establishment, distribution and physiological significance of the intestinal microbiota in chicken[J]. Acta Microbiologica Sinica, 2020, 60(4): 641-652 (in Chinese). DOI:10.13343/j.cnki.wsxb.20190376 |
| [26] |
TAKⅡSHI T, FENERO C I M, CÂMARA N O S. Intestinal barrier and gut microbiota: shaping our immune responses throughout life[J]. Tissue Barriers, 2017, 5(4): e1373208. DOI:10.1080/21688370.2017.1373208 |
| [27] |
LI Z, WANG W W, LIU D, et al. Effects of Lactobacillus acidophilus on gut microbiota composition in broilers challenged with Clostridium perfringens[J]. PLoS One, 2017, 12(11): e0188634. DOI:10.1371/journal.pone.0188634 |
| [28] |
张利环, 贾浩, 张若男, 等. 复合益生菌对肉鸡肠道微生物区系的影响[J]. 中国畜牧兽医, 2022, 49(2): 488-500. ZHANG L H, JIA H, ZHANG R N, et al. Effects of compound probiotics on the intestinal microflora of broilers[J]. China Animal Husbandry & Veterinary Medicine, 2022, 49(2): 488-500 (in Chinese). DOI:10.16431/j.cnki.1671-7236.2022.02.010 |
| [29] |
SINGH K M, SHAH T, DESHPANDE S, et al. High through put 16S rRNA gene-based pyrosequencing analysis of the fecal microbiota of high FCR and low FCR broiler growers[J]. Molecular Biology Reports, 2012, 39(12): 10595-10602. DOI:10.1007/s11033-012-1947-7 |
| [30] |
CHANG C H, TENG P Y, LEE T T, et al. Effects of multi-strain probiotic supplementation on intestinal microbiota, tight junctions, and inflammation in young broiler chickens challenged with Salmonella enterica subsp. enterica[J]. Asian-Australasian Journal of Animal Sciences, 2020, 33(11): 1797-1808. DOI:10.5713/ajas.19.0427 |
| [31] |
刘凤美, 张磊, 黄彬. 日粮添加益生菌对肉鸡生产性能、免疫功能和肠道菌群的影响[J]. 中国饲料, 2018(24): 39-43. LIU F M, ZHANG L, HUANG B. Effects of probiotics on production performance, immunologic function and gut bacteria of broiler chickens[J]. China Feed, 2018(24): 39-43 (in Chinese). DOI:10.15906/j.cnki.cn11-2975/s.20182410 |
| [32] |
BLOEMENDAAL M, SZOPINSKA-TOKOV J, BELZER C, et al. Probiotics-induced changes in gut microbial composition and its effects on cognitive performance after stress: exploratory analyses[J]. Translational Psychiatry, 2021, 11(1): 300. DOI:10.1038/s41398-021-01404-9 |
| [33] |
孙桂霞, 徐世文. 鸡源空肠弯曲菌调查与耐药性分析[J]. 东北农业大学学报, 2015, 46(9): 64-68. SUN G X, XU S W. Investigation and resistance analysis of Campylobacter jejuni in broiler[J]. Journal of Northeast Agricultural University, 2015, 46(9): 64-68 (in Chinese). DOI:10.3969/j.issn.1005-9369.2015.09.010 |
| [34] |
LIN E R, CHENG Y H, HSIAO F S H, et al. Optimization of solid-state fermentation conditions of Bacillus licheniformis and its effects on Clostridium perfringens-induced necrotic enteritis in broilers[J]. Revista Brasileira de Zootecnia, 2019, e20170298. |
| [35] |
任忠磊. 猪出血性痢疾的诊断与治疗[J]. 广东蚕业, 2020, 54(8): 46-47. REN Z L. Diagnosis and treatment of swine hemorrhagic dysentery[J]. Guangdong Canye, 2020, 54(8): 46-47 (in Chinese). DOI:10.3969/j.issn.2095-1205.2020.08.22 |
| [36] |
BORDA-MOLINA D, SEIFERT J, CAMARINHA-SILVA A. Current perspectives of the chicken gastrointestinal tract and its microbiome[J]. Computational and Structural Biotechnology Journal, 2018, 16: 131-139. DOI:10.1016/j.csbj.2018.03.002 |




