线粒体作为细胞代谢的中枢,是细胞能量供应、新陈代谢以及凋亡的重要场所,通过氧化磷酸化产生ATP,同时也是脂肪酸、激素和氨基酸生物合成的关键中间体,也参与细胞凋亡和免疫等机体功能[1]。因此,线粒体功能稳定对于细胞存活和机体的正常生理功能具有重要意义。线粒体是细胞在各种压力下的第1道防线。功能失调的线粒体可以通过一系列复杂的适应性反应修复,如线粒体生物发生、线粒体裂变/融合、线粒体未折叠蛋白反应(mitochondrial unfolded protein reaction,UPRmt)和线粒体自噬[2]。本文综述了线粒体功能损伤的调控机制(主要阐述线粒体自噬和UPRmt)的研究进展和线粒体功能研究方法,旨在为进一步促进以线粒体为靶标的营养调控剂在动物生产上的应用提供参考。
1 线粒体功能损伤调控机制生物体已经进化出多种机制来识别和感知线粒体的功能障碍,用于恢复可抢救的细胞器,并降解无法修复的细胞器,用以维持健康的线粒体网络[3]。线粒体应激反应途径主要分为3种:1)介导受损线粒体的识别和选择性降解的线粒体自噬通路;2)转录翻译衰减,线粒体功能障碍后真核翻译起始因子2α激酶4(EIF2AK4)/丝氨酸/苏氨酸蛋白激酶2(GCN2)被激活,通过磷酸化真核类起始因子2(eukaryotic initiation factor 2,eIF2α)介导翻译衰减,这有助于减少蛋白质流入线粒体[4];3)损伤初期能限制有缺陷的线粒体损伤并促进回收可用的线粒体恢复的调控手段——UPRmt[5-6]。
1.1 线粒体自噬线粒体自噬是维持线粒体质量所必需的关键机制之一,是调控线粒体数量和质量的核心机制。缺陷线粒体可产生过量活性氧(reactive oxygen species,ROS),通过逆转三磷酸腺苷合酶(adenosine triphosphate synthase,ATPase)活性消耗ATP,损害线粒体代谢功能,触发细胞凋亡[7-8]。因此,线粒体自噬旨在迅速清除这些功能失调的线粒体,以保护细胞完整性。线粒体自噬是一种选择性自噬过程,将多余或者损伤的线粒体运送到溶酶体进行降解。含有损伤线粒体的自噬小体可直接或通过内体融合与溶酶体融合,形成中间的双裂体,形成自溶酶体,降解和回收线粒体[9]。调控线粒体自噬的通路主要包括泛素化依赖的磷酸酯酶与张力蛋白同源物诱导激酶1(PTEN-induced kinase 1,PINK1)/帕金蛋白(Parkin)通路以及受体介导线粒体自噬[B淋巴细胞瘤-2(Bcl-2)/腺病毒E1B19kDa互作蛋白3(E1B19kDa-interacting protein,BINP3)、NIP3样蛋白X(NIP3-like protein X,NIX)和FUN14含结构域蛋白1(FUN14 domain-containing protein 1,FUNDC1)][10]。
1.1.1 泛素化依赖的线粒体自噬通路泛素依赖性线粒体自噬主要指PINK1/Parkin途径。该途径需要线粒体相关的丝氨酸/苏氨酸激酶PINK1和Parkin。PINK1-Parkin激活的线粒体自噬通路是目前机制研究最为透彻的通路。PINK1是一种丝氨酸/苏氨酸蛋白酶,位于Parkin的上游,是线粒体的运营及传感器,其功能依赖于线粒体膜电位。PINK1与线粒体自噬的选择性启动密切相关,通过引起线粒体分裂和降解来调控线粒体质量和数量[11]。线粒体损伤后膜电位快速降低,PINK1蛋白在损伤的线粒体外膜上积聚,招募Parkin对损伤线粒体进行泛素化修饰,受体蛋白p62等识别线粒体泛素化信号,并通过与自噬微管相关蛋白1轻链3(autophagy microtubule-associated protein-1 light chain 3,LC3)[12]启动自噬形成。Parkin可泛素化线粒体外膜上的多种蛋白,目前发现Parkin的下游蛋白有线粒体融合蛋白1/2(mitochondrial fusion protein 1/2,Mfn1/2)、线粒体Rho鸟苷三磷酸酶(mitochondrial Rho GTPase,Miro)、线粒体电压依赖性阴离子通道1(mitochondrial voltage dependent anionic channel 1,VADC1)和线粒体移位酶复合体(mitochondrial translocation enzyme complex,包括TOM70、TOM40和TOM20)[13-14]。PINK1-Parkin通路能通过靶向Mfn和Miro进行蛋白酶体降解来调节线粒体动力学。
其他E3-泛素连接酶也参与线粒体表面蛋白的泛素化,如线粒体锚定蛋白连接酶1(mitochondrial-anchored protein ligase 1,MUL1)、Ariadne-1同源体(Ariadne-1 homolog,ARIH1)、7个缺席同源体1(seven in absentia homologs 1,SIAH1)、Smad特异性E3泛素蛋白连接酶1(Smad specific E3 ubiquitin protein ligase 1,SMURF1)等。这些泛素连接酶可单独作用,也可以与Parkin相互作用,通过泛素结合域和LC3相互作用区域(LIR)基序将泛素化的蛋白与形成自噬小体连接起来[15]。p62也被发现可作用于泛素化级联的上游,介导基底肝细胞线粒体自噬,而不依赖于Parkin。在DRP1敲除的肝细胞中,p62在线粒体中积累,通过与Kelch样ECH相关蛋白1(Kelch-like ECH-associated protein 1,Keap1)相互作用和招募E3连接酶环状盒1(ring-box 1,RBX1)促进线粒体泛素化[16]。去泛素化酶(deubiquitinating enzymes,DUBs)如泛素蛋白特异性蛋白酶(ubiquitin-specific protease,USP)30、USP35和USP15能抑制线粒体自噬。研究表明,USP30是唯一与线粒体表面构成相关的DUB,它在线粒体表面去泛素化Parkin底物以抑制Parkin依赖的线粒体自噬[17]。
1.1.2 非泛素化依赖的线粒体自噬通路BINP3、NIX和FUNDC1是线粒体外膜定位蛋白。BINP3和NIX是LC3互作位点(LC3-interacting region,LIR)的启动器,LIR位点朝向胞质侧,具有调节线粒体自噬的作用[18]。这些蛋白介导线粒体自噬的主要模式被认为是介导线粒体与新生自噬小体的直接相互作用。这一过程在2个不同的水平上被调控:1)蛋白丰度。BNIP3和NIX受到上游线粒体自噬信号通路的转录控制。线粒体自噬触发这些受体的表达,并提高它们在线粒体外膜(outer mitochondrial membrane,OMM)中的含量。目前还不清楚在这些情况下,线粒体靶向受体的选择性是如何发生的[19]。2)翻译后修饰。线粒体受体功能主要通过磷酸化调节,促进或增强受体与LC3/LC3/GABAA型受体相关蛋白(GABA type A receptor-associated protein,GABARAP)相互作用以驱动线粒体自噬的能力。例如,自噬相关基因(autophagy-related gene,ATG)32在LIR基序附近被磷酸化,可能是被CK2磷酸化,以促进与适配器蛋白ATG11的相互作用[20-21]。同样,NIX也被一种未知激酶磷酸化,以加强与ABARAP蛋白的相互作用[22]。相反,在正常生理条件下,LIR基序的酪蛋白激酶2(casein kinase 2,CK2)/Src磷酸化可抑制FUNDC1。在这种情况下,PGAM5磷酸酶的活性促进了线粒体自噬活性,而ULK1磷酸化加强了FUNDC1与ATG8的相互作用。FUNDC1也可以被泛素化和蛋白酶体降解,以限制线粒体自噬反应[23]。虽然磷酸化目前是调节的主要机制,其他仍然没有特征的翻译后修饰可能是重要的微调受体的活性[24]。总的来说,线粒体自噬受体的上游调控和标记线粒体的内在选择性仍然知之甚少。
有研究表明,某些线粒体蛋白有不同的基础自噬转换率,线粒体蛋白是如何在线粒体自噬消除的网络中被选择性地分类和调节的,目前尚不十分清楚,同时也缺乏对线粒体自噬如何在线粒体生物发生、功能和最终细胞反应与功能的动力学在更大范围内协调的理解。线粒体网络稳态也可能涉及蛋白酶体依赖的线粒体蛋白、线粒体蛋白酶和伴侣的降解或线粒体未折叠蛋白反应。然而,线粒体自噬和所有其他线粒体质量控制途径之间的分子和生理相互作用仍然知之甚少。
1.2 UPRmtUPRmt是在应激条件下响应线粒体未折叠或错误折叠的蛋白质,诱导核基因编码的线粒体分子伴侣热休克蛋白(heat shock proteins,HSP)60、HSP70及蛋白酶等的表达量,促进线粒体蛋白稳态的信号传导过程[5-6]。应激状态下激活的UPRmt通过上调基因转录或促进泛素蛋白酶体系统降解蛋白质来优化线粒体蛋白的进出口质量控制。如果UPRmt不能完全修复线粒体损伤,则诱导线粒体裂变将受损区域从健康线粒体网络中分离出来,然后通过线粒体吞噬作用清除结构受损的线粒体[25]。可以认为,UPRmt和线粒体自噬是不同的线粒体修复途径。与UPRmt相比,线粒体自噬减少了线粒体数量,这种改变有时与ATP缺乏和细胞死亡有关。过度诱导线粒体自噬是致命的,由于多数线粒体被降解,ATP产生被抑制,导致细胞死亡。与线粒体自噬不同,UPRmt动态地控制线粒体蛋白的输入或输出,并微调线粒体行为。
UPRmt的调节信号研究的最清楚的是在线虫中。通过遗传方法发现了许多UPRmt调控因子,包括线粒体基质蛋白酶酪蛋白水解肽酶P(caseinolytic peptidase P,ClpP)、泛素样蛋白Ub-5、转录因子DVE-1、线粒体ABC转运蛋白Haf-1和应激激活转录因子-1(stress-activated transcription factor-1,ATFS-1)。ClpP通过蛋白质水解方式降解折叠不当的线粒体蛋白,随后通过HAF-1释放线粒体产生的多肽,从而激活UPRmt。在生理条件下,线粒体靶向序列引导ATFS-1定位到线粒体,而后被蛋白酶Lon降解[5]。通常,ATFS-1被输入线粒体并被降解。然而,在线粒体应激期间,我们发现输入效率降低,允许一定比例的ATFS-1在细胞质中积累并运输到细胞核[26-27]。ATFS-1调节多种转录反应以恢复线粒体功能,包括诱导线粒体蛋白酶和分子伴侣基因(HSP60、HSP70等基因)及ROS解毒基因(如谷胱甘肽硫转移酶、超氧化物歧化酶或过氧化氢酶基因等)等[5]。在哺乳动物中,UPRmt受碱性亮氨酸拉链(basic leucine zipper,bZIP)转录因子ATF4、ATF5和CCAAT/增强子绑定的同源蛋白质(CCAAT/enhancer-binding homologous protein,CHOP)的调控。受ATFS-1发现的启发,Fiorese等[28]也发现,ATF5是哺乳动物UPRmt应答的调节因子。线粒体输入效率似乎以一种类似于ATFS-1的方式调节ATF5,ATF5也包含线粒体定位序列。正常生理状态下,ATF5定位于线粒体。而当线粒体受到应激时,ATF5通过调节UPRmt保护线粒体功能。与ATFS-1类似,ATF5在转录上调节线粒体保护基因的表达,这是线粒体应激期间细胞生长所必需的[29]。因此,在应激状态下ATF5/UPRmt会激活由核DNA编码的线粒体伴侣蛋白和蛋白酶的转录激活程序,同时通过诱导和激活泛素化-蛋白酶体系统以最大限度地清除损伤的线粒体蛋白,维持细胞蛋白稳态[30]。氧化应激可通过UPRmt缓解线粒体蛋白损伤改善线粒体功能。百草枯产生的过量ROS增加受损蛋白质的含量,最终激活UPRmt,UPRmt下调线粒体伴侣基因HSP6和HSP60,转录因子SKN-1和低氧相关因子1(hypoxia-associated factor 1,HAF1)[31-32]。
目前的研究表明,哺乳动物由4种不同的被线粒体蛋白错误折叠/聚集激活的UPRmt轴[33](图 1):1)典型的UPRmt。导致CHOP、ATF4和ATF5从细胞质转移到细胞核,导致诱导伴侣素和蛋白酶基因表达,以增加线粒体内部的折叠能力[34-36];2)UPRmt-组蛋白去乙酰化酶(sirtuin,SIRT)轴。SIRT1/SIRT3作为UPRmt-Sirtuin轴的一部分被激活,导致叉头样转录因子3A(forkhead box O3A, FOXO3A)去乙酰化并重新定位到细胞核,诱导超氧化物歧化酶2(superoxide dismutase 2,SOD2)和过氧化氢酶等抗氧化酶表达缓解氧化应激[37-38];3)UPRIMS/雌激素受体α(estrogen receptor alpha,ERα)轴。被蛋白质在膜间空间的错误折叠激活,该轴通过蛋白激酶B(protein kinase B,PKB)-AKT和ROS依赖的ERα磷酸化作用,导致核转录因子1(nuclear transcription factor 1,NRF1)的诱导,增加蛋白酶水平和蛋白酶体活性,调节呼吸水平,以增加蛋白质质量控制能力[39];4)UPRmt翻译轴。这是一种局部反应,基质中的蛋白质展开导致RNA前加工机制的组分快速降解,并关闭线粒体翻译,以降低线粒体折叠负荷[33]。
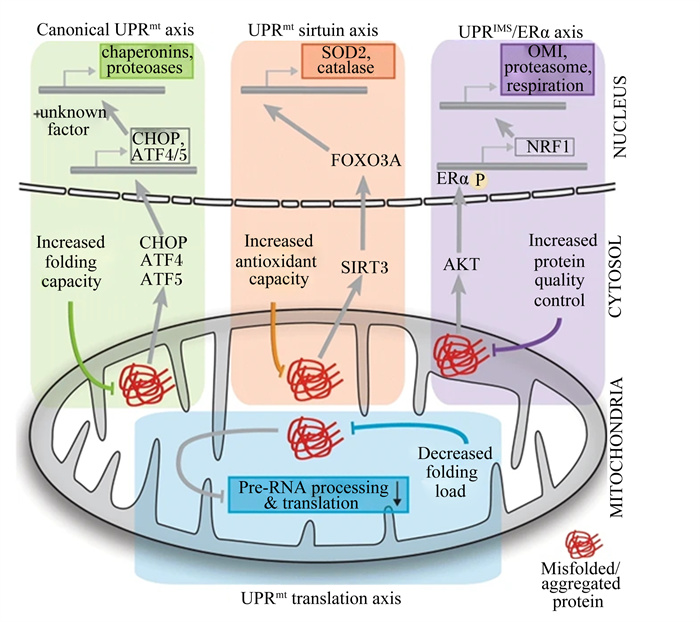
|
NUCLEUS:细胞核;CYTOSOL: 细胞质;MITOCHONDRIA: 线粒体;UPRmt:线粒体未折叠蛋白反应mitochondrial unfolded protein reaction;canonical UPRmt:典型的UPRmt; UPRmt sirtuin axis:UPRmt-组蛋白去乙酰化酶轴UPRmt-histone deacetylase axis;IMS:膜间隙intermembrane space;UPRIMS/ERα axis:UPRIMS/雌激素受体α轴UPRIMS/estrogen receptor α axis;UPRmt translation axis:UPRmt翻译轴;chaperonins:伴侣蛋白;proteoases:蛋白酶;unknown factor:未知因子;CHOP:CCAAT/增强子绑定的同源蛋白质CCAAT/enhancer-binding homologous protein;ATF: 转录激活因子4 activating transcription factor 4;Increased folding capacity:提高折叠能力;FOXO3:叉头样转录因子3A forkhead box O3;SIRT3:去乙酰化酶3 sirtuin 3;SOD2:超氧化物歧化酶2 superoxide dismutase 2;catalase:过氧化氢酶;Increased antioxidant capacity:增加抗氧化能力;AKT:蛋白激酶B protein kinase B;ERα:雌激素受体α estrogen receptor alpha;NRF1:核转录因子1 nuclear transcription factor 1;OMI:线粒体膜间隙酶mitochondrial intermembrane enzyme;proteasome:蛋白酶体;respiration:呼吸代谢;Increased protein quality control:提高蛋白质量控制;Pre-RNA processing & translation:前体-RNA处理 & 转录翻译;Decreased folding load:减少折叠负载。 图 1 4种哺乳动物UPRmt轴 Fig. 1 Four mammals UPRmt axis[33] |
研究极端线粒体损伤是通常使用化学物质,如质子载体-羰基氰化物间氯苯基腙(phenylhydrazones carbonyl cyanide m-chlorophenyl hydrazone,CCCP)、菜籽油氰化物4-(三氟甲氧基)苯腙[carbonyl cyanide-p-(trifluoromethoxy)phenylhydrazone,FCCP]和2, 4-二硝基酚,或选择性电子传递链抑制剂(如鱼藤酮、抗霉素A或寡霉素A),它们会损害线粒体呼吸或使线粒体去极化[40-41],用来研究线粒体损伤和激活线粒体自噬的具体机制,并探索其与动物生理功能损伤的相关性。引起帕金森表型的毒素如百草枯或6-羟多巴胺,也可以去极化并损伤线粒体[40]。迄今为止发现的绝大多数线粒体自噬诱导剂本质上是通过抑制线粒体呼吸来启动线粒体自噬反应的相关毒素。
2.1.2 外加压力构建损伤模型压力增加细胞水平的能量需求,并激活系统的线粒体和内分泌过程来维持这种增加的需求。线粒体生物发生需要将核编码的线粒体蛋白(NEMP)递送至线粒体,并由内源性(例如ROS、氮氧化物、一氧化碳和硫化氢)和外部信号(例如雌激素)诱导,这些信号通过核呼吸因子1(nuclear respiratory factor 1,NRF1)增加过氧化物酶体增殖物激活受体γ共激活因子-1α (PGC-1α)和雌激素受体。因此,能量限制、运动、一氧化氮、一氧化碳、硫化氢和ROS等都有影响线粒体生物发生的功能,也可以用作模型[42]。线粒体功能与氧的可利用性密切相关,特别是氧化磷酸化代谢。在缺氧条件下,细胞激活缺氧诱导因子1(hypoxia-induced factor 1,HIF1)信号通过调节基因表达来驱动应激和代谢适应程序。缺氧会导致线粒体呼吸效率低下、线粒体应激和能量危机。因此,HIF1信号通路控制一个代谢开关,上调糖酵解基因,减弱线粒体呼吸,触发线粒体自噬[19]。温度应激也被证明会影响线粒体自噬。小鼠受到慢性冷应激刺激β-3肾上腺素能信号通路,依赖FUNDC1通过翻译后修饰和PGC-1α基因表达的增加激活PGC-1α,诱导形成富含线粒体的褐色脂肪细胞[43]。最近也有研究表明,低温应激在恢复到正常生理温度后也会触发人成纤维细胞的线粒体自噬[44]。
2.2 线粒体功能研究方法评估线粒体功能的方法主要包括线粒体活力及膜电位检测、线粒体生化分析、线粒体能量学功能(呼吸代谢检测和呼吸链复合体活性检测)以及线粒体抗氧化功能检测等(图 2)。评估线粒体代谢功能主要通过代谢组学利用质谱等方法对三羧酸循环(tricarboxylic acid cycle,TCA)、糖酵解以及谷氨酰胺分解等通路的代谢产物进行检测分析,并通过酶学方法对这些通路的代谢酶活性进行分析。评估细胞自噬及线粒体自噬主要包括3种:1)利用电子显微镜观察线粒体自噬体结构或者利用荧光染色LC3II等蛋白因子或线粒体特异性荧光探针TMRM,Mito Tracker和溶酶体特异性荧光探针LysoTracker进行成像分析追踪线粒体自噬的动态过程[45-46];2)流式细胞术检测线粒体膜电位JC-1等强度变化,线粒体总量以及蛋白印迹分析线粒体自噬相关蛋白表达量的变化;3)利用LC3和Mitodsred等质粒转染构建可视化模型观察[47]。线粒体质量分析主要是线粒体蛋白及mtDNA含量分析,同时检测线粒体蛋白和DNA的损伤。评估线粒体能量学功能主要是通过Seahorse XF对线粒体呼吸代谢进行定量分析,并结合线粒体呼吸链(ETC)中蛋白活力及含量等进行具体分析(图 2)[48]。ATP酶和ATP含量的变化也可检测线粒体能量代谢变化。线粒体氧化反应检测主要包括测定ROS含量、线粒体内丙二醛以及抗氧化酶如超氧化物歧化酶、过氧化氢酶和谷胱甘肽过氧化物酶等活性[49]。
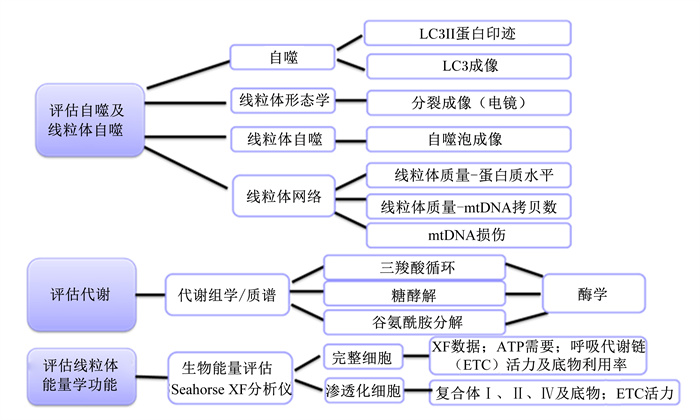
|
ETC:呼吸代谢链respiratory metabolic chain;ATP:三磷酸腺苷adenosine triphosphate;LC3:自噬微管相关蛋白1轻链3 autophagy microtubule-associated protein-1 light chain 3。 图 2 线粒体功能研究方法 Fig. 2 Research methods for mitochondrial function |
针对机体内模型,研究开发了mito-Timer、mt-Keima和mito-QC小鼠模型,为研究生理应激如何在体内影响哺乳动物的线粒体自噬开辟了一条新的途径[50]。例如,经过力竭运动的小鼠骨骼肌和心肌线粒体自噬增加。Sun等[51]利用mt-Keima报告对几种应激对肝脏线粒体噬的影响发现,在遭受长时间缺氧或损伤的线粒体聚合酶G的应激小鼠中,线粒体自噬增加。
3 小结和展望线粒体作为细胞中损伤的第1道防线以及代谢中枢,维持线粒体蛋白平衡和功能稳定对缓解各种应激,促进细胞正常运转十分重要。近年来,通过刺激线粒体能量的产生、促进线粒体代谢以及减轻氧化应激等改善线粒体功能的手段,研究对繁殖期母猪及仔猪进行营养干预提高其生产性能。靶向线粒体调控的营养物质,既可以通过积极参与特定的线粒体生物化学途径直接发挥其功能,也可以通过提高编码线粒体蛋白的基因表达来间接发挥其功能。对于线粒体的蛋白平衡和功能调控的研究还需要深入,如何通过营养物质调控线粒体蛋白平衡和功能完整也将逐渐成为缓解畜禽繁殖期机体应激损伤的重点方向。
| [1] |
SPINELLI J B, HAIGIS M C. The multifaceted contributions of mitochondria to cellular metabolism[J]. Nature Cell Biology, 2018, 20(7): 745-754. DOI:10.1038/s41556-018-0124-1 |
| [2] |
PICKLES S, VIGIÉ P, YOULE R J. Mitophagy and quality control mechanisms in mitochondrial maintenance[J]. Current Biology, 2018, 28(4): R170-R185. DOI:10.1016/j.cub.2018.01.004 |
| [3] |
MELBER A, HAYNES C M. UPRmt regulation and output: a stress response mediated by mitochondrial-nuclear communication[J]. Cell Research, 2018, 28(3): 281-295. DOI:10.1038/cr.2018.16 |
| [4] |
BAKER B M, NARGUND A M, SUN T, et al. Protective coupling of mitochondrial function and protein synthesis via the eIF2α kinase GCN-2[J]. PLoS Genetics, 2012, 8(6): e1002760. DOI:10.1371/journal.pgen.1002760 |
| [5] |
NARGUND A M, PELLEGRINO M W, FIORESE C J, et al. Mitochondrial import efficiency of ATFS-1 regulates mitochondrial UPR activation[J]. Science, 2012, 337(6094): 587-590. DOI:10.1126/science.1223560 |
| [6] |
SONG J Y, HERRMANN J M, BECKER T. Quality control of the mitochondrial proteome[J]. Nature Reviews Molecular Cell Biology, 2021, 22(1): 54-70. DOI:10.1038/s41580-020-00300-2 |
| [7] |
PFANNER N, WARSCHEID B, WIEDEMANN N. Mitochondrial proteins: from biogenesis to functional networks[J]. Nature Reviews Molecular Cell Biology, 2019, 20(5): 267-284. DOI:10.1038/s41580-018-0092-0 |
| [8] |
YOULE R J, VAN DER BLIEK A M. Mitochondrial fission, fusion, and stress[J]. Science, 2012, 337(6098): 1062-1065. DOI:10.1126/science.1219855 |
| [9] |
ZACHARI M, GANLEY I G. The mammalian ULK1 complex and autophagy initiation[J]. Essays in Biochemistry, 2017, 61(6): 585-596. DOI:10.1042/EBC20170021 |
| [10] |
MONTAVA-GARRIGA L, GANLEY I G. Outstanding questions in mitophagy: what we do and do not know[J]. Journal of Molecular Biology, 2020, 432(1): 206-230. DOI:10.1016/j.jmb.2019.06.032 |
| [11] |
BARODIA S K, CREED R B, GOLDBERG M S. Parkin and PINK1 functions in oxidative stress and neurodegeneration[J]. Brain Research Bulletin, 2017, 133: 51-59. DOI:10.1016/j.brainresbull.2016.12.004 |
| [12] |
EIYAMA A, OKAMOTO K. PINK1/parkin-mediated mitophagy in mammalian cells[J]. Current Opinion in Cell Biology, 2015, 33: 95-101. DOI:10.1016/j.ceb.2015.01.002 |
| [13] |
RAKOVIC A, GRVNEWALD A, KOTTWITZ J, et al. Mutations in PINK1 and parkin impair ubiquitination of mitofusins in human fibroblasts[J]. PLoS One, 2011, 6(3): e16746. DOI:10.1371/journal.pone.0016746 |
| [14] |
YOSHⅡ S R, KISHI C, ISHIHARA N, et al. Parkin mediates proteasome-dependent protein degradation and rupture of the outer mitochondrial membrane[J]. The Journal of Biological Chemistry, 2011, 286(22): 19630-19640. DOI:10.1074/jbc.M110.209338 |
| [15] |
PALIKARAS K, LIONAKI E, TAVERNARAKIS N. Mechanisms of mitophagy in cellular homeostasis, physiology and pathology[J]. Nature Cell Biology, 2018, 20(9): 1013-1022. DOI:10.1038/s41556-018-0176-2 |
| [16] |
YAMADA T, MURATA D, ADACHI Y, et al. Mitochondrial stasis reveals p62-mediated ubiquitination in parkin-independent mitophagy and mitigates nonalcoholic fatty liver disease[J]. Cell Metabolism, 2018, 28(4): 588-604.e5. DOI:10.1016/j.cmet.2018.06.014 |
| [17] |
BINGOL B, TEA J S, PHU L, et al. The mitochondrial deubiquitinase USP30 opposes parkin-mediated mitophagy[J]. Nature, 2014, 510(7505): 370-375. DOI:10.1038/nature13418 |
| [18] |
SANDOVAL H, THIAGARAJAN P, DASGUPTA S K, et al. Essential role for Nix in autophagic maturation of erythroid cells[J]. Nature, 2008, 454(7201): 232-235. DOI:10.1038/nature07006 |
| [19] |
ZHANG H F, BOSCH-MARCE M, SHIMODA L A, et al. Mitochondrial autophagy is an HIF-1-dependent adaptive metabolic response to hypoxia[J]. The Journal of Biological Chemistry, 2008, 283(16): 10892-10903. DOI:10.1074/jbc.M800102200 |
| [20] |
AOKI Y, KANKI T, HIROTA Y, et al. Phosphorylation of serine 114 on Atg32 mediates mitophagy[J]. Molecular Biology of the Cell, 2011, 22(17): 3206-3217. DOI:10.1091/mbc.e11-02-0145 |
| [21] |
KANKI T, KURIHARA Y, JIN X L, et al. Casein kinase 2 is essential for mitophagy[J]. EMBO Reports, 2013, 14(9): 788-794. DOI:10.1038/embor.2013.114 |
| [22] |
ROGOV V V, SUZUKI H, MARINKOVI M, et al. Phosphorylation of the mitochondrial autophagy receptor Nix enhances its interaction with LC3 proteins[J]. Scientific Reports, 2017, 7(1): 1131. DOI:10.1038/s41598-017-01258-6 |
| [23] |
WU W X, TIAN W L, HU Z, et al. ULK1 translocates to mitochondria and phosphorylates FUNDC1 to regulate mitophagy[J]. EMBO Reports, 2014, 15(5): 566-575. DOI:10.1002/embr.201438501 |
| [24] |
WANG L M, QI H, TANG Y C, et al. Post-translational modifications of key machinery in the control of mitophagy[J]. Trends in Biochemical Sciences, 2020, 45(1): 58-75. DOI:10.1016/j.tibs.2019.08.002 |
| [25] |
PELLEGRINO M W, NARGUND A M, HAYNES C M. Signaling the mitochondrial unfolded protein response[J]. Biochimica et Biophysica Acta (BBA): Molecular Cell Research, 2013, 1833(2): 410-416. DOI:10.1016/j.bbamcr.2012.02.019 |
| [26] |
WANG X W, CHEN X J. A cytosolic network suppressing mitochondria-mediated proteostatic stress and cell death[J]. Nature, 2015, 524(7566): 481-484. DOI:10.1038/nature14859 |
| [27] |
WROBEL L, TOPF U, BRAGOSZEWSKI P, et al. Mistargeted mitochondrial proteins activate a proteostatic response in the cytosol[J]. Nature, 2015, 524(7566): 485-488. DOI:10.1038/nature14951 |
| [28] |
FIORESE C J, SCHULZ A M, LIN Y F, et al. The transcription factor ATF5 mediates a mammalian mitochondrial UPR[J]. Current Biology, 2016, 26(15): 2037-2043. DOI:10.1016/j.cub.2016.06.002 |
| [29] |
QURESHI M A, HAYNES C M, PELLEGRINO M W. The mitochondrial unfolded protein response: signaling from the powerhouse[J]. The Journal of Biological Chemistry, 2017, 292(33): 13500-13506. DOI:10.1074/jbc.R117.791061 |
| [30] |
D'AMICO D, SORRENTINO V, AUWERX J. Cytosolic proteostasis networks of the mitochondrial stress response[J]. Trends in Biochemical Sciences, 2017, 42(9): 712-725. DOI:10.1016/j.tibs.2017.05.002 |
| [31] |
RUNKEL E D, LIU S, BAUMEISTER R, et al. Surveillance-activated defenses block the ROS-induced mitochondrial unfolded protein response[J]. PLoS Genetics, 2013, 9(3): e1003346. DOI:10.1371/journal.pgen.1003346 |
| [32] |
WU C G, KARAKUZU O, GARSIN D A. Tribbles pseudokinase NIPI-3 regulates intestinal immunity in Caenorhabditis elegans by controlling SKN-1/Nrf activity[J]. Cell Reports, 2021, 36(7): 109529. DOI:10.1016/j.celrep.2021.109529 |
| [33] |
MVNCH C. The different axes of the mammalian mitochondrial unfolded protein response[J]. BMC Biology, 2018, 16(1): 81. DOI:10.1186/s12915-018-0548-x |
| [34] |
TESKE B F, FUSAKIO M E, ZHOU D H, et al. CHOP induces activating transcription factor 5 (ATF5) to trigger apoptosis in response to perturbations in protein homeostasis[J]. Molecular Biology of the Cell, 2013, 24(15): 2477-2490. DOI:10.1091/mbc.e13-01-0067 |
| [35] |
FUSAKIO M E, WILLY J A, WANG Y P, et al. Transcription factor ATF4 directs basal and stress-induced gene expression in the unfolded protein response and cholesterol metabolism in the liver[J]. Molecular Biology of the Cell, 2016, 27(9): 1536-1551. DOI:10.1091/mbc.E16-01-0039 |
| [36] |
MVNCH C, HARPER J W. Mitochondrial unfolded protein response controls matrix pre-RNA processing and translation[J]. Nature, 2016, 534(7609): 710-713. DOI:10.1038/nature18302 |
| [37] |
TAO R D, COLEMAN M C, PENNINGTON J D, et al. Sirt3-mediated deacetylation of evolutionarily conserved lysine 122 regulates MnSOD activity in response to stress[J]. Molecular Cell, 2010, 40(6): 893-904. DOI:10.1016/j.molcel.2010.12.013 |
| [38] |
SALMINEN A, KAARNIRANTA K, KAUPPINEN A. Crosstalk between oxidative stress and SIRT1:impact on the aging process[J]. International Journal of Molecular Sciences, 2013, 14(2): 3834-3859. DOI:10.3390/ijms14023834 |
| [39] |
PAPA L, GERMAIN D. Estrogen receptor mediates a distinct mitochondrial unfolded protein response[J]. Journal of Cell Science, 2011, 124(9): 1396-1402. DOI:10.1242/jcs.078220 |
| [40] |
HEYTLER P G. Uncoupling of oxidative phosphorylation by carbonyl cyanide phenylhydrazones.Ⅰ.Some characteristics of m-Cl-CCP action on mitochondria and chloroplasts[J]. Biochemistry, 1963, 2: 357-361. DOI:10.1021/bi00902a031 |
| [41] |
GEORGAKOPOULOS N D, WELLS G, CAMPANELLA M. The pharmacological regulation of cellular mitophagy[J]. Nature Chemical Biology, 2017, 13(2): 136-146. DOI:10.1038/nchembio.2287 |
| [42] |
YOO S M, JUNG Y K. A molecular approach to mitophagy and mitochondrial dynamics[J]. Molecules and Cells, 2018, 41(1): 18-26. |
| [43] |
LIU L, LI Y J, WANG J N, et al. Mitophagy receptor FUNDC1 is regulated by PGC-1α/NRF1 to fine tune mitochondrial homeostasis[J]. EMBO Reports, 2021, 22(3): e50629. |
| [44] |
ALTSHULER-KEYLIN S, SHINODA K, HASEGAWA Y, et al. Beige adipocyte maintenance is regulated by autophagy-induced mitochondrial clearance[J]. Cell Metabolism, 2016, 24(3): 402-419. DOI:10.1016/j.cmet.2016.08.002 |
| [45] |
WANG H M, ZHANG T, GE X H, et al. Parkin overexpression attenuates Aβ-induced mitochondrial dysfunction in HEK293 cells by restoring impaired mitophagy[J]. Life Science, 2020, 244: 117322. DOI:10.1016/j.lfs.2020.117322 |
| [46] |
CAO S T, WANG C C, YAN J T, et al. Curcumin ameliorates oxidative stress-induced intestinal barrier injury and mitochondrial damage by promoting Parkin dependent mitophagy through AMPK-TFEB signal pathway[J]. Free Radical Biology & Medicine, 2020, 147: 8-22. |
| [47] |
CAO S T, XIAO H, LI X, et al. AMPK-PINK1/parkin mediated mitophagy is necessary for alleviating oxidative stress-induced intestinal epithelial barrier damage and mitochondrial energy metabolism dysfunction in IPEC-J2[J]. Antioxidants (Basel, Switzerland), 2021, 10(12): 2010. |
| [48] |
XIAO H, WU M M, Shao F Y, et al. N-acetyl-L-cysteine protects the enterocyte against oxidative damage by modulation of mitochondrial function[J]. Mediators of inflammation, 2016, 2016: 8364279. |
| [49] |
XIAO H, ZHA C F, SHAO F Y, et al. Amino acids regulate energy utilization through mammalian target of rapamycin complex 1 and adenosine monophosphate activated protein kinase pathway in porcine enterocytes[J]. Animal Nutrition, 2020, 6(1): 98-106. DOI:10.1016/j.aninu.2019.12.001 |
| [50] |
RODGER C E, MCWILLIAMS T G, GANLEY I G. Mammalian mitophagy-from in vitro molecules to in vivo models[J]. The FEBS Journal, 2018, 285(7): 1185-1202. DOI:10.1111/febs.14336 |
| [51] |
SUN N, YUN J, LIU J, et al. Measuring in vivo mitophagy[J]. Molecular Cell, 2015, 60(4): 685-696. DOI:10.1016/j.molcel.2015.10.009 |




