反刍动物甲烷(CH4)排放与全球气候变暖和饲料能损失密切相关。甲烷能损失占饲料总能的2%~12%[1]。研究表明,反刍动物甲烷排放量占全球甲烷排放量的15%[2]。除此之外,畜牧业产生的甲烷是人为排放甲烷的重要来源之一,占全球人类活动甲烷排放量的28%[3]。因此,减少反刍动物的甲烷排放量对提高饲粮能量利用率和保护环境有重要作用。
反刍动物甲烷主要通过瘤胃产甲烷菌经二氧化碳(CO2)/氢气(H2)还原途径生成[4]。产甲烷菌属于瘤胃古菌,可利用多种降解底物生成甲烷。瘤胃产甲烷菌受多种因素的影响,其中饲粮组成对产甲烷菌影响较大[5-6]。研究表明,高纤维饲粮可促进产甲烷菌的增殖,并提高反刍动物甲烷日排放量[7]。与高纤维饲粮比,高淀粉饲粮可降低产甲烷菌多样性和甲烷菌代谢活性[8]。此外,研究表明饲粮非纤维性碳水化合物(NFC)/中性洗涤纤维(NDF)比值对甲烷排放量有显著影响[9],随着饲粮NDF/NFC比值的降低,甲烷日排放量逐渐降低。这可能是低NDF/NFC比值的饲粮改变了瘤胃发酵模式,提高了丙酸利用菌与产甲烷菌竞争氢的能力,减少了甲烷菌合成甲烷的底物含量,并降低了产甲烷菌的活性,从而抑制了甲烷的生成[10]。目前对反刍动物甲烷减排的研究主要集中在调整饲粮组成;但是,通过调控饲粮NDF/淀粉比值来减少荷斯坦公牛甲烷排放量的相关研究较少。因此,本试验通过研究饲粮NDF/淀粉比值对荷斯坦公牛瘤胃古菌和甲烷排放量的影响,确定饲粮NDF/淀粉比值与甲烷排放量的相关关系,旨在为反刍动物甲烷减排提供理论依据和生产指导。
1 材料与方法 1.1 试验时间与地点饲养试验于2018年2月初至2018年5月中旬在保定市宏达奶牛场完成。
1.2 试验设计试验采用单因素完全随机试验设计,选择36头健康、膘情正常、体重[(282±37) kg]相近的荷斯坦公牛作为试验牛,随机分为3组,每组12头,散栏饲养。Ⅰ、Ⅱ、Ⅲ组试验牛分别饲喂NDF/淀粉比值为1.13、1.38、1.63的全混合日粮(TMR)。各组TMR能量水平相同,粗蛋白质水平接近。预试期7 d,正试期90 d。试验饲粮组成及营养水平见表 1。
|
|
表 1 试验饲粮组成及营养水平(干物质基础) Table 1 Composition and nutrient levels of experimental diets (DM basis) |
试验牛每天饲喂2次TMR(07:00和17:00),自由采食与饮水。每天打扫料槽,每周清洗水槽2~3次,保持水槽和料槽干净、卫生。
1.4 瘤胃液的采集与处理于正试期第88天晨饲前用口腔导管通过真空泵抽取瘤胃液,每组采集8头试验牛的瘤胃液,将采集的瘤胃液用4层纱布进行过滤后,装于已灭菌的离心管,立即投入液氮中,送回实验室后于-80 ℃冰箱保存。
1.5 绝对定量PCR 1.5.1 总DNA的提取瘤胃液总DNA的提取主要参考郭嫣秋等[15]、Qiao等[16]、李长青等[17]的方法,具体理论依据参考李长青等[17]、李萌等[18]。将样品涡旋1 min,取200 μL瘤胃液样品,按Solarbio通用基因组DNA试剂盒(北京索莱宝科技有限公司)说明书提取瘤胃液总DNA。
1.5.2 总DNA的质量检测使用Thermo NANo DROP 2000分光光度计检测瘤胃液总DNA的吸光度(OD)260 nm/280 nm在1.77~1.95,DNA浓度在25.0~97.7 ng/μL,并于-20 ℃保存。
1.5.3 引物序列试验引物选用β-肌动蛋白(β-actin)[19]为内参基因,目的基因为反刍兽甲烷短杆菌(Methanobrevibacter ruminantium)[15],引物合成由生工生物工程(上海)股份有限公司完成,基因引物序列见表 2。
|
|
表 2 基因引物序列 Table 2 Primer sequences of genes |
打开Eppendorp PCR仪预热20 min,反应体系和条件见表 3。
|
|
表 3 反应体系和条件 Table 3 Reaction system and conditions |
配制1%琼脂糖凝胶[称取0.2 g琼脂糖加20 mL 0.5×硼酸(TBE)]放入带盖的瓶子中,置于微波炉中(调到中低档,1~2 min),待瓶身微热加2 μL无毒核酸染料Super GelRed(产品货号:S2001,Everbright Inc,美国),倒胶,20~30 min后拔梳子,倒0.5×TBE,没过胶面,加样2 μL,电泳条件设为:120 V,100 mA。琼脂凝胶电泳图谱结果见图 1。
|
M:标记物marker;M.r:为反刍兽甲烷短杆菌Methanobrevibacter ruminantium;β:β-肌动蛋白β-actin。图 2同the same as Fig. 2。 图 1 琼脂凝胶电泳图谱 Fig. 1 Agar gel electrophoretogram |
称取0.25 g琼脂糖加25 mL 0.5×TBE(1%琼脂糖凝胶)放入带盖的瓶子中,置于微波炉中(调中低档,1~2 min),待瓶身微热加2.5 μL无毒核酸染料Super GelRed,倒胶,20~30 min后拔梳子,倒0.5×TBE,没过胶面,加样20 μL,电泳条件设为:120 V,100 mA,切胶。使用SanPrep柱式DNA胶回收试剂盒[生工生物工程(上海)股份有限公司]回收做标准品,测其浓度在10~30 ng/μL,于-20 ℃保存。胶回收产物电泳图谱结果见图 2。
|
图 2 胶回收产物电泳图谱 Fig. 2 Gel recovery product electrophoretogram |
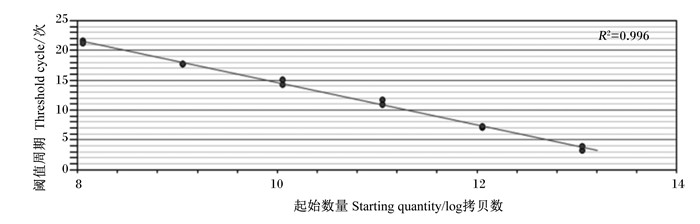
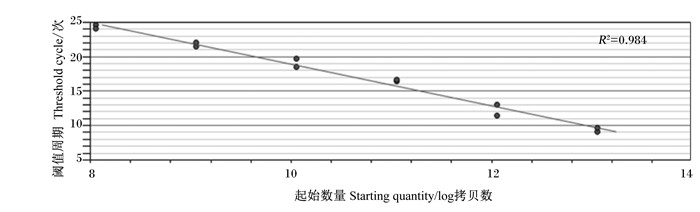
10倍稀释标准品(5 μL标准品加45 μL Easy dilution)后做8个梯度(目的基因:1.13×1013~1.13×107;内参基因:5.74×1014~1.13×108),每次加Easy dilution需涡旋3 min,离心30 s(保证混匀),全部溶解后,做好标记,于-20 ℃保存。使用Bio-Rad荧光定量PCR仪检测样品,反应体系及条件见表 4,选用6个点做标准曲线。熔解曲线有明显高峰,出峰温度在81 ℃,说明扩增产物特异性高。目的基因标准曲线:y=-3.543x+50.063(y是Ct值,x是log拷贝数),扩增效率为91.5%,r2=99.6%;内参基因标准曲线:y=-3.029x+49.217(y是Ct值,x是log拷贝数),扩增效率为113.0%,r2=98.4%。扩增曲线见图 3,熔解曲线见图 4,目的基因标准曲线见图 5,内参基因标准曲线见图 6。
|
|
表 4 反应体系及条件 Table 4 Reaction system and conditions |
|
图 3 扩增曲线 Fig. 3 Amplification curve |
|
图 4 熔解曲线 Fig. 4 Melting curve |
|
图 5 目的基因标准曲线 Fig. 5 Standard curve line of target gene |
|
图 6 内参基因标准曲线 Fig. 6 Standard curve line of reference gene |
同一批次96孔板目的基因、内参基因各设标准曲线7个点、24个待测混池样品、空白样品各2个重复,待测样品目的基因、内参基因各3个重复,反应体系及条件同表 4。
1.6 指标测定 1.6.1 瘤胃古菌多样性测定对古菌16S V9区进行Illumina HiSeq测序,由北京诺禾致源科技有限公司代测。数据库测定方法:参照Qiime V1.9.1软件的Tags质量控制流程对下机样本数据进行拼接、过滤后为有效数据(clean data)。利用Uparse v7.0.1001软件[20]以97%的一致性对有效数据进行操作分类单元(OTUs)聚类,对具有OTUs代表序列使用Mothur方法与SILVA 132数据库[21]进行物种注释。
1.6.2 样品六氟化硫(SF6)和甲烷浓度的测定以及甲烷排放量的计算甲烷浓度测定:每次测定样品前应先用2.74 mg/m3的甲烷标准气体进行校正。色谱条件:载气为99.999%高纯氮气,燃气为99.999%高纯H2,空气为助燃气,柱温为70 ℃,前进样口温度为50 ℃,后进样口温度为375 ℃,火焰离子化检测器(FID)温度为200 ℃,进样量为1 mL,空气流速为400 mL/min,H2流速为30 mL/min,氮气流速为25 mL/min,分流比为20 ∶ 1。SF6送北京中科院大气物理研究所中国生态系统研究网络大气分中心代为检测,SF6标准气体浓度为1.99×10-4 mg/m3。SF6测定试验条件:载气为氩气(Ar)-甲烷(10%),载气流速为30 mL/min,电子捕获检测器(ECD)温度为350 ℃,分析柱温度为55 ℃,进样量为5 mL。反刍动物甲烷排放量计算公式如下:
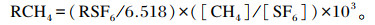
|
式中:RCH4是甲烷排放速率(L/d);RSF6是SF6渗透速率(mg/d);6.518是SF6的密度(kg/m3);[CH4]是样品气体中甲烷浓度(×10-6摩尔分数);[SF6]是样品气体中SF6浓度(×10-12摩尔分数)。
1.7 未知样品拷贝数未知样品拷贝数计算公式[15]如下:
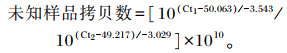
|
式中:Ct1为目的基因Ct值;Ct2为内参基因Ct值;指数(10)为胶回收产物中目的基因标准曲线拷贝数与内参基因标准曲线拷贝数的比值。
1.8 统计数据应用SPSS 19.0统计软件的单因素方差分析对试验数据进行统计分析,试验结果用“平均值±标准差”表示。
2 结果与分析 2.1 饲粮NDF/淀粉比值对荷斯坦公牛甲烷排放量的影响由表 5可知,Ⅰ组甲烷排放量较Ⅱ、Ⅲ组极显著降低了33.13%、67.89%(P<0.01)。
|
|
表 5 荷斯坦公牛甲烷排放量 Table 5 Methane production of Holstein bulls |
由图 7可知,大部分样本量的稀释曲线在序列数为60 000~80 000时趋于平坦,说明测序数据量渐进合理,更多测序数据对于产生新的OTUs边际效应较小。这说明该样本量可反映瘤胃古菌的真实情况。
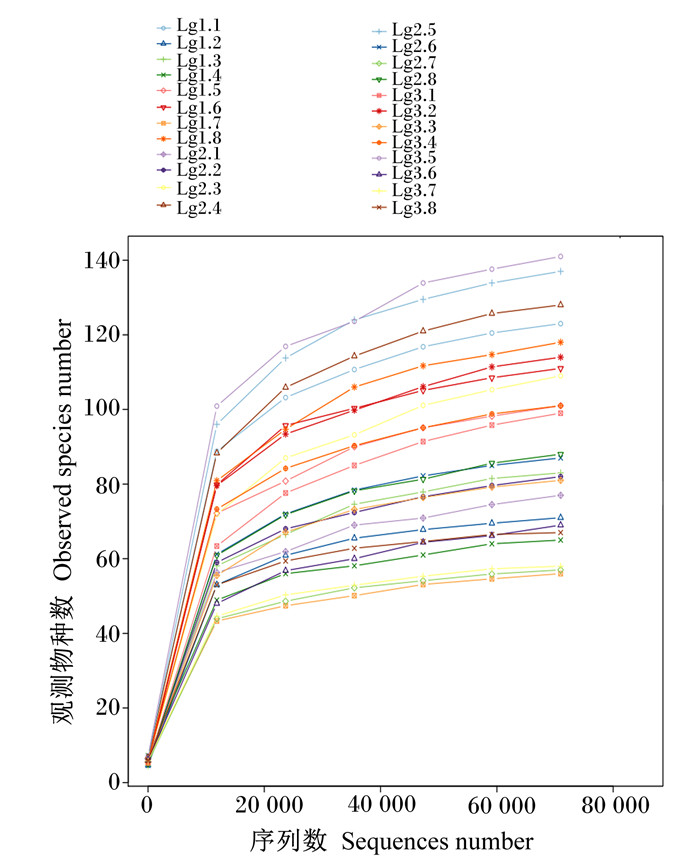
|
Lg1.1~Lg1.8、Lg2.1~Lg2.8、Lg3.1~Lg3.8分别为Ⅰ、Ⅱ、Ⅲ组瘤胃液样品稀释曲线。 Lg1.1 to Lg1.8, Lg2.1 to Lg2.8 and Lg3.1 to Lg3.8 were the dilution curves of rumen fluid samples in groups Ⅰ, Ⅱ and Ⅲ, respectively. 图 7 稀释曲线 Fig. 7 Dilution curve |
由表 6可知,各组的覆盖率(goods-coverage)均为1.000,说明测序深度高,足以反映古菌的菌群多样性和丰富度。Ⅲ组Chao1指数显著高于Ⅰ组(P<0.05),系统发育树(PD_whole_tree)极显著高于Ⅰ组(P<0.01)。
|
|
表 6 Alpha多样性指数 Table 6 Alpha diversity index |
由表 7可知,各组甲烷短杆菌属(Methanobrevibacter)在瘤胃古菌中占绝对优势,其相对丰度≥97%。Ⅲ组甲烷短杆菌属相对丰度高于Ⅰ、Ⅱ组(P<0.05)。
|
|
表 7 荷斯坦公牛属水平瘤胃古菌组成 Table 7 Composition of rumen archaea of Holstein bulls at genus level |
Beta多样性是对不同样品的微生物菌落进行比较分析,非加权组平均法(unweighted pair-group method with arithmetic mean,UPGMA)属于Beta多样性的一种分析方法,可以用于研究不同样品的相似性,构建样品的聚类树,对样品进行聚类分析。由图 8可知,Ⅰ、Ⅱ组样品瘤胃古菌菌群相似度较高,故聚于一类。
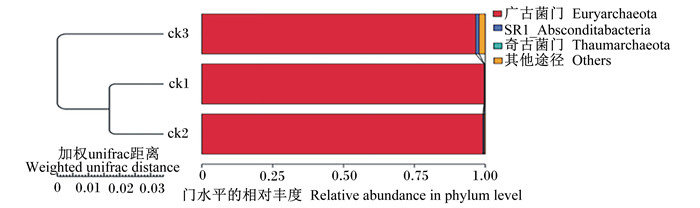
|
ck1、ck2、ck3分别代表Ⅰ、Ⅱ、Ⅲ组。下图同。 ck1, ck2 and ck3 represented for groups Ⅰ, Ⅱ and Ⅲ, respectively. The same as below. 图 8 UPGMA聚类树 Fig. 8 UPGMA cluster tree |
由图 9可知,Ⅰ组荷斯坦公牛瘤胃甲烷菌产甲烷途径有4种,包括CO2/H2还原途径、甲烷生成途径、暗氢氧化途径、氢营养型途径;Ⅱ组荷斯坦公牛瘤胃甲烷菌产甲烷途径有5种,包括CO2/H2还原途径、甲烷生成途径、暗氢氧化途径、氢营养型途径、乙酸发酵途径;Ⅲ组荷斯坦公牛瘤胃甲烷菌产甲烷途径有7种,包括乙酸发酵途径、甲基化合物/H2还原途径、甲酸生成甲烷途径、固氮途径、化能异养、甲基原子团失衡生成甲烷途径、其他途径。这说明各组甲烷产生途径存在差异。

|
methanogenesis by_CO2 reduction with H2:CO2/H2还原途径;methanogenesis:甲烷生成途径;dark_hydrogen_oxidation:暗氢氧化途径;hydrogenotrophic_methanogenesis:氢营养型途径;acetoclastic_methanogenesis:乙酸发酵途径;methanogenesis_by_reduction_of_methyl_compounds_with H2:甲基化合物/H2还原途径;methanogenesis_using_formate:甲酸生成甲烷途径;nitrogen_fixation:固氮途径;chemoheterotrophy:化能异养methanogenesis_by_disproporionation of metyl groups:甲基原子团失衡生成甲烷途径;others:其他途径。 图 9 PICRUSt功能基因预测聚类热图 Fig. 9 PICRUSt functional genes predicted clustering heat map |
基于功能基因的主成分分析(PCA)表示,如果样品的功能组成越相似,则它们在PCA图中的距离越接近。由图 10可知,Ⅰ、Ⅱ组功能组成较为接近。
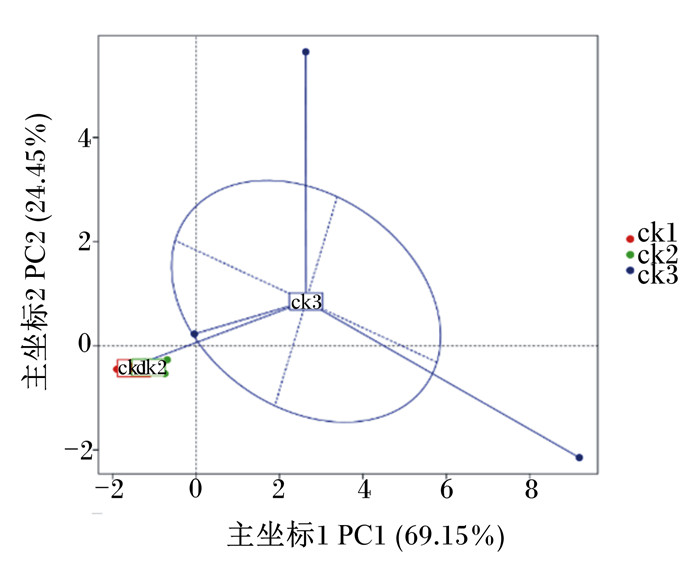
|
图 10 基于功能基因的PCA Fig. 10 PCA based on functional gene |
由表 8可知,各组之间反刍兽甲烷短杆菌数量存在极显著差异(P<0.01)。
|
|
表 8 饲粮NDF/淀粉比值对荷斯坦公牛反刍兽甲烷短杆菌数量的影响 Table 8 Effects of dietary NDF/starch ratio on Methanobrevibacter ruminantium number of Holstein bulls |
反刍动物的甲烷排放量约占畜牧业总甲烷排放量的81%,其中90%是由瘤胃微生物甲烷菌产生[22],甲烷作为饲料发酵的终产物也意味着机体能量损失,如果将这些能量在饲料转化过程中转化为瘤胃发酵产物的能量在能量守恒理论上可用于动物的生产。因此,降低甲烷排放量有助于提高动物的生产效率。
反刍动物甲烷生成量受采食量、饲料种类、饲养水平、饲喂次数等因素的影响[23]。合理调控饲粮精粗比是减少甲烷排放量的关键。研究表明,当饲粮精料比例从0提高到70%,瘤胃甲烷排放量可降低40%[24]。与高纤维饲粮相比,高淀粉饲粮能降低甲烷排放量[25]。丁静美等[10]研究表明,NDF/NFC比值为2.32饲粮组的肉羊甲烷排放量显著高于NDF/NFC比值为1.58和1.04饲粮组。淀粉是NFC主要成分,是机体重要的能量供应来源,淀粉较NFC测定准确,用饲粮NDF/淀粉比值来反映饲粮易发酵程度和瘤胃发酵特性更为直观。本试验研究结果表明,饲粮NDF/淀粉比值为1.63组的荷斯坦公牛甲烷排放量显著高于饲粮NDF/淀粉比值为1.13和1.38组,这与Popova等[8]研究结果一致,其原因是随着饲粮淀粉水平的升高,产甲烷菌的活性会降低,甲烷排放量随之减少。
3.2 饲粮NDF/淀粉比值对荷斯坦公牛瘤胃古菌多样性的影响Alpha多样性是用于分析样品内物种的丰富度和多样性[26]。研究表明,纤维摄入能够提高产甲烷菌群多样性,表现为与高淀粉饲粮组比,高纤维饲粮组Shannon指数更高[27-28],原因是饲粮组成会通过影响产甲烷菌产甲烷途径或基于基因水平的差异对产甲烷过程起重要作用[29]。研究表明,补饲精料组瘤胃甲烷菌群组成发生显著变化。本试验中,饲粮NDF/淀粉比值为1.63组Chao1指数显著高于饲粮NDF/淀粉比值为1.13组(P<0.05),PD_whole_tree极显著高于饲粮NDF/淀粉比值为1.13组,这与杨硕[7]的研究结果一致。这说明随饲粮NDF/淀粉比值的增加瘤胃古菌物种多样性有所提高,原因可能是高饲粮NDF/淀粉比值促进了纤维分解菌、原虫、纤毛虫、真菌的增殖,促进了以不同底物为生的甲烷菌的生长繁殖和甲烷合成途径的多样性,菌群种类更加丰富;也可能是宿主动物个体差异对瘤胃产甲烷菌产生了较大影响[30]。
饲粮组成是影响瘤胃产甲烷菌种群组成的因素之一[31]。研究表明,高淀粉饲粮能够显著降低甲烷排放量,且瘤胃甲烷排放量与产甲烷菌密切相关[31]。饲粮精料比例增加能够提高优势菌群相对丰度[7]。本试验中,各组瘤胃甲烷短杆菌属在古菌中占绝对优势。饲粮NDF/淀粉比值为1.63组甲烷短杆菌属相对丰度高于其他2组,其他2组之间甲烷短杆菌属相对丰度无显著差异,而各组之间甲烷排放量均存在极显著差异,说明饲粮NDF/淀粉比值不仅对甲烷短杆菌属相对丰度产生了影响,也在种水平数量上产生了较大影响。
3.3 饲粮NDF/淀粉比值对荷斯坦公牛瘤胃甲烷菌的功能预测PICRUSt能够根据标记物基因(如16S rRNA)对元基因组进行功能预测[32]。金磊等[33]基于高通量测序技术对山羊盲肠进行预测分析发现,盲肠菌群对山羊营养物质消化代谢影响较大。本试验中,各组之间甲烷产生途径存在明显差异,这与UPGMA聚类树、基于功能基因的PCA结果相似,高饲粮NDF/淀粉比值组的荷斯坦公牛瘤胃甲烷菌的甲烷生成途径多于中、低饲粮NDF/淀粉比值组;而从实测甲烷排放量可知,高饲粮NDF/淀粉比值组的甲烷排放量是中、低粮NDF/淀粉比值甲烷排放量之和的1.25倍。这说明瘤胃古菌多样性提高是甲烷排放量大幅提高的主要诱导因素。
3.4 饲粮NDF/淀粉比值对荷斯坦公牛反刍兽甲烷短杆菌数量的影响高通量测序技术主要基于OTUs进行结果分析,但序列数并不能完全反映所代表细菌相对丰度[34]。研究表明,高通量测序技术可能会高估或低估一些微生物类群的丰度或多样性(如死的生物也被检测到)[35]。一项研究指出,实时定量PCR能够准确检测绵羊瘤胃未知细菌的种群大小及分布情况[36]。因此,实时定量PCR技术可能是检测古菌种数量的重要途径。
相对定量PCR是将待测靶基因片段和内参基因片段同时扩增,就两者Ct值之差进行测量[18]。陈小连[37]对3种瘤胃优势纤维菌绝对定量与相对定量的比较发现,2种方法都能准确测定瘤胃微生物数量。反刍兽甲烷短杆菌属于甲烷短杆菌属,能够从奶牛、公牛、鹿[38-39]瘤胃中分离得到。甲烷短杆菌属属于甲烷杆菌科,是氢营养型甲烷菌[40],对甲烷产生贡献率较大[41],且在荷斯坦公牛瘤胃古菌中为优势甲烷菌。因此,本试验采用绝对定量方法对反刍兽甲烷短杆菌数量进行检测。从绝对定量分析结果发现,各组之间反刍兽甲烷短杆菌数量有极显著差异。这说明饲粮NDF/淀粉比值对种水平甲烷短杆菌具有显著影响。杨硕[7]研究表明,采食高纤维含量牧草的绒山羊瘤胃以乙酸发酵为主,从而生成大量的H2,导致以此为生长底物的甲烷菌大量繁殖,且瘤胃产甲烷菌数量随纤维降解菌的升高而增多。本试验中,低饲粮NDF/淀粉比值组荷斯坦公牛瘤胃反刍兽甲烷短杆菌数量显著低于其他2组,说明低NDF/淀粉比值的饲粮改变了荷斯坦公牛瘤胃发酵模式,瘤胃丙酸利用菌与产甲烷菌竞争H2产生丙酸,减少了产甲烷菌可利用底物,导致反刍动物甲烷菌株受到抑制。
4 结论降低饲粮NDF/淀粉比值能够减少甲烷排放量,降低瘤胃古菌丰富度指数,降低甲烷短杆菌属相对丰度和反刍兽甲烷短杆菌数量,并减少了荷斯坦公牛瘤胃甲烷菌生成甲烷途径。
| [1] |
JOHNSON K A, JOHNSON D E. Methane emissions from cattle[J]. Journal of Animal Science, 1995, 73(8): 2483-2492. DOI:10.2527/1995.7382483x |
| [2] |
MCGINN S M, FLESCH T K, HARPER L A, et al. An approach for measuring methane emissions from whole farms[J]. Journal of Environmental Quality, 2006, 35(1): 14-20. DOI:10.2134/jeq2005.0250 |
| [3] |
YUSUF R O, NOOR Z Z, ABBA A H, et al. Methane emission by sectors: a comprehensive review of emission sources and mitigation methods[J]. Renewable and Sustainable Energy Reviews, 2012, 16(7): 5059-5070. DOI:10.1016/j.rser.2012.04.008 |
| [4] |
王胤晨, 袁扬, 张锦华, 等. 反刍动物瘤胃甲烷产生的营养调控[J]. 中国牛业科学, 2015, 41(3): 100-104. WANG Y C, YUAN Y, ZHANG J H, et al. Nutritional regulation of rumen methane production in ruminant animals[J]. China Cattle Science, 2015, 41(3): 100-104 (in Chinese). DOI:10.3969/j.issn.1001-9111.2015.03.030 |
| [5] |
LIN C Z, RASKIN L, STAHL D A. Microbial community structure in gastrointestinal tracts of domestic animals: comparative analyses using rRNA-targeted oligonucleotide probes[J]. FEMS Microbiology Ecology, 1997, 22(4): 281-294. DOI:10.1111/j.1574-6941.1997.tb00380.x |
| [6] |
WRIGHT A D G, WILLIAMS A J, WINDER B, et al. Molecular diversity of rumen methanogens from sheep in Western Australia[J]. Applied and Environmental Microbiology, 2004, 70(3): 1263-1270. |
| [7] |
杨硕. 养殖方式和绒山羊类型对瘤胃产甲烷菌及相关微生物区系的影响[D]. 硕士学位论文. 呼和浩特: 内蒙古农业大学, 2018. YANG S. Effects of feeding types and breeds on rumen methanogens and related microflora in Inner Mongolia cashmere goats[D]. Master's Thesis. Hohhot: Inner Mongolia Agricultural University, 2018. (in Chinese) |
| [8] |
POPOVA M, MARTIN C, EUGōNE M, et al. Effect of fibre- and starch-rich finishing diets on methanogenic Archaea diversity and activity in the rumen of feedlot bulls[J]. Animal Feed Science and Technology, 2011, 166/167: 113-121. DOI:10.1016/j.anifeedsci.2011.04.060 |
| [9] |
周艳, 刁其玉, 董利锋, 等. 饲粮非纤维性碳水化合物(NFC)与中性洗涤纤维(NDF)比例对育成期杜寒杂交母羊生长性能、营养物质消化率和甲烷产量的影响[J]. 畜牧兽医学报, 2018, 49(7): 1405-1415. ZHOU Y, DIAO Q Y, DONG L F, et al. The effect of different ratios of dietary non-fibrous carbohydrate (NFC) to neutral detergent fiber (NDF) on production performance, nutrient digestibility and methane emissions of Dorper×Thin-tailed Han crossbred growing ewes[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(7): 1405-1415 (in Chinese). |
| [10] |
丁静美, 屠焰, 成述儒, 等. 不同NDF/NFC饲粮下肉羊瘤胃甲烷排放量和微生物区系变化研究[C]//2018年全国养羊生产与学术研讨会论文集. 蚌埠: 中国畜牧兽医学会养羊学分会, 2018: 29-30. DING J M, TU Y, CHENG S R. Study on rumen methane emission and microflora changes of meat sheep under different NDF/NFC diets[C]//Proceedings of 2018 National Sheep Breeding Production and Academic Symposium. Bengbu: Sheep Breeding Branch of Chinese Association of Animal Science and Veterinary Medicine, 2018: 29-30. (in Chinese) |
| [11] |
国家技术监督局. 饲料中粗蛋白测定方法: GB/T 6432-1994[S]. 北京: 中国标准出版社, 1994. The State Bureau of Quality and Technical Supervision. Method for the determination of crude protein in feedstuffs: GB/T 6432-1994[S]. Beijing: Standards Press of China, 1994. (in Chinese) |
| [12] |
中华人民共和国国家质量监督检验检疫总局. 饲料中钙的测定: GB/T 6436-2002[S]. 北京: 中国标准出版社, 2002. General Administration of Quality Supervision, Inspection and Quarantine of the People's Republic of China. Determination of calcium in feed: GB/T 6436-2002[S]. Beijing: Standards Press of China, 2002. (in Chinese) |
| [13] |
中华人民共和国国家质量监督检验检疫总局. 饲料中总磷的测定分光光度法: GB/T 6437-2002[S]. 北京: 中国标准出版社, 2002. General Administration of Quality Supervision, Inspection and Quarantine of the People's Republic of China. Determination of phosphorus in feed-spectphotometry: GB/T 6437-2002[S]. Beijing: Standards Press of China, 2002. (in Chinese) |
| [14] |
VAN SOEST P J, ROBERTSON J B, LEWIS B A. Methods for dietary fiber, neutral detergent fiber, and nonstarch polysaccharides in relation to animal nutrition[J]. Journal of Dairy Science, 1991, 74(10): 3583-3597. DOI:10.3168/jds.S0022-0302(91)78551-2 |
| [15] |
郭嫣秋. 瘤胃产甲烷菌定量检测与微生物菌群调控研究[D]. 博士学位论文. 杭州: 浙江大学, 2008. GUO Y Q. Quantification of methanogens and manipulation of microbial community in the rumen[D]. Ph. D. Thesis. Hangzhou: Zhejiang University, 2008. (in Chinese) |
| [16] |
QIAO J Y, TAN Z L, GUAN L L, et al. Effects of hydrogen in headspace and bicarbonate in media on rumen fermentation, methane production and methanogenic population using in vitro gas production techniques[J]. Animal Feed Science and Technology, 2015, 206: 19-28. DOI:10.1016/j.anifeedsci.2015.05.004 |
| [17] |
李长青, 金海, 栗原光规. 瘤胃总甲烷菌实时荧光定量PCR方法的构建[J]. 畜牧与饲料科学, 2010, 31(6): 467-469. LI C Q, JIN H, LI Y G G. Development of a real-time absolute quantitative PCR for quantification of total methanogens in rumen[J]. Animal Husbandry and Feed Science, 2010, 31(6): 467-469 (in Chinese). DOI:10.3969/j.issn.1672-5190.2010.06.156 |
| [18] |
李萌, 李雪, 郑志强. 实时荧光定量PCR技术的应用及研究进展[J]. 饲料博览, 2018(7): 94. LI M, LI X, ZHENG Z Q. Application and research progress of real-time fluorescent quantitative PCR[J]. Feed Review, 2018(7): 94 (in Chinese). DOI:10.3969/j.issn.1001-0084.2018.07.052 |
| [19] |
岳康宁. 日粮能量水平对淘汰荷斯坦母牛育肥性能的影响及机理研究[D]. 硕士学位论文. 保定: 河北农业大学, 2018. YUE K N. Influence of dietary energy levels on fattening performance and mechanism of culling Holstein cows[D]. Master's Thesis. Baoding: Hebei Agricultural University, 2018. (in Chinese) |
| [20] |
HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [21] |
EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [22] |
DU Y Y, GE Y, REN Y, et al. A global strategy to mitigate the environmental impact of China's ruminant consumption boom[J]. Nature Communications, 2018, 9(1): 4133. DOI:10.1038/s41467-018-06381-0 |
| [23] |
胡红莲, 卢德勋, 高民. 反刍动物甲烷的排放及其减排调控技术[J]. 畜牧与饲料科学, 2011, 32(9/10): 26-29. HU H L, LU D X, GAO M. The emission and its control technology of ruminant methane[J]. Animal Husbandry and Feed Science, 2011, 32(9/10): 26-29 (in Chinese). |
| [24] |
BENCHAAR C, POMAR C, CHIQUETTE J. Evaluation of dietary strategies to reduce methane production in ruminants: a modelling approach[J]. Canadian Journal of Animal Science, 2001, 81(4): 563-574. DOI:10.4141/A00-119 |
| [25] |
BOUGOUIN A, FERLAY A, DOREAU M, et al. Effects of carbohydrate type or bicarbonate addition to grass silage-based diets on enteric methane emissions and milk fatty acid composition in dairy cows[J]. Journal of Dairy Science, 2018, 101(7): 6085-6097. DOI:10.3168/jds.2017-14041 |
| [26] |
LI B, ZHANG X X, GUO F, et al. Characterization of tetracycline resistant bacterial community in saline activated sludge using batch stress incubation with high-throughput sequencing analysis[J]. Water Research, 2013, 47(13): 4207-4216. DOI:10.1016/j.watres.2013.04.021 |
| [27] |
MACHMVLLER A, SOLIVA C R, KREUZER M. Effect of coconut oil and defaunation treatment on methanogenesis in sheep[J]. Reproduction, Nutrition, Development, 2003, 43(1): 41-55. DOI:10.1051/rnd:2003005 |
| [28] |
HOOK S E, STEELE M A, NORTHWOOD K S, et al. Impact of high-concentrate feeding and low ruminal pH on methanogens and protozoa in the rumen of dairy cows[J]. Microbial Ecology, 2011, 62(1): 94-105. DOI:10.1007/s00248-011-9881-0 |
| [29] |
ZHOU M, HERNANDEZ-SANABRIA E, GUAN L L. Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions, as determined by PCR-denaturing gradient gel electrophoresis analysis[J]. Applied and Environmental Microbiology, 2010, 76(12): 3776-3786. DOI:10.1128/AEM.00010-10 |
| [30] |
ZHOU M, HVNERBERG M, BEAUCHEMIN K A, et al. Individuality of ruminal methanogen/protozoa populations in beef cattle fed diets containing dried distillers' grain with solubles[J]. Acta Agriculturae Scandinavica, Section A-Animal Science, 2012, 62(4): 273-288. DOI:10.1080/09064702.2013.788206 |
| [31] |
张学燕, 刘书杰, 崔占鸿, 等. 应用16S rRNA基因序列技术分析青海高原放牧牦牛瘤胃产甲烷菌的多样性[J]. 青海大学学报, 2018, 36(1): 9-16, 46. ZHANG X Y, LIU S J, CUI Z H, et al. Study on ruminal methanogens diversity of grazing yak using 16S rRNA library-based technique in Qinghai plateau[J]. Journal of Qinghai University, 2018, 36(1): 9-16, 46 (in Chinese). |
| [32] |
LANGILLE M G I, ZANEVELD J, CAPORASO J G, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [33] |
金磊, 王立志, 王之盛, 等. 基于高通量测序技术对山羊盲肠细菌多样性的分析[J]. 微生物学通报, 2019, 46(6): 1423-1433. JIN L, WANG L Z, WANG Z S, et al. Analysis of cecum bacterial diversity of goat based on Illumina MiSeq sequencing[J]. Microbiology, 2019, 46(6): 1423-1433 (in Chinese). |
| [34] |
KIM M, YU Z T. Quantitative comparisons of select cultured and uncultured microbial populations in the rumen of cattle fed different diets[J]. Journal of Animal Science and Biotechnology, 2012, 3(1): 28. DOI:10.1186/2049-1891-3-28 |
| [35] |
艾铄, 张丽杰, 肖芃颖, 等. 高通量测序技术在环境微生物领域的应用与进展[J]. 重庆理工大学学报(自然科学), 2018, 32(9): 111-121. AI S, ZHANG L J, XIAO P Y, et al. Application and progress of high-throughput sequencing technology in the field of environmental microorganisms[J]. Journal of Chongqing University of Technology (Natural Science), 2018, 32(9): 111-121 (in Chinese). |
| [36] |
STIVERSON J, MORRISON M, YU Z T. Populations of select cultured and uncultured bacteria in the rumen of sheep and the effect of diets and ruminal fractions[J]. International Journal of Microbiology, 2011, 2011: 750613. |
| [37] |
陈小连. 稻草与补充料组合影响瘤胃纤维消化和微生物生态的效应研究[D]. 博士学位论文. 杭州: 浙江大学, 2007. CHEN X L. Effects of rice straw and supplementary feed on rumen fiber digestion and microbial ecology[D]. Ph. D. Thesis. Hangzhou: Zhejiang University, 2007. (in Chinese) |
| [38] |
MILLER T L, WOLIN M J, ZHAO H X, et al. Characteristics of methanogens isolated from bovine rumen[J]. Applied and Environmental Microbiology, 1986, 51(1): 201-202. DOI:10.1128/aem.51.1.201-202.1986 |
| [39] |
SMITH P H, HUNGATE R E. Isolation and characterization of Methanobacterium ruminantium n. sp[J]. Journal of Bacteriology, 1958, 75(6): 713-718. DOI:10.1128/jb.75.6.713-718.1958 |
| [40] |
HUWS S A, CREEVEY C J, OYAMA L B, et al. Addressing global ruminant agricultural challenges through understanding the rumen microbiome: past, present, and future[J]. Frontiers in Microbiology, 2018, 9: 2161. DOI:10.3389/fmicb.2018.02161 |
| [41] |
朱伟云. 反刍动物瘤胃甲烷生成机理及其营养调控[C]//生态环境与畜牧业可持续发展学术研讨会暨中国畜牧兽医学会2012年学术年会和第七届全国畜牧兽医青年科技工作者学术研讨会会议-特邀报告. 北京: 中国农业工程学会, 中国畜牧兽医学会, 2012: 25-27. ZHU W Y. Rumen methane generation mechanism and nutrition regulation in ruminants[C]//Academic seminar on ecological environment and sustainable development of animal husbandry, 2012 annual meeting of Chinese Society of Animal Husbandry and Veterinary Science and technology and the 7th national conference of young scientific and technological workers in animal husbandry and veterinary science and technology-invited report. Beijing: Chinese Society of Agricultural Engineering, Chinese Association of Animal Science and Veterinary Medicine, 2012: 25-27. (in Chinese) |




